2. 南方医科大学 网络中心, 广东 广州 510515
2. Network Center, Southern Medical University, Guangzhou 510515, China
X染色体失活、基因簇印迹和基因组区域的组蛋白修饰和DNA甲基化都是表观基因组修饰,但调控这些修饰的分子机制多年未能揭示。最近研究揭示,大量表观基因组修饰是由长链非编码RNA(long non-coding RNA, lncRNA)调控的[1],它们长度大于200 bp,结构保守性高于序列保守性,包含多个功能域。许多lncRNA能与DNA和蛋白质结合,由此能把polycomb家族蛋白和DNA甲基转移酶(DNMT)携带到特定的基因组位点调控组蛋白修饰和DNA甲基化[2]。近来发现许多基因表达错误是由基因组修饰错误导致的,因此lncRNA成为生物医学研究一个重要而发展迅速的领域。大量研究揭示lncRNA参与的基因表达调控对许多生理与病理活动有重要影响,与癌症[3]、心血管疾病[4]、神经退行性疾病[5]等许多疾病的发生发展有关,从新的角度揭示了肿瘤、干细胞、衰老等过程在基因组层次的控制机制。
与蛋白质编码基因相比lncRNA有3个特性,即数量大、呈现突出的组织特异性表达[6]、具有明显的种系特异性[7]。随着大量lncRNA在越来越多的物种被发现,用生物信息学方法收集、整合、分析lncRNA数据也日益必要,成为lncRNA功能研究的重要前提。《核酸研究》的数据库专辑等已报道了若干lncRNA数据库,包括lncRNAdb [8]、LncRNADisease [9]、ChIPBase [10]、DeepBase [11]等,它们各有特点(表 1)。LncRNAdb收集经实验验证的lncRNA,包括序列、物种、功能、表达等信息,目前的LncRNAdb v2.0收集了294条lncRNA,数量如此少的原因是大量由RNA-seq鉴定的lncRNA尚未得到实验验证,因此LncRNAdb对分析新lncRNA基因和分析lncRNA功能作用有限。LncRNADisease收集了人类lncRNA与疾病的关系,从500余篇文献中收录了和221种疾病相关的322条lncRNA。根据lncRNA调控基因组修饰的机制,一个lncRNA导致何种疾病取决于其所调控的基因组位点和靶基因,因此预测其基因组位点和靶基因才能从机制上揭示lncRNA与疾病的关系。ChIPBase根据ChIP-Seq数据对lncRNA的转录调节功能进行注释,但所包含的lncRNA也较少。DeepBase则根据深度测序数据鉴别和注释非编码RNA。上述数据库都只收录已鉴定的lncRNA(主要在人类和小鼠),不含lncRNA的多物种同源序列,对大规模lncRNA比较研究和功能分析帮助有限。
| 表 1 LongMan与已有lncRNA数据库的比较 Table 1 LongMan and some other lncRNA databases(by Nov 30, 2015) |
国际合作的GENECODE项目首期报道了人类的13562个lncRNA [12],深入分析这些逾万个lncRNA极其必要,但显然需要计算方法。分析的第一步是确定同源序列,然后对同源序列进行保守性和功能域分析,这些分析是进一步功能研究的前提。同源lncRNA对于研究lncRNA的起源和进化以及lncRNA的种系特异性也必不可少。目前大规模的lncRNA同源数据尚无报道,本文报道的LongMan(Long noncoding RNAs orthologous to huMan)是首个lncRNA同源序列数据库。为了获得人类lncRNA在哺乳动物的同源lncRNA,根据GENCODE [12]v18报道的13 562个人类lncRNA,我们用Infernal软件在16个哺乳动物基因组的同源区域搜索同源lncRNA,在此基础上建立的lncRNA同源序列数据库目前包含133 646条lncRNA记录(http://lncrna.smu.edu.cn)。
1 数据和方法 1.1 人类lncRNA数据及基因组数据人类lncRNA数据来自于www.gencodegenes.org/releases/18.html,根据GENCODE v18发布的人类lncRNA注释文件(gtf文件)从人类基因组(ftp.ensembl.org,GRC37/hg19)获取13562个lncRNA的序列。
1.2 哺乳动物基因组数据16个哺乳动物的基因组数据下载自UCSC网站(hgdownload.soe.ucsc.edu/downloads.html),物种和基因组版本号为Chimpanzee(CSAC 2.1.4/panTro4),Macaque(BGI CR_1.0/rheMac3),Marmoset(WUGSC 3.2/calJac3),Tarsier(Broad/tarSyr1),Mouse lemur(Broad/micMur1),Tree shrew(Broad/tupBel1),Mouse(GRCm38/mm10),Rat(Baylor3.4/rn4),Guinea pig(Broad/cavPor3),Rabbit(Broad/oryCun2),Dog(Broad CanFam3.1/canFam3),Cow(Baylor Btau_4.6.1/bosTau7),Elephant(Broad/loxAfr3),Hedgehog(EriEur2.0/eriEur2),Opossum(Broad/monDom5),Platypus(WUGSC 5.0.1/ornAna1)。
1.3 人类-哺乳动物全基因组双序列比对数据16组人类-哺乳动物全基因组双序列比对数据下载自UCSC[13]网站(hgdownload.soe.ucsc.edu),分别为Human/Chimpanzee,Human/Macaque,Human/Marmoset,Human/Tarsier,Human/Mouse lemur,Human/Tree shrew,Human/Mouse,Human/Rat,Human/Guinea pig,Human/Rabbit,Human/Dog,Human/Cow,Human/Elephant,Human/Hedgehog,Human/Opossum,Human/Platypus,基因组版本号同1.1及1.2。
1.4 人类lncRNA外显子同源序列搜索首先,根据人类lncRNA的基因组地址以及人类-哺乳动物全基因组双序列比对,确定每个人类lncRNA基因在其它16个基因组的同源区域。为保证同源区域有可靠的长度,我们在每个同源区域两端拓展了四倍于同源区域的上下游序列作为该lncRNA的搜索区域。
如同其它非编码RNA序列,lncRNA序列可能存在补偿性突变,由此使得lncRNA序列差异度大但结构保守度高,不能用通常的序列搜索方法与软件(如BLAST)搜索lncRNA的同源序列。我们首先用RNAfold [14(]采用默认参数)对13562个人类lncRNA的每个外显子进行结构预测;随后用Infernal[15]中的cmbuild程序对这些外显子的二级结构构建CM模型;然后以CM模型作为query,使用Infernal的cmsearch程序在16个哺乳动物基因组(同源序列搜索区域)中搜索13562个人类lncRNA的每个外显子。由于Infernal难以对太长的RNA序列进行有效搜索,我们对长度> 1200 bp的外显子以1000 bp为单元进行切割,对每个外显子或每个切割后的单元构建CM模型并进行搜索。
最后,对一个外显子(或一个1000 bp的搜索单元)是否有同源序列,按以下条件判定:(1)搜索结果的长度;(2)搜索结果的Infernal分数。而对一个人类lncRNA是否有可能的同源基因,按以下条件判定:(1)所有外显子的同源序列连续分布在同一条染色体的同一条链上;(2)同源外显子数目必须至少占长链非编码RNA外显子数目总和的50%。
1.5 数据库软件与环境LongMan数据库采用MYSQL 5.1在CentOS 6.5环境下构建,web服务器采用Apache HTTP Server,web程序在基于PHP5的Symfony框架下开发。数据库结构由包括基因、同义名、转录本、外显子、转座子等在内的多个数据表构成。例如,“基因”数据表包括基因ID、序列名称、数据来源、基因起始地址以及所在链等字段。数据库的主要结构见图 1。
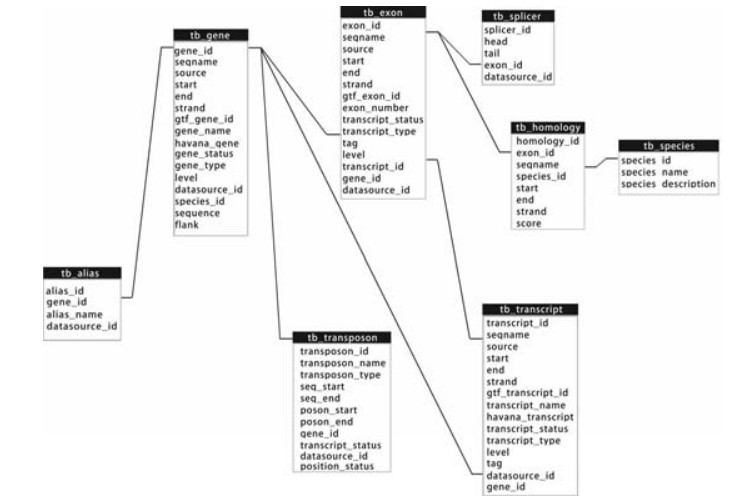
|
图 1 LongMan数据库的主要数据结构 Figure 1 Main data structure of LongMan. |
目前LongMan数据库的beta版收录了GENCODE 18中的13562个人类lncRNA以及用Infernal搜索获得的这13562个lncRNA基因在16个哺乳动物中的同源序列。图 2展示了主要搜索结果,其揭示由原猴亚目(prosimians)到类人猿(simians)lncRNA基因的数量有显著增加,提示大量人类lncRNA与其说是灵长类特有的[12],不如说是类人猿特有的,为研究人类lncRNA的起源和功能提供了重要信息。此外,在从家兔到啮齿类的分枝里,人类lncRNA的同源基因数量不断减少,从rabbit的7230个到mouse的4416个和rat的4099个,提示随着啮齿类的进化它们与灵长类同源的lncRNA越来越少。再者,在有袋类哺乳动物负鼠(opossum)和更原始的哺乳动物鸭嘴兽(platypus),人类lncRNA的同源基因数量极其稀少,提示许多lncRNA在有袋类哺乳动物有独立起源[16]。值得注意的是,劳亚兽总目(Laurasiatheria)和非洲兽总目(Afrotheria)有相当多的人类lncRNA的同源基因,提示许多lncRNA在真哺乳动物起源后可能随着进化而在一些种系分枝(如啮齿类)逐渐丢失了。

|
图 2 人类lncRNA同源基因在各物种基因组中的分布 Figure 2 Distribution of homologs of human lncRNA in multiple mammalian genomes. |
LongMan还收录了若干其它lncRNA数据库的数据,主要是lncRNAdb、NONCODE等中的数据和注释信息。此外LongMan数据库还将允许用户提交lncRNA数据及注释信息。上述数据和特征使LongMan成为迄今最全面的lncRNA同源基因数据库。一个特别重要的lncRNA是ANRIL(也称CDKN2B-AS),它调控CDKN2A/ARF/CDKN2B的表达,表现出特别的种系特异性和进化特征(图 3)[17]。

|
图 3 LncRNA ANRIL在哺乳动物中的同源序列 Figure 3 Homologs of lncRNA ANRIL in mammals (see 2013He S et al [17] for more details). |
LncRNA的重要特征之一是包含大量转座子,尤其是种系特异性转座子[17-18],这些转座子对lncRNA的形成、进化与功能具有重要作用[17, 19]。我们分析了每个同源lncRNA是否包含转座子以及转座子的类别与序列,并将这些信息存储于数据库中。
对lncRNA同源基因初步分析与注释的另一个方面是剪切信号,它们为判定一个保守的同源序列是否为一个外显子提供了重要信息。我们分析了同源lncRNA内含子中的经典剪切信号,并将这些信息存储于数据库中。
2.3 数据检索与下载功能为了方便用户快速查阅lncRNA的信息,LongMan允许用户定义多属性多条件查询,通过多个关键词提高数据库搜索的效率和精度。LongMan数据库还提供了数据下载功能,允许用户批量下载数据。
2.4 同源序列比对与显示LongMan实现了方便的可视化显示功能,能够显示lncRNA基因的重要属性,包括序列、转座子和剪切信号等,并允许用户对图形化显示进行移动和缩放,将lncRNA序列放大到碱基级别或粗略到外显子级别(图 4)。

|
图 4 LongMan的数据显示界面示意图 Figure 4 Graphic display of search results in LongMan. |
LongMan按不同周期实行更新,将每季度根据相关数据库(lncRNAdb等)的数据进行增补更新,每半年根据转座子数据库(RepeatMasker等)进行转座子注释更新,每年按GENCODE数据库进行记录更新。
3 讨论本研究的一个重要问题是Infernal搜索是否产生可靠的直系同源lncRNA序列。对Infernal的有关分析及我们先前的工作均提示Infernal是可靠的RNA序列搜索软件[17]。除了人类,GENCODE项目也系统鉴定了小鼠的lncRNA,Airn和H19在human和mouse的已知结果也为我们的搜索结果提供了支持例证。由于起源不同,人类Airn在小鼠没有同源序列(图 5A);而人类Airn的每一个外显子也确实没有在小鼠的同源区域搜索到同源序列。与之相反,H19是一个保守度高的lncRNA,我们的搜索结果表明,人类H19的exon2对应小鼠H19的exon1(重合度60.7%),人类H19的exon3对应小鼠H19的exon2(重合度100%),人类H19的exon4对应小鼠H19的exon3+exon4(重合度100%+99.7%),图 5B显示了Infernal搜索结果在小鼠同源区域的情况,鉴于lncRNA大多有数个转录本,我们对所有转录本进行了合并以确保没有遗漏信息。
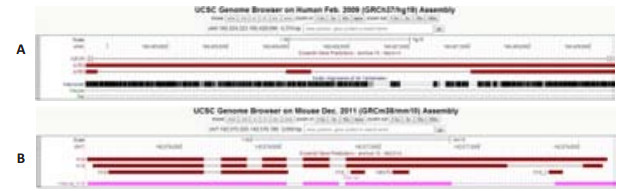
|
图 5 Airn(高度种系特异)与H19(高度保守)的Infernal搜索结果 Figure 5 Infernal search results of Airn (highly species-specific) and H19 (highly conserved). A: The whole-genome alignments indicate that human Airn has an orthologous region in marmoset but not in mouse or rat. Consistent with the alignment result, Infernal only identified orthologous sequences of human Airn in marmoset but not in mouse or rat; B: The pink track indicates that the Infernal-identified human H19 in mouse overlaps exactly with the GENCODE-identified mouse H19. |
GENCODE项目的最新研究揭示人类基因组存在多达数万的lncRNA基因,这些新基因对人类进化、生理与疾病具有重要作用,它们所调控的表观基因组修饰是许多疾病发生与发展的重要机制。基于同源基因的基因序列分析是基因功能研究的重要前提,根据大量同源基因的序列可有效确定序列的保守性、保守段和种系特异性插入和缺失,进而分析基因的功能域。
使用RNA-seq可鉴定一个物种的lncRNA。但由于lncRNA表达具有高度的组织特异性,对少量组织测序无法可靠鉴定某物种的lncRNA,而对大量组织测序则花费过于昂贵。大量研究证明使用计算方法可以鉴定和分析lncRNA同源基因,且具有较好的可行性和经济性,可分析大量lncRNA。我们构建的人类lncRNA同源序列数据库LongMan不仅具有基因多、物种多的特点,而且包含了许多次级信息,是目前全面收录lncRNA同源基因的公开、免费数据库(http://lncrna.smu.edu.cn),能够为lncRNA研究提供有力的支持和帮助。
| [1] |
Morlando M, Ballarino M, Fatica A, et al. The role of long noncoding RNAs in the epigenetic control of gene expression[J].
Chem Med Chem,2014, 9 (3) : 505-10.
DOI: 10.1002/cmdc.201300569. ( 0) 0)
|
| [2] |
Singh DK, Prasanth KV. Functional insights into the role of nuclear-retained long noncoding RNAs in gene expression control in mammalian cells[J].
Chromosome Res,2013, 21 (6/7) : 695-711.
( 0) 0)
|
| [3] |
Spizzo R, Almeida MI, Colombatti A, et al. Long non-coding RNAs and cancer:a new frontier of translational research?[J].
Oncogene,2012, 31 (43) : 4577-87.
DOI: 10.1038/onc.2011.621. ( 0) 0)
|
| [4] |
Congrains A, Kamide K, Oguro R, et al. Genetic variants at the 9p21 locus contribute to atherosclerosis through modulation of ANRIL and CDKN2A/B[J].
Atherosclerosis,2012, 220 (2) : 449-55.
DOI: 10.1016/j.atherosclerosis.2011.11.017. ( 0) 0)
|
| [5] |
Johnson R. Long non-coding RNAs in Huntington's disease neurodegeneration[J].
Neurobiol Dis,2012, 46 (2) : 245-54.
DOI: 10.1016/j.nbd.2011.12.006. ( 0) 0)
|
| [6] |
Hezroni H, Koppstein D, Schwartz MG, et al. Principles of long noncoding RNA evolution derived from direct comparison of transcriptomes in 17 species[J].
Cell Rep,2015, 11 (7) : 1110-22.
DOI: 10.1016/j.celrep.2015.04.023. ( 0) 0)
|
| [7] |
Ulitsky I, Shkumatava A, Jan CH, et al. Conserved function of lincRNAs in vertebrate embryonic development despite rapid sequence evolution[J].
Cell,2011, 147 (7) : 1537-50.
DOI: 10.1016/j.cell.2011.11.055. ( 0) 0)
|
| [8] |
Quek XC, Thomson DW, Maag JL, et al. ncRNAdb v2.0:expanding the reference database for functional long noncoding RNAs[J].
Nucleic Acids Res,2015, 43 (Database issue) : D168-73.
( 0) 0)
|
| [9] |
Chen G, Wang Z, Wang D, et al. LncRNADisease:a database for long-non-coding RNA-associated diseases[J].
Nucleic Acids Res,2013, 41 (Database issue) : D983-6.
( 0) 0)
|
| [10] |
Yang JH, Li JH, Jiang S, et al. ChIPBase:a database for decoding the transcriptional regulation of long non-coding RNA and microRNA genes from ChIP-Seq data[J].
Nucleic Acids Res,2013, 41 (Database issue) : D177-87.
( 0) 0)
|
| [11] |
Zheng LL, Li JH, Wu J, et al. deepBase v2.0:identification, expression, evolution and function of small RNAs, LncRNAs and circular RNAs from deep-sequencing data[J].
Nucleic Acids Res,2016, 44 (D1) : D196-202.
DOI: 10.1093/nar/gkv1273. ( 0) 0)
|
| [12] |
Derrien T, Johnson R, Bussotti G, et al. The GENCODE v7 catalog of human long noncoding RNAs:analysis of their gene structure, evolution, and expression[J].
Genome Res,2012, 22 (9) : 1775-89.
DOI: 10.1101/gr.132159.111. ( 0) 0)
|
| [13] |
Fujita PA, Rhead B, Zweig AS, et al. The UCSC genome browser database:update 2011[J].
Nucleic Acids Res,2011, 39 (Database issue) : D876-82.
( 0) 0)
|
| [14] |
Lorenz R, Bernhart SH, Höner Zu Siederdissen C, et al. ViennaRNA package 2.0[J].
Algorithms Mol Biol,2011, 6 : 26.
DOI: 10.1186/1748-7188-6-26. ( 0) 0)
|
| [15] |
Nawrocki EP, Kolbe DL, Eddy SR. Infernal 1.0:inference of RNA alignments[J].
Bioinformatics,2009, 25 (10) : 1335-7.
DOI: 10.1093/bioinformatics/btp157. ( 0) 0)
|
| [16] |
Grant J, Mahadevaiah SK, Khil P, et al. Rsx is a metatherian RNA with Xist-like properties in X-chromosome inactivation[J].
Nature,2012, 487 (746) : 254-8.
( 0) 0)
|
| [17] |
He S, Gu W, Li Y, et al. ANRIL/CDKN2B-AS shows two-stage clade-specific evolution and becomes conserved after transposon insertions in simians[J].
BMC Evol Biol,2013, 13 : 247.
DOI: 10.1186/1471-2148-13-247. ( 0) 0)
|
| [18] |
Kelley D, Rinn J. Transposable elements reveal a stem cell-specific class of long noncoding RNAs[J].
Genome Biol,2012, 13 (11) : R107.
DOI: 10.1186/gb-2012-13-11-r107. ( 0) 0)
|
| [19] |
Kapusta A, Kronenberg Z, Lynch VJ, et al. Transposable elements are major contributors to the origin, diversification, and regulation of vertebrate long noncoding RNAs[J].
PLoS Genet,2013, 9 (4) : e1003470.
DOI: 10.1371/journal.pgen.1003470. ( 0) 0)
|
 2016, Vol. 36
2016, Vol. 36
