2. 南方医科大学 病理系//广东省分子肿瘤病理学重点实验室, 广东 广州 510515 ;
3. 南方医科大学 南方医院, 广东 广州 510515
2. Department of Pathology, College of Basic Medical Sciences, Key Laboratory of Molecular Tumor Pathology of Guangdong Province, Southern Medical University, Guangzhou 510515, China ;
3. Nanfang Hospital, Southern Medical University, Guangzhou 510515, China
肿瘤细胞的无限增殖能力以及高度侵袭性是其区别于正常体细胞的主要特征[1-2],恶性肿瘤全身重要器官转移以及晚期继发的多器官功能衰竭是影响临床患者预后的最主要因素。而从分子基础水平探讨,与正常体细胞相比,肿瘤细胞高度增殖、迁移、侵袭能力是受到不同的原癌基因激活以及抑癌基因失活等紊乱调控,从而导致的异常生命活动后果,这一系列活动都离不开细胞骨架系统的参与[3]。了解肿瘤发展过程中,与正常细胞有序的骨架活动行为相比较,高度动态紊乱的肿瘤细胞骨架调节机制,发展针对细胞骨架信号通路的特异性分子抗体,对实现肿瘤的分子靶向治疗有潜在的积极意义。
细胞骨架是细胞内部的纤维蛋白网络,其在维持细胞正常形态、承受外界压力、保持细胞内部结构的有序位性方面起重要作用,而且直接参与调控了多种重要的细胞功能活动。小G蛋白家族成员是细胞骨架调节活动的主要调节分子,其中,Rho GTPase是普遍存在于真核生物体内的一类小G蛋白家族亚家族,CDC42作为该亚家族研究比较多的3个主要成员(RAC、Rho和CDC42)之一,曾被证实参与调控了肿瘤细胞运动过程中多个主要环节,包括运动细胞前端丝状伪足的突起、细胞前端与细胞外基质的锚定链接、细胞内微丝骨架的稳定性等多个方面[ 7, 10- 13]。但是,目前为止,CDC42在多种肿瘤组织包括结直肠癌中的具体作用尚未完全明确,其对结直肠癌发生、发展、患者预后的影响以及影响机制尚处于未知。研究CDC42对结直肠癌细胞骨架运动的具体调节作用以及作用机制,可能为结直肠癌临床治疗提供潜在的分子靶标,对实现针对结直肠癌的靶向治疗有积极意义。
本研究旨在通过分子生物学技术方法构建CDC42干扰载体,转染结直肠癌细胞,进而实现在结直肠癌细胞内CDC42的稳定沉默,为进一步研究CDC42对结直肠癌发生、进展的明确作用以及作用机制提供分子生物学工具;观察CDC42沉默对结直肠癌细胞形态影响,为后续进一步实验提供实践基础。
1 材料和方法 1.1 材料与试剂 1.1.1 细胞株、细菌与载体结直肠癌SW480细胞、大肠杆菌DH5α与真核表达载体GV112均为本实验室保存。
1.1.2 生物试剂限制性内切酶EcoRⅠ/AgeⅠ、特效连接酶、连接buffer购自TOYOBO,DNA Marker购自Fermentas,质粒小量提取以及纯化试剂盒购自Qiagen,质粒中量提取试剂盒、质粒转染用脂质体Lipofectamine2000购自Life Technologies,RNA逆转录及QPCR检测试剂盒购自广州复能生物技术有限公司,全蛋白提取以及BCA蛋白浓度定量检测试剂盒购自南京凯基生物科技公司,兔抗人CDC42及小鼠抗人GAPDH单克隆抗体均购自AbClonal,RPMI 1640培养基购自Hyclone,胎牛血清购自BI。
1.2 方法 1.2.1 细胞培养人结直肠癌SW480细胞用含10%胎牛血清的RPMI 1640培养基,常规置于37 ℃、含5% CO2、饱和湿度的培养箱中培养,细胞生长密度至80%~90%饱和时,用含2.5% EDTA的胰酶消化传代。
1.2.2 CDC42干扰siRNA序列的设计CDC42的CDS区全长573 bp,编码191AA,在序列的不同位点CDC42蛋白经过了α螺旋、β折叠卷曲等二级结构,根据预测蛋白质二级结构域,避开siRNA不可接近的复杂二级结构,在CDS区散在选取4个位点作为siRNA的目标靶点。在各位点逐个碱基之间前后移动进行序列筛选,保证所选4条siRNA靶序列长度19~22 bp,GC含量在40%~50%左右,并逐条经过NCBI blast比对,检测序列的特异性,验证是否为人类其它基因的同源序列。同时选用RNAi设计时公认的scramble序列作为对照,对照序列: TTCTCCGAACGTGTCACGT,使用前再次blast比对验证与CDC42基因是否有同源性。将所选4条siRNA及1条scramble序列,联系广州维真生物技术公司进行化学合成。
1.2.3 CDC42 siRNA片段转染结直肠癌SW480细胞将SiRNA及scramble片段按照1 nmoL:50 μL DEPC水混匀稀释,配制成10μmol/L的工作液,-20 ℃储存备用。将生长状态良好的结直肠癌SW480细胞接种于6孔培养板,常规生长24 h后,待细胞密度约50%~70%时,使用lipofectamine2000阳离子脂质体法瞬时转染细胞。步骤如下:
(1)配制A液及B液每孔各250 μL:
A液:245 μL optiMEM+5 μL lipo2000
B液:245 μL optiMEM+5 μL SiRNA
(2)A、B液配好后,分别室温孵育约5 min;混匀A、B液体,孵育20 min。
(3)去掉含血清的RPMI 1640细胞培养基,换为1.5 mL opti-MEM培养基/每孔。
(4)将孵育20 min后的A,B混合液,按组加入到六孔板细胞内,十字法轻轻摇匀,细胞常规培养4~6 h后换为含10%胎牛血清的RPMI 1640培养基继续培养,48~72 h后提取蛋白质进行Western blotting鉴定。
1.2.4 Western blotting检测并筛选最有效干扰片段分别收集六孔板内经过siRNA瞬时转染的各组目的细胞与对照组细胞,提取细胞全蛋白,BCA法测定蛋白浓度,用lysis buffer调整各组样品浓度至相近水平,按比例加入loading buffer,煮沸变性蛋白质;配置10%的SDS-PAGE胶,在胶内各泳道中,按顺序将处理好的蛋白样品各30 μg进行加样并分离电泳,电泳后切取目的条带湿法转至0.22 μm PVDF膜,5%的脱脂奶粉室温封闭1 h,一定稀释比例的一抗(CDC42 1:1000)4 ℃孵育过夜,PBST清洗5 min×5次,辣根酶标记的二抗(山羊抗兔IgG抗体,1:5000)室温孵育1h,PBST清洗5 min× 5次,ECL曝光观察,选取干扰效果最强的siRNA条带。以GAPDH做内参。
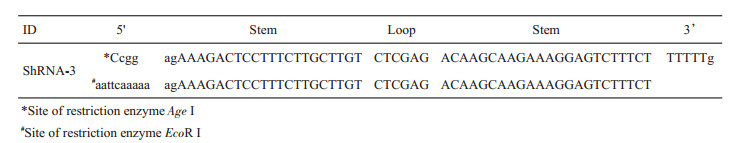
1.2.5 shRNA质粒载体的构建与测序筛选最有效的siRNA片段,以靶标序列为主链,根据碱基互补配对原则,设计siRNA序列所对应的“互补片段”。以此特异性siRNA靶标序列以及其“互补片段”为环状RNA的两端“枝干”,在片段中间插入构建环状RNA所特需的6碱基“茎环”结构,“茎环”结构碱基序列选用结构稳定的“CTCGAG”序列。设计好shRNA茎环与枝干主体结构后,在枝干两端插入EcoRI/AgeI限制性内切酶酶切位点序列,化学合成法逐个碱基合成设计好的的双链Oligo序列。
用特异性快切酶快速双酶切碱基序列(酶切条件: 37 ℃,0.5~1 h),露出BamHⅠ/AgeⅠ粘性末端,与同步双酶切并纯化后的GV112真核表达载体用ligase buffer特效连接(连接条件: 22 ℃,45 min~1 h),连接产物转化DH5α感受态细菌,挑选阳性克隆菌,摇菌后菌液测序鉴定。
1.2.6 shRNA质粒载体转染结直肠癌SW480细胞挑选经测序结果显示构建成功,碱基序列与预期相符和的阳性克隆菌,大量摇菌进行质粒中量提取,所提取质粒用无菌DEPC水溶解,测定质粒浓度,用lipofectimane2000阳离子脂质体法转染结直肠癌SW480细胞。质粒中量提取按照Life Techonologies操作手册进行,阳离子脂质体转染方法同前。
1.2.7 QPCR检测细胞株mRNA水平表达变化细胞瞬时转染后48 h,用PBS清洗目的细胞以及对照组细胞各2次,加入Trizol逐级提取细胞总RNA,逆转录得到cDNA,QPCR分析目的细胞以及对照细胞中CDC42相对表达量。GAPDH做内参。所用引物序列:
CDC42: sense: CCATCGGAATATGTACCGACTG;
antisense: CTCAGCGGTCGTAATCTGTCA,
GAPDH: sense: TGAAGGTCGGAGTCAACGGA;
antisense: CCATTGATGACAAGCTTCCCG
1.2.8 Western blotting检测细胞株蛋白水平表达变化细胞瞬时转染后72 h,收集目的以及对照组细胞,体积细胞蛋白质,Western blotting检测分析目的细胞以及对照细胞株中CDC42的相对表达量,GAPDH做内参。细胞蛋白提取以及Western blotting实验方法同前述。
1.2.9 观察CDC42有效沉默对SW480细胞形态影响将结直肠癌SW480细胞接种到六孔培养板,常规培养24 h后,细胞生长密度达到50%~70%对数生长期时,将CDC42 shRNA瞬时转染进入细胞,继续培养72 h后,光学显微镜观察SW480细胞形态变化。
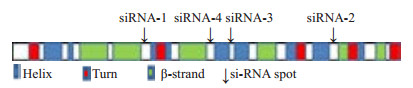
2 结果 2.1 CDC42干扰siRNA序列的设计CDC42的CDS区全长573 bp,编码191AA,根据预测蛋白质二级结构域,避开siRNA不可接近的复杂二级结构域(图 1),在CDS区分散选取4个位点作为siRNA的目标靶点,选用通用scramble序列做对照。4条siRNA靶序列以及对照序列长度、GC含量以及在CDC42基因CDS区内起始位点如下表(表 1),siRNA目标序列逐条经过NCBI blast比对后,未与人类其它的基因同源,scramble序列与CDC42基因无重合同源性。
|
图 1 根据CDC42基因二级结构设计siRNA片段位点 Figure 1 Design of the 4 CDC42 siRNA sequences based on the target gene 2D structure. |
| 表 1 设计CDC42 siRNA序列以及scramble序列具体信息 Table 1 Sequences of the 4 siRNAs targeting CDC42 and the scramble sequence |
Western blotting检测结果显示,在GAPDH内参齐平时,与sramble对照组相比,siRNA-3对CDC42的沉默效果最好。而且以对照组条带作为100%标准值,Western blotting条带灰度值分析结果显示,siRNA-3对CDC42的干扰效率>50%,可以有效沉默CDC42,筛选用于后续实验(图 2)。

|
图 2 Western blotting筛选CDC42最有效siRNA序列 Figure 2 Identification of the most efficient siRNA for knocking down CDC42 by Western blotting. |
以siRNA序列与其碱基互补序列作为shRNA的两侧“枝干”(stem),序列中间通过6碱基“茎环”结构(loop)连接,“枝干”两侧末端辅加EcoRⅠ/AgeⅠ限制性内切酶酶切位点序列(图 3)。所设计序列经过化学合成后,通过快速双酶切露出粘性末端,与真核表达载体连接,连接产物转化大肠杆菌DH5α,挑选阳性克隆菌进行摇菌,菌液进行测序分析。测序结果显示片段在测序茎环处中断前序列与NCBI官方公布序列核对,碱基序列与预期完全一致,提示CDC42基因有效shRNA质粒载体构建成功,可通过大肠杆菌DH5α进行稳定扩增后提取以进行后续长期实验。
|
图 3 根据CDC42 siRNA-3设计shRNA载体序列 Figure 3 Design of CDC42 shRNA sequences based on the siRNA-3 structure. |
QPCR对mRNA水平检测结果显示,与scramble对照组相比,转染CDC42 shRNA载体的细胞,CDC42 mRNA表达明显下降,差异具有统计学意义。
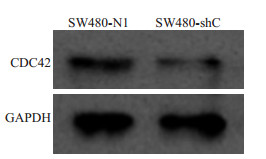
Western blotting对蛋白水平表达检测结果显示,在GAPDH内参齐平时,与scramble对照组相比,转染CDC42 shRNA载体后的细胞,CDC42蛋白表达明显降低(图 4)。二者结果综合提示结直肠癌SW480细胞在瞬时转染了CDC42 shRNA后,CDC42基因表达被有效沉默。提示CDC42基因的干扰载体构建成功,转染结直肠癌后可以有效沉默CDC42基因表达,可用于后期进一步观察研究CDC42沉默对结直肠癌细胞生物学表型、细胞形态、功能的影响。
|
图 4 WB验证SW480细胞中CDC42沉默效率 Figure 4 Detection of CDC42 protein expression in SW480.shCDC/NC cells with Western blotting. |
将CDC42 shRNA载体瞬时转染SW480细胞后,常规培养72 h,光学显微镜下观察细胞,发现在CDC42被沉默后,SW480细胞前端伪足伸出明显减少,细胞周边变的光滑,细胞整体“占地面积”减小,提示细胞内微丝骨架伸展减弱,运动细胞前端与细胞外基质的连接出现改变,细胞运动能力可能因此减弱(图 5)。

|
图 5 CDC42沉默后结直肠癌SW480细胞形态变化 Figure 5 SW480 cells transfected with CDC42 shRNA vector. A: SW480-N1 cells (Original magnification: ×200); B: SW480-N1 cells (Original magnification: × 400); C: SW480.shCDC42 cells (Original magnification: × 200); D: SW480. shCDC42 cells (Original magnification: ×400). |
恶性肿瘤与心脑血管病、糖尿病为我国目前发病率最高的3大慢性疾病,并称人类3大“健康杀手” [14-15],而其中,恶性肿瘤发病率依旧在逐年攀升,临床患者死亡率居高不下。其无限增值、转移活动离不开精细的细胞骨架调节。系统的研究肿瘤发生、发展过程中细胞骨架的调节活动,从而发展针对细胞内信号通路终末端—骨架调节活动的特异性分子抗体,对实现肿瘤的分子靶向治疗有积极意义。
肿瘤细胞的迁移运动包括4个主要环节,细胞前端伪足的伸出突起,细胞伸出前端与细胞外基质锚定连接,细胞胞体向前定向移动,细胞末端从原细胞位点的脱离向前[16-19]。CDC42属于小G蛋白家族Rho GTPases亚家族3个主要成员之一,被广泛证实参与调控了这一系列肿瘤细胞迁移运动过程中多个主要环节,包括运动细胞前端丝状伪足的突起、细胞前端与细胞外基质的锚定链接,以及可以与另一个主要成员RhoA联合调控细胞胞体收缩过程中细胞内微丝骨架的稳定性等多个方面[20-23]。
而且通过直接检测基因表达量,也证实CDC42在多种肿瘤,包括淋巴瘤、肺腺癌、肝癌中相较正常组织中表达明显异常增高,这些都提示CDC42基因与肿瘤的发生、发展具有密切相关性[24-25]。但是在消化系统肿瘤中,CDC42基因的异常表达与结直肠癌的关系目前尚未完全明确,而且人为的实现对CDC42基因靶向干扰技术,目前尚没有相关文献见诸报端。本研究旨在靶向沉默CDC42的表达,并观察CDC42基因对结直肠癌细胞生物学行为特性的影响,对进一步研究CDC42对结直肠癌发生、发展的影响作用以及进一步研究具体的作用机制提供研究基础。
RNA干扰是目前国内外发展都已经相当成熟的,利用小分子RNA片段(siRNAs)实现对同源mRNA的特异性降解的分子技术。其可以实现对同源mRNA的特异性、而且非常高效的切割,实现对靶蛋白表达的有效沉默[26-33]。本课题组直接通过外源性转入化学合成的双链siRNA序列,外源性siRNA进入体内后可以利用细胞内RNA酶解链形成单体,通过单体siRNA片段与CDC42基因CDS区同源序列的特异性互补结合,从结合位点降解靶mRNA,从而实现对目标基因蛋白的特异性表达敲低。为了筛选最有效的siRNA位点,我们结合蛋白质的二级结构域,在不同的CDC42 CDS区碱基位点,设计了4条siRNA,并将其分别瞬时转染结直肠癌SW480细胞,筛选得到了可以将CDC4最有效沉默的siRNA片段。
为了实现后续对CDC42的长期稳定敲低,我们进一步在siRNA片段两端插入“茎环”(loop)结构以及限制性内切酶酶切位点,将基因片段通过酶切后再连接技术插入真核表达载体。因为GV112载体骨架采用适合表达小分子RNA的U6启动子,而不同于其它常用的真核表达载体骨架,如pCDNA3.0内所采用的是更适合长片段基因表达的CMV启动子,被我们首选考虑为CDC42 shRNA构建的骨架载体。CDC42 shRNA质粒酶切再连接后,基因序列经过测序验证,证实与预期完全符合,提示载体构建成功。成功构建的CDC42 shRNA质粒载体转化感受态大肠杆菌DH5α后,可以稳定扩增以大量提取质粒,质粒转染结直肠癌SW480细胞后,QPCR与Western blotting验证结果证实,与阴性对照组细胞相比,瞬时转染后的细胞CDC42 mRNA与蛋白表达均出现明显降低,差异具有统计学意义,提示结直肠癌内CDC42有效沉默载体构建成功,为后续长期进行CDC42沉默对结直肠癌生物学表型、功能的影响作用以及作用机制提供了分子工具。
而且,细胞形态学观察结果显示,CDC42沉默后,结直肠癌SW480细胞前端伪足伸出减少,细胞周边变的“光滑”,而且细胞“占地面积”减少。结合肿瘤细胞的迁移运动所必须的主要骨架活动环节,如细胞前端伪足的伸出突起、进而与细胞外基质锚定连接,以及后续细胞胞体整体前移[16-19]等主要步骤,CDC42沉默后结直肠癌细胞形态变化,高度提示CDC42沉默导致细胞内微丝骨架延伸减弱,运动细胞前端与细胞外基质的连接出现改变。肿瘤细胞运动能力可能因此减弱,为后续进一步研究CDC42通过调节细胞骨架稳定性影响结直肠癌的发生、发展、转移作用奠定初步的实践基础。
综上所述,本实验成功构建了小G蛋白家族成员蛋白CDC42的稳定干扰载体,并转染结直肠癌SW480细胞系。根据形态学结果初步可以推测CDC42可能通过调节细胞骨架活动影响结直肠癌的发生、发展,为后续深入研究CDC42在结直肠癌发生、发展中的具体作用以及作用机制提供了分子工具,对寻找针对肿瘤细胞骨架信号通路的潜在分子治疗靶点,实现对结直肠癌的特异性治疗有积极意义。
| [1] |
Hainaut P, Plymoth A. Targeting the hallmarks of cancer:towards a rational approach to next-generation cancer therapy[J].
Curr Opin Oncol,2013, 25 (1) : 50-1.
DOI: 10.1097/CCO.0b013e32835b651e. ( 0) 0)
|
| [2] |
Hanahan D, Weinberg RA. Hallmarks of cancer:the next Generation[J].
Cell,2011, 144 (5) : 646-74.
DOI: 10.1016/j.cell.2011.02.013. ( 0) 0)
|
| [3] |
Friedl P, Alexander S. Cancer invasion and the microenvironment:plasticity and reciprocity[J].
Cell,2011, 147 (5) : 992-1009.
DOI: 10.1016/j.cell.2011.11.016. ( 0) 0)
|
| [4] |
Zugasti O, Rul W, Roux P, et al. Raf-MEK-Erk cascade in anoikis is controlled by Rac1 and Cdc42 via Akt[J].
Mol Cell Biol,2001, 21 (19) : 6706-17.
DOI: 10.1128/MCB.21.19.6706-6717.2001. ( 0) 0)
|
| [5] |
Ge Q, Chen L, Jaffar J, et al. Fibulin1C peptide induces cell attachment and extracellular matrix deposition in lung fibroblasts[J].
Sci Rep,2015, 5 (3) : 9496.
( 0) 0)
|
| [6] |
Bao W, Thullberg M, Zhang H, et al. Cell attachment to the extracellular matrix induces proteasomal degradation of p21(CIP1) via Cdc42/Rac1 signaling[J].
Mol Cell Biol,2002, 22 (13) : 4587-97.
DOI: 10.1128/MCB.22.13.4587-4597.2002. ( 0) 0)
|
| [7] |
Sharma M, Urano F, Jaeschke A. Cdc42 and Rac1 are major contributors to the saturated fatty acid-stimulated JNK pathway in hepatocytes[J].
J Hepatol,2012, 56 (1) : 192-8.
DOI: 10.1016/j.jhep.2011.03.019. ( 0) 0)
|
| [8] |
Hines R, Markossian T, Johnson A, et al. Geographic residency status and census tract socioeconomic status as determinants of colorectal cancer outcomes[J].
Am J Public Health,2014, 104 (3) : e63-71.
DOI: 10.2105/AJPH.2013.301572. ( 0) 0)
|
| [9] |
Henry KA, Sherman RL, Mcdonald K, et al. Associations of census-tract poverty with subsite-specific colorectal cancer incidence rates and stage of disease at diagnosis in the United States[J].
J Cancer Epidemiol,2014, 14 (8) : 823484.
( 0) 0)
|
| [10] |
Begum R, Nur-E-Kamal MS, Zaman MA. The role of Rho GTPases in the regulation of the rearrangement of actin cytoskeleton and cell movement[J].
ExpMol Med,2004, 36 (4) : 358-66.
( 0) 0)
|
| [11] |
Pouwels J, Van DN, Van Lent J, et al. The cytoskeleton and the secretory pathway are not involved in targeting the cowpea Mosaic virus movement protein to the cell periphery[J].
Virology,2002, 297 (1) : 48-56.
DOI: 10.1006/viro.2002.1424. ( 0) 0)
|
| [12] |
Giretti MS, Simoncini T. Rapid regulatory actions of sex steroids on cell movement through the actin cytoskeleton[J].
Steroids,2008, 73 (9/10) : 895-900.
( 0) 0)
|
| [13] |
Jiang P, Enomoto A, Takahashi M. Cell biology of the movement of breast cancer cells:intracellular signalling and the actin cytoskeleton[J].
Cancer Lett,2009, 284 (2) : 122-30.
DOI: 10.1016/j.canlet.2009.02.034. ( 0) 0)
|
| [14] |
Fu XD, Flamini M, Sanchez AM, et al. Progestogens regulate endothelial actin cytoskeleton and cell movement via the actin-binding protein moesin[J].
Mol Hum Reprod,2008, 14 (4) : 225-34.
DOI: 10.1093/molehr/gan010. ( 0) 0)
|
| [15] |
Chen Y, Yang Z, Meng M, et al. Cullin mediates degradation of RhoA through evolutionarily conserved BTB adaptors to control actin cytoskeleton structure and cell movement[J].
Mol Cell,2009, 35 (6) : 841-55.
DOI: 10.1016/j.molcel.2009.09.004. ( 0) 0)
|
| [16] |
Small JV, Rottner K, Kaverina I, et al. Assembling an actin cytoskeleton for cell attachment and movement[J].
Biochim Biophys Acta,1998, 1404 (3) : 271-81.
DOI: 10.1016/S0167-4889(98)00080-9. ( 0) 0)
|
| [17] |
Lim HS, Lee MY, Moon JS, et al. Actin cytoskeleton and golgi involvement in barley stripe Mosaic virus movement and cell wall localization of triple gene block proteins[J].
The Plant Pathology J,2013, 29 (1) : 17-30.
DOI: 10.5423/PPJ.OA.09.2012.0144. ( 0) 0)
|
| [18] |
Giretti M S, Simoncini T. Rapid regulatory actions of sex steroids on cell movement through the actin cytoskeleton[J].
Steroids,2008, 73 (9-10) : 895-900.
DOI: 10.1016/j.steroids.2008.01.011. ( 0) 0)
|
| [19] |
Jiang P, Enomoto A, Takahashi M. Cell biology of the movement of breast cancer cells:intracellular signalling and the actin cytoskeleton[J].
Cancer Lett,2009, 284 (2) : 122-30.
DOI: 10.1016/j.canlet.2009.02.034. ( 0) 0)
|
| [20] |
Chang H. RNAi-mediated knockdown of target genes:a promising strategy for pancreatic cancer research[J].
Cancer Gene Ther,2007, 14 (8) : 677-85.
DOI: 10.1038/sj.cgt.7701063. ( 0) 0)
|
| [21] |
Lv W, Zhang C, Zhou DH, et al. RNAi-mediated gene silencing of vascular endothelial growth factor inhibits growth of colorectal cancer[J].
Cancer BiotherRadiopharm,2007, 22 (6) : 841-52.
( 0) 0)
|
| [22] |
Lee AJ, Kolesnick R, Swanton C. RNAi-mediated functional analysis of pathways influencing cancer cell drug resistance[J].
Expert Rev Mol Med,2009, 11 (8) : e15.
( 0) 0)
|
| [23] |
Dave RS. RNAi and tumor angiogenesis:bridging the gap towards anti-cancer therapy[J].
Leuk Res,2007, 31 (4) : 421-2.
DOI: 10.1016/j.leukres.2006.10.024. ( 0) 0)
|
| [24] |
Romano N, Macino G. Quelling:transient inactivation of gene expression in Neurosporacrassa by transformation with homologous sequences[J].
Mol Microbiol,1992, 6 (22) : 3343-53.
DOI: 10.1111/mmi.1992.6.issue-22. ( 0) 0)
|
| [25] |
Deveraux QL, Aza-Blanc P, Wagner KW, et al. Exposing oncogenic dependencies for cancer drug target discovery and validation using RNAi[J].
Semin Cancer Biol,2003, 13 (4) : 293-300.
DOI: 10.1016/S1044-579X(03)00043-9. ( 0) 0)
|
| [26] |
Gil-Ranedo J, Mendiburu-Eliçabe M, García-Villanueva M, et al. An off-target nucleostemin RNAi inhibits growth in human glioblastoma-derived cancer stem cells[J].
PLoS One,2011, 6 (12) : e28753.
DOI: 10.1371/journal.pone.0028753. ( 0) 0)
|
| [27] |
Deng KX, Zhong L, Jiang MX, et al. Effect of RNAi-mediated survivin gene silencing on apoptosis of ovarian cancer cell lines SKOV3 and SKOV3/ADM[J].
Ai Zheng,2005, 24 (8) : 945-50.
( 0) 0)
|
 2016, Vol. 36
2016, Vol. 36
