中南大学 公共卫生学院,湖南 长沙 410083 ;
3. 蚌埠医学院 公共课程部,安徽 蚌埠 233003 ;
4. 蚌埠医学院 病原生物学教研室,安徽 蚌埠 233003
1Instituteof Information Security and Big Data, 2School of Public Health, Central South University, Changsha 410083, China ;
3. 3Department of Public Courses, 4Department of Microbiology and Parasitology, Bengbu Medical College, Bengbu 233003, China ;
4. 3Department of Public Courses, 4Department of Microbiology and Parasitology, Bengbu Medical College, Bengbu 233003, China
科学文献为学者提供了一个巨大的信息财富,它既可以作为特定领域研究的起点,也可以作为新的研究思路的信息来源[1]。在海量的生物医学文献中,生物实体之间存在大量的关联,对这些异构数据进行系统分析给生物学家带来前所未有的机遇[2],使得他们能够在个性化医疗与转化医学背景下,推断不同生物实体间的关联程度[3],然而,这些关联是非常复杂且稀疏的,直接查询的计算量非常具有挑战性。
最早也是最著名的利用文献挖掘算法挖掘实体关联的是Swanson,他意识到研究人员的专业性越来越强,但是文献阅读却不够专业,使得文献成为信息孤岛,交互性低,所以他引入ABC理论促进知识发现,以潜在知识识别推断生没有直接关联的生物实体。同时,他也强调这种文献挖掘方法只是辅助科学假设或对假设生成的支持,若要证实这种关联必须通过科学严谨的生物实验来证明[4]。Swanson[5-6]用ABC理论得到鱼油与雷诺氏病,以及镁与偏头痛具有关联的假设,并最终用生物医学实验证明了其中的关联。目前,生物实体关联的研究有:蛋白质与蛋白质的关联[7-8],蛋白质与基因的关联[9],药物与药物的关联[10],药物与疾病的关联[11]等。但尚无文献基于文本挖掘对乳腺癌相关基因药物间的关联进行过研究报道。
过去的十年里,在文献中基于网络的计算方法已得到普及,并成为一个研究药物疾病基因关联的新范式。这些方法的应用包括疾病候选基因的排序[12-13],鉴定疾病之间的关联[14-15]和药物再定位[16-17]等。例如,Hu和Agarwar[18]从基因表达数据库(Gene Expression Omnibus)中收集数据,创建了基于基因组表达谱的人类疾病药物网络。Bauer等[19]通过整合多种来源,开发了一个综合的疾病基因关联网络,用于揭示不同疾病间的关联。为了系统地分析药物疾病基因之间的关联,Daminelli等[20]通过在网络中完善不完全双派系,提出了一种基于网络的新型的预测药物与基因、药物与疾病间关联的方法,这种方法对药物再定位和发现药物潜在的新关联具有极大的帮助。
基于网络的计算方法能够通过将文献中有用信息分解成小型子网模型的方法来分析整个异构网络(如药物疾病基因网络),这种模式称之为网络基序(network motifs, NMs)[21]。NMs是具有统计学意义的重复性结构模式,是生物网络中具有基本功能和保守进化的最小单位,是重要的子网模式,代表了网络的骨干,是节点(如:基因、药物)[22-23]重要组成部分,这些NMs也可以形成一个大型汇总模块,利用在重叠的NMs中形成的关联来实现特定的功能,挖掘隐含关联。将这些复杂网络模型可视化,并基于关联度评估来定义表达量间的相似性,从而形成数据分析的网络范式,有利于对网络节点间相互作用关系的复杂系统和高维数据进行分析[24]。
本文基于ABC理论和关联规则的文本挖掘算法来获取文献中生物实体间的关联,并基于网络分析所得关联模型。以乳腺癌为例,先从PubMed数据库中获取乳腺癌相关生物医学文献,通过数据清洗,得到乳腺癌基因与基因、药物与药物间的关联,再使用ABC理论和关联规则对乳腺癌基因药物之间是否存在关联以及关联的程度进行量化,然后使用R语言实现网络模型可视化,最后分析了网络节点关联和模型结构,用ROC曲线验证了本文算法的可靠性,同时提出了实验性的研究假设,为科研人员对今后乳腺癌相关的诊断与治疗、疾病候选基因的筛选、靶向药物、药物再定位和个性化医疗等提供研究依据和研究思路。同样,也可将本算法模型运用于分析其他临床疾病。
1 资料和方法 1.1 词典与文献资料首先,分别从Entrez GENE[25-26]、Gene Ontology[27]、OMIM[28]、DrugBank[29]等重要数据库中获取并建立Gene、Drug标准词典,命名为“Gene_Dictionary”(共计40172个人类基因词条)和“Drug_dictionary”(共计1763种药物词条)词典,词典包括每个基因或药物的标准名称、别名、同义词、标准编号等属性,在研究过程中需要以这2个权威词典为基准来过滤文献。
然后在PubMed数据库中使用“" breast neoplasms" [MeSH Terms]AND(" 2013/09/01" [PDAT]: " 2015/ 09/01" [PDAT])”为检索策略,获取近2年内与乳腺癌有关的文献共计17037篇,并以txt格式保存至本地磁盘,这是本文主要研究对象。
1.2 数据清洗由于生物医学文献专业性词汇较多,若要进行文本(如摘要)挖掘,须先对其进行数据清洗,结合实际需求,本研究采用以下算法进行预处理。
(1)由于大小写不影响本文最终处理结果,所以先将文献所有英文字母全部转为小写;(2)把文本转化为单独句子;(3)采用文本标准化定义分割每个句子,使之变成规范文本;(4)去除标点符号以及无意义词,如:“the”、“a”、“from”、“to”等;(5)将希腊字母变为英文音译,如“:α→Alpha”等;(6)对比规范文本与词典对象名称,若2者相同或与词典中别名、编号等相同,则可以认定发现了一个实体对象;(7)对于已发现的实体,在文献中基于网络模式分析提取上下文实体对象的关联;(8)词项集(即最终得到的实体集合,如:乳腺癌基因词集)以字母排序。
通过上述数据清理算法,将收集到的乳腺癌相关文献处理成规范文本项集,依据已知的“Gene_Dictionary”和“Drug_dictionary”这2个词典来处理、合并文献中所需要提取的基因、药物同义词,将其替换为统一标准,以便获取这些生物实体间的关联。
1.3 ABC理论共现的方法可以确定2种生物实体概念间的关联,若它们出现在同一文章时,则可以认为这两者具有关联。目前基于共现来寻找2者之间隐含关联的最主要算法就是ABC理论,其基本思想是:假设A和C都与B有关联,那么A、C之间就可能存在关联,而且可能这种关联甚至是从未发现过的。
Frijters[30]对“ABC”理论加以改进,将通过对实体A、C关联的假设来确认与量化隐藏在海量生物医学文献中生物实体间的关联的过程,称之为“封闭探索(Closed Discovery)”进程;在这个进程中,若A、C之间存在关联,那么在文献中挖掘出共享的生物实体概念B来支持这个假设,这个过程称之为“开放探索(Open Discovery)”进程(图 1)。
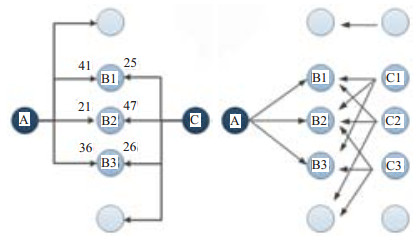
|
图 1 文献内实体之间隐藏关联的ABC原理 Figure 1 ABC principle of hidden relationships in literature. Hidden relationships in literature between biomedical concepts(e.g., genes, drugs), for which A and C have no direct relationship, but are connected indirectly via B-intermediates, can be analyzed in a closed discovery by summation of the Relevance Score of the weakest links, divided by the number of intermediates. |
与Frijters的ABC理论不同的是,本文将其与关联规则相结合,算法如下:首先基于共现得到A、B间的共现频次,然后使用ABC理论推断是否与实体C有关,最后使用关联规则设定阈值,并计算关联程度和优先级,关联程度越高,则2个实体间存在关联的可能性越大;若未见相关文献报道,那么A可能是C的潜在靶点。同时,算法应用范围也有所不同,本文不仅考虑不同类型实体间的关联,对同一类实体间的关联也加以探讨。
1.4 关联规则在对生物实体关联进行度量时,需要用到以下术语和度量指标。
(1)设I={I1, I2, I3, …, Im}是项的集合,设事务相关的数据D是数据库事务的集合,其中每个事务T是一个非空项集,使得T⊆I。每一个事物都有一个标识符,成为TID。设A是一个像集,事务T包含A,当且仅当A⊆T。关联规则是形成如A⇒B的蕴涵式,其中A⊂I,B⊂I,A≠ ,B≠ ,且A∩B≠ 。规则A⇒B在事务集D中成立,具有支持度s,其中s是D中事务包含在A∪B的百分比,它是概率P(A∪B),表示事务包含集合A和B的并的概率。规则A⇒B在事务D中具有置信度c,其中c是D中包含A事务同时也包含B的事务的百分比,即条件概率P(A|B)。
(2)支持度support用于衡量集合内各项出现的频次阈值。
| $ \sup \;port(A \Rightarrow B) = P(A \cup B) = a/N $ |
其中a是词项在所有文献中出现的频次,N为集合A中所有词项在文献中出现的频次总数,两者的比值即可求出某个集合内各项出现的频次。
(3)置信度confidence可以度量关联规则的属性。
| $ confidence(A \Rightarrow B) = P(B|A) = \frac{{\sup \;port(A \cup B)}}{{\sup \;port(A)}} $ |
上式表明规则A⇒B的置信度可以从A和A∪B的支持度计数推出。同时满足最小支持度阈值(min_sup)和最小置信度阈值(min_conf)的规则称为强规则,一般使用0%~100%来表示支持度和置信度。
(4)由于支持度和置信度不足以过滤掉无用的关联规则,可以使用相关性度量来扩充关联规则框架,如下所示:
| $ A \Rightarrow B[sup\;port, confidence, correlation] $ |
我们使用提升度lift作为correlation的相关性度量,而lift定义如下:如果P(A ∪ B)=P(A)P(B),则项集A的出现独立于项集B的出现;否则,项集A和B的事件是相互依赖的和相关的。依据定义,lift能够评估一个预测模型是否有效,体现集合{A}对{B}的重要性,若值为0,说明{A}与{B}之间无关联;若值为正,{B的概率上升;若值为负,{B}的概率下降。
| $ lift(A, B) = \frac{{(P(A \cup B))}}{{P{{(A)}^ * }P(B)}} = ({a^ * }N)/({(a + c)^ * }(a + b)) $ |
如果该值为1,说明A与B是独立的,没有任何关联;若值小于1,说明A与B是负相关,A的出现可能导致B的不出现;若值大于1,则A和B是正相关的,意味着每一个A的出现都蕴涵着B的出现,值越大出现的几率也就越大,即A的出现“提升”B出现的程度,一般认为lift的值越高,其关联规则越有价值[31-32]。在本文中,考虑到实体可能在文献中偶尔或对比提及,不是研究内容,所以设定life阈值为3,这样得到的结果可能会更有意义。
1.5 阈值设定利用Gene词典在已经进行过规范化处理后的乳腺癌词项集进行过滤,考虑到部分基因可能只是在文献中偶尔提及或只是对比介绍,没有具体的研究,所以本文设定乳腺癌基因Support_count的阈值大于等于3;利用Drug词典对下载下来的并已经清理过的乳腺癌文献进行全文检索,并设定药物的Support_count阈值大于等于3。
1.6 网络模型算法基于上述理论,即可构建生物实体网络模型,其拓扑结构包含不同的子网模式,它们具有相同类型的网络特定的处理任务。在关联网络中,所有连接的子网节点整理成同构模式,以及使用模式频率的计数方式。
综上所述,本文构建乳腺癌基因药物网络框架的算法:首先,给定最小支持度阈值,计算出所有大于等于support的项集(本文主要指的是过滤文献后留下的词项集),得到单个item的项集;再次,基于关联度量计算出item的项集内之间的关联,过滤掉那些不满足最小lift阈值的项集;最后,基于第二步和ABC理论生成新item的项集以及它们之间的关联,过滤掉那些不满足最小lift值的项集,得到无向网络模型数据集。
1.7 R语言实现和ROC曲线本文采用R语言这个开源的数据分析系统作为主要的研究工具,它对特定的统计问题具有非常强大的分析与作图能力[33],适用于本研究中的数据清洗、统计分析以及网络模型可视化操作。本文使用ROC曲线判断算法性能。ROC曲线现以广泛应用于医学诊断实验性能的评价,同样也适应于判别模型诊断效果[34]。
2 结果 2.1 乳腺癌基因之间的关联得到185种不同基因及其Support值(表 1)。
| 表 1 部分乳腺癌相关基因及其Support值 Table 1 Part of breast cancer-associated genes and their Support values |
由表 1可以发现,基因ERBB2(鸟类v-erb-b2成红细胞白血病病毒基因同源2,神经/成胶质细胞瘤衍生癌基因同源)在近2年的文献中出现频次最高,属于研究热点。
基于前述网络模式算法,对所有乳腺癌基因之间关联的Lift值进行计算,选取Lift值大于3的基因关联,去重后,最终得到88种不同基因以及它们之间存在的98种不同关联,并以此生成乳腺癌基因网络模型(图 2)。
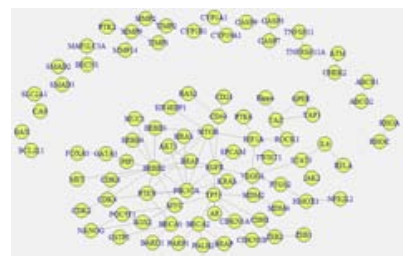
|
图 2 乳腺癌基因网络模型 Figure 2 Breast cancer genes network model. |
图 2的基因关联网络中,大部分节点的度很小,少部分节点(ERBB2等)的度较大,符合幂律分布,属于无标度网络,这种网络的特点就是对随机故障的鲁棒性和针对性蓄意攻击的脆弱性。在生物医学领域中,则说明关键节点的重要性。例如:网络图中的ERBB2与MUC1等11种不同基因存在关联,意味着ERBB2可能在乳腺癌基因相互作用中有着极为重要的地位,也是研究热点。
由图 2中可以看出,单独关联的基因有:ATM和CHEK2、TNFSF11和TNFRSF11A、BCL2L1和BAX、CA9和SLC2A1、SMAD2和SMAD3、MAP1LC3A和BECN1、ABCC2和ABCB1、RHOA和RHOC这8对基因与其它基因没有关联;基因CYP1B1、CYP19A1只与CYP1A1相关,基因CASP9、CASP3只与CASP7相关;网络模型中的相关度较高的基因节点为:ERBB2、EGFR、MTOR、TP53、PLK3CA和BACR2这6种基因,同时这6种基因也是近两年来在乳腺癌疾病方面研究中的热点。
2.2 乳腺癌药物之间的关联得到乳腺癌相关药物共计113种及其Support值(表 2)。
| 表 2 部分乳腺癌相关药物及其Support值 Table 2 Part of breast cancer-associated drugs and their Support values |
基于前述网络模型算法,对所有乳腺癌基因的关联强度与Lift值进行计算,为了更好分析具有高关联度的药物,我们设置Lift的阈值为10,得到97种药物和它们之间的170种高关联,生成乳腺癌药物网络模型(图 3)。
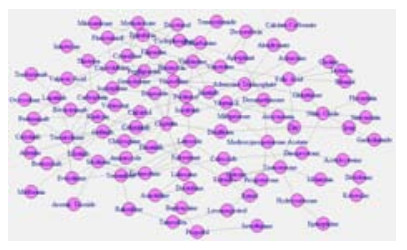
|
图 3 乳腺癌药物网络模型 Figure 3 Breast cancer drug network model. |
图 3的药物关联网络模型与图 2类似,也是只有少部分节点(长春花碱等)的度较大,也属于无标度网络。其中的关键节点有:吉非替尼、注射用顺铂等,这些关键节点在乳腺癌药物研究中属于研究热点,并且可能与其他多种药物之间鹅存在相互作用。
图 3中删除了16种药物(炔雌醇等)孤立节点,余下97种药物。可以发现,酮咯酸和双氯芬酸这2种药物最为特殊,只具有单相关性[35-36],且与其它药物均无关联,且关联度最高。2种药物之间关联度排名为第2至第5名的分别是:替莫唑胺与达卡巴嗪、异丙酚与七氟醚、辛伐他汀与氟伐他汀、地塞米松与米非司酮,对这些药物存在的关联,已有多篇文献进行了报道,如文献[37-40]等。乳腺癌药物研究热点排名前5的是:他莫昔芬、曲妥珠单抗、阿霉素、紫杉醇和注射用雌二醇。图中还可以发现,乳腺癌相关药物之间的关联较多,与其它药物关联最多的是:吉非替尼和顺铂,这2者分别与其它10种药物相关;其次,而与其它8种药物相关的药物为:白细胞生成素、酒石酸长春瑞滨、表阿霉素、长春花碱、依西美坦和卡培他滨这6种药物。有16种药物(七氟醚等)具有单相关性。
2.3 乳腺癌基因药物之间的关联在已得到的乳腺癌基因关联和药物关联的基础上,基于ABC理论来判断乳腺癌相关基因与药物之间是否存在关联或隐含关联,同时计算出两者之间的Relevance Score值,去重后得到639种不同关联。再利用前述关联公式得到乳腺癌基因药物之间关联的Lift值(结果四舍五入取整),设定的Lift阈值大于等于3,所以最终得到基因127种,药物77种,它们之间存在384种不同关联数(表 3),同时以基因与药物之间的Lift值排序建表(表 4)。
| 表 3 部分乳腺癌相关基因药物关联以及其Lift值 Table 3 Part of breast cancer-associated gene-drug correlations and their Lift value |
| 表 4 Lift值排名前15的乳腺癌基因药物 Table 4 Top 15 associations between genes and drugs of the breast cancer selected according to Lift values |
从表 3中可以发现,有些基因只与1种药物具有关联,如:ATM与咖啡因、BMI1与氟尿嘧啶、CA9与紫杉醇等36种基因。同样,有些药物只与1种基因相关,如:利多卡因、酒石酸长春瑞滨、长春花碱、腺苷等10种药物。从表 4可以得出,基因与药物关联度最高的是Atg7与腺嘌呤、CAV1与咖啡因、CAV1与氟伐他汀、PGRMC1与雌激素三醇,这4种关联强度并列第一。同样依据前述网络框架算法,构建乳腺癌基因药物网络模型(图 4)。
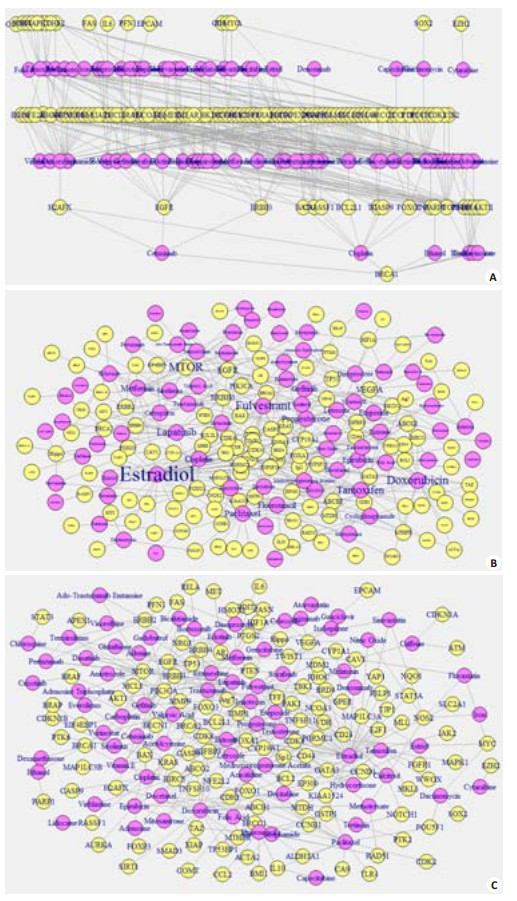
|
图 4 乳腺癌基因药物网络模型 Figure 4 Breast cancer gene-drug network model(purple nodes are drugs, and yellow nodes are genes). A: Tree network diagram; B: Network diagram(based on vertex degree); C: Network diagram(based on vertex type). |
由于乳腺癌基因药物节点关联较多,为了更加清楚地显示可视化效果,本文分别采用树状、基于度和节点的方法构建网络模型(图 4A、B、C)。图 4A中黄色节点为基因,紫色节点为药物,每行只与相邻行存在关联,有助于观察关键节点和特殊节点的关联情况;图 4B中的文字大小体现该节点的度,字体越大,说明该节点在网络结构中的位置越关键,可以很容易得到雌二醇、阿霉素、MTOR等是乳腺癌基因药物网络模型的关键节点,也就可能是研究乳腺癌相关基因药物间关联的重要突破点;再结合图 4C可以发现其模型结构也是属于无标度网络。
图 4中的乳腺癌基因药物之间存在较多关联,单独成对出现的基因与药物只有1对,为:EZH2与阿糖胞苷。图中关联度大于5的基因有15个,关联度排名前5的是:MTOR、EGFR、VEGFA、ERBB3和ABCG2;关联度大于5的药物有24种,关联度排名前5的是:注射用雌二醇、氟维司琼、阿霉素、他莫昔芬和拉帕替尼。乳腺癌基因与药物只具有单相关性的有45个,如:APEX1、ATM、BMI1等;而乳腺癌药物与基因的只具有单相关性的有12种,如:利多卡因、酒石酸长春瑞滨、长春花碱等。
2.4 预测结果本文将乳腺癌的基因-基因、药物-药物和基因-药物之间的所有关联结果一一验证,列表显示关联程度排名靠前但尚未报道的实体对(表 5),基因的中文名称是对照《英汉人类基因词典》[41]得到的。
| 表 5 预测部分关联程度较高但尚未证实的生物实体间新关联 Table 5 Partial prediction of close relationships between biomedical entities that have not yet been confirmed |
表 5中尚未证实的成对关联,可为研究人员提供新的研究思路,例如:基因EZH2(果蝇味增强子同源2)与药物阿糖胞苷在本文的研究结果中显示存在关联,其中基因EZH2是细胞增殖所必需的,在许多肿瘤组织中存在不同程度的高表达,直接参与了乳腺癌演变过程,是肿瘤发生早期阶段分子事件[42],而阿糖胞苷主要作用于细胞S增殖期的嘧啶类抗代谢药物,通过抑制细胞DNA的合成,干扰细胞的增殖,目前主要应用于白血病[43];目前尚无文献对这2者是否存在关联进行报道,但是前者作用于增殖,而后者产生抑制作用,那么这2个生物实体之间可能会存在关联。
2.5 ROC曲线评价本文对乳腺癌的基因-基因、药物-药物和基因-药物之间的所有关联结果进行验证,并在SPSS 20环境下使用ROC曲线判断算法性能(图 5)。可以得到ROC曲线下的面积分别为0.863、0.819和0.763,关联准确度中等偏上,相应的标准误分别为0.068、0.054和0.027,P值均为0.000,95%置信区间分别为(0.730,0.996)、(0.713,0.925)和(0.710,0.816)。本文算法优于CoPub[31]生物实体关联提取算法。
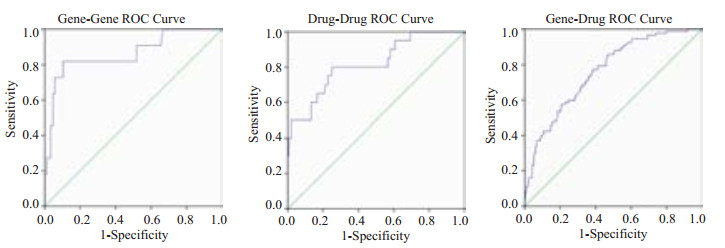
|
图 5 ROC曲线性能评价 Figure 5 Statistical evaluation of the receiver-operating characteristic (ROC) curve. |
通过该方法,验证了本文算法具有较高性能,能够提取生物实体关联。与其他关联提取算法[44-45]类似,本文也得到了一些尚未验证的实体关联,即有一些假阳性的预测性的结果,不过这是允许的[1],因为这也是生物实体关联提取所需要达到的目标之一:提出预测性的研究假设,帮助科研人员设计相关实验方向[46]。
3 讨论本文在近2年乳腺癌相关文献中识别基因、药物实体,提取它们之间的关联,并进行集成整合预测,有助于生物医学研究人员设计实验方向。
在得到的乳腺癌基因药物网络模型中“,节点”代表生物医学实体存储在RDF三元组(即疾病、药物、基因)“,边”表示两生物医学实体之间的关联(例如,关系“谓词”)。为简单起见,在本研究中仅考虑单向关联关系,在原始的RDF图丢弃方向和类型。换句话说,只要两节点之间有关联,即认为这两个节点之间有边缘。假设这样的简化疾病药物基因的关联网络中,网络中的网络模式有下2点作用:(1)基本可以代表疾病基因药物之间的相互关系;(2)反映了一个可以有效实现特定功能的框架。对图 2、图 3和图 4C的网络结构进行分析,得到在乳腺癌基因药物网络的核心中,药物、基因节点的分布服从幂律分布,表明不同类型节点相关的网络属于无标度网络。网络中的部分节点只有少数的关联(数量 < 4),但其它大部分节点均有大量的关联。类似这样的分布,许多关于生物实体网络的研究中也得到同样的结果[3]。本研究分析表明,在一个具有集成性质的异质关联组成的网络中,依然具有无标度的网络结构。
通过对基因-基因、药物-药物和基因-药物这3个不同的网络模型的可视化,可以发现大部分节点都可以通过“第三者节点”连通,从而发现它们之间的潜在关联,定量评估实体关联。通过网络模型所得到的结果,可以把这些基因、药物与乳腺癌表型关联在一起,有助于发现乳腺癌中的候选基因和候选药物,以及基因-基因、药物-药物和基因-药物间的新关联。例如,对基因-基因网络模型分析显示,基因ERBB2是该网络模型核心节点之一,它是细胞膜表面结合的受体酪氨酸激酶,已有多篇文献[47-48]证明它参与了乳腺体发育,并对未成熟的T细胞在胸腺增殖具有负调控等,是可能导致乳腺癌、胃癌、卵巢癌等疾病的致病基因,同时,ERBB2与其它基因的关联研究也取得了一定进展[49-50];另外,本文得到基因ERBB2与PIK3CA可能存在关联,而PIK3CA基因突变是乳腺癌肿瘤中最常见的突变之一,在肿瘤形成过程中有着重要作用[51],它的激活可引起乳腺癌患者对靶向药物曲妥珠单抗的耐药[52],两者之间的关联尚未见报道,不过ERBB2参与乳腺腺体发育,促进细胞增殖;PIK3CA参与信号转导,促进蛋白结合,而且这2种基因均与乳腺癌肿瘤的早期形成有着密切关系,可推测这两者可能存在关联。
对比其他同类算法,本文算法优点在于:(1)不仅使用经典的ABC理论,还采用了关联规则进行综合评估,而其他算法大多只使用ABC理论;(2)提取了基因-基因、药物-药物和基因-药物这3种不同的生物实体关联,而PubGene[53]仅提取基因-基因间的关联,Sun等[44]提取的是药物-药物间的关联,CoPub[31]提取的是基因-疾病、药物-疾病和药物-生物过程的关联;(3)本文算法使用ROC曲线验证得到曲线下面积分别为0.863、0.819和0.763,而Frijters等[30]以R-scaled值为阈值对CoPub所得结果进行ROC曲线验证,曲线下面积最高约为0.7(R-sacled值大于30),PubGene仅有60%的精确率,因此本文算法精确度更高。本文已成功将算法应用于乳腺癌相关基因、药物关联的研究中,下一步工作就是需要在更大规模数据中评估本算法的性能,确保进一步推广使用。
| [1] |
Fleuren WW, Alkema W. Application of text mining in the biomedical domain[J].
Methods,2015, 74 : 97-106.
DOI: 10.1016/j.ymeth.2015.01.015. ( 0) 0)
|
| [2] |
Ananiadou S, Mcnaught J.
Text mining for biology and biomedicine[M]. Artech House Inc, 2006 : 3 -4.
( 0) 0)
|
| [3] |
Zhang Y, Tao C, Jiang G, et al. Network-based analysis reveals distinct association patterns in a semantic MEDLINE-based drugdisease-gene network[J].
J Biomed Semantics,2014, 5 : 33.
DOI: 10.1186/2041-1480-5-33. ( 0) 0)
|
| [4] |
Swanson DR. Medical literature as a potential source of new knowledge[J].
Bull Med Libr Assoc,1990, 78 (1) : 29-37.
( 0) 0)
|
| [5] |
Swanson DR. Fish oil, Raynaud's syndrome, and undiscovered public knowledge[J].
Perspect Biol Med,1986, 30 (1) : 7-18.
DOI: 10.1353/pbm.1986.0087. ( 0) 0)
|
| [6] |
Swanson DR. Migraine and Magnesium: eleven neglected connections[J].
Perspect Biol Med,1988, 31 (4) : 526-57.
DOI: 10.1353/pbm.1988.0009. ( 0) 0)
|
| [7] |
Miwa M, Saetre R, Miyao Y, et al. Protein-protein interaction extraction by leveraging multiple kernels and parsers[J].
Int J Med Inform,2009, 78 (12) : e39-46.
DOI: 10.1016/j.ijmedinf.2009.04.010. ( 0) 0)
|
| [8] |
Chatr-Aryamontri A, Winter A, Perfetto L, et al. Benchmarking of the 2010 BioCreative challenge III text-mining competition by the BioGRID and MINT interaction databases[J].
BMC Bioinformatics,2011, 12 (Suppl 8) : S8.
DOI: 10.1186/1471-2105-12-S8-S8. ( 0) 0)
|
| [9] |
Fundel K, Küffner R, Zimmer R. RelEx--relation extraction using dependency parse trees[J].
Bioinformatics,2007, 23 (3) : 365-71.
DOI: 10.1093/bioinformatics/btl616. ( 0) 0)
|
| [10] |
Bui QC, Sloot PM, Van Mulligen EM, et al. A novel feature-based approach to extract drug-drug interactions from biomedical text[J].
Bioinformatics,2014, 30 (23) : 3365-71.
DOI: 10.1093/bioinformatics/btu557. ( 0) 0)
|
| [11] |
Xu R, Wang Q. Large-scale extraction of accurate drug-disease treatment pairs from biomedical literature for drug repurposing[J].
BMC Bioinformatics,2013, 14 (13) : 181.
( 0) 0)
|
| [12] |
Piro RM, Di Cunto F. Computational approaches to disease-gene prediction: rationale, classification and successes[J].
FEBS J,2012, 279 (5) : 678-96.
DOI: 10.1111/j.1742-4658.2012.08471.x. ( 0) 0)
|
| [13] |
Chen J, Aronow BJ, Jegga AG. Disease candidate gene identification and prioritization using protein interaction networks[J].
BMC Bioinformatics,2009, 10 (1) : 73.
DOI: 10.1186/1471-2105-10-73. ( 0) 0)
|
| [14] |
Goh KI, Cusick ME, Valle D, et al. The human disease network[J].
Proc Nat Aca Sci,2007, 104 (21) : 8685-90.
DOI: 10.1073/pnas.0701361104. ( 0) 0)
|
| [15] |
Suthram S, Dudley JT, Chiang AP, et al. Network-Based elucidation of human disease similarities reveals common functional modules enriched for pluripotent drug targets[J].
PLoS Comput Biol,2010, 6 (2) : 1000662.
DOI: 10.1371/journal.pcbi.1000662. ( 0) 0)
|
| [16] |
Arrell DK, Terzic A. Network systems biology for drug discovery[J].
Clin Pharm Therap,2010, 88 (1) : 120-5.
DOI: 10.1038/clpt.2010.91. ( 0) 0)
|
| [17] |
Dudley JT, Deshpande T, Butte AJ. Exploiting drugdisease relationships for computational drug repositioning[J].
Brief Bioinform,2011, 12 (4) : 303-11.
DOI: 10.1093/bib/bbr013. ( 0) 0)
|
| [18] |
Hu GH, Agarwal P. Human Disease-Drug network based on genomic expression profiles[J].
PLoS One,2009, 4 (8) : e6536.
DOI: 10.1371/journal.pone.0006536. ( 0) 0)
|
| [19] |
Bauer MA, Bundschus M, Rautschka M, et al. Gene-diseasenetwork analysis reveals functional modules in mendelian, complex andenvironmental diseases[J].
PLoS One,2011, 6 (6) : e20284.
DOI: 10.1371/journal.pone.0020284. ( 0) 0)
|
| [20] |
Daminelli S, Haupt VJ, Reimann M, et al. Drug repositioning through incomplete bi-cliques in anintegrated drug-target-disease network[J].
Integr Biol (Camb),2012, 4 (7) : 778-88.
DOI: 10.1039/c2ib00154c. ( 0) 0)
|
| [21] |
Milo R, Itzkovitz S, Kashtan N, et al. Superfamilies of evolved and designed networks[J].
Science,2004, 303 (5663) : 1538-42.
DOI: 10.1126/science.1089167. ( 0) 0)
|
| [22] |
Zhang Y, Xuan J, Bg DR, et al. Reconstruction of gene regulatory modules in cancer cell cycle by multi-source data integration[J].
PLoS One,2010, 5 (4) : e10268.
DOI: 10.1371/journal.pone.0010268. ( 0) 0)
|
| [23] |
Zhang Y, Xuan J, Reyes BL, et al. Reverse engineering module networks by PSO-RNN hybrid modeling[J].
BMC Genomics,2009, 10 (1) : S15.
DOI: 10.1186/1471-2164-10-15. ( 0) 0)
|
| [24] |
周琳, 孔雷, 赵方庆. 生物大数据可视化的现状及挑战[J].
科学通报,2015 (Z1) : 547-57.
( 0) 0)
|
| [25] |
Maglott D, Ostell J, Pruitt KD, et al. Entrez gene: gene-centered · 178 ·http://www[J].
Nucleic Acids Res,2005, 33 (Database issue) : D54-8.
( 0) 0)
|
| [26] |
Pruitt KD, Tatusova T, Maglott DR. NCBI reference sequences (RefSeq): a curated non-redundant sequence database of genomes, transcripts and proteins[J].
Nucleic Acids Res,2007, 35 (Database issue) : D61-5.
( 0) 0)
|
| [27] |
Ashburner M, Ball CA, Blake JA, et al. Gene ontology: tool for the unification of biology[J].
Nat Genet,2000, 25 (1) : 25-9.
DOI: 10.1038/75556. ( 0) 0)
|
| [28] |
Hamosh A, Scott AF, Amberger J, et al. Online mendelian inheritance in man (OMIM), a knowledgebase of human genes and genetic disorders[J].
Nucleic Acids Res,2002, 30 (1) : 52-5.
DOI: 10.1093/nar/30.1.52. ( 0) 0)
|
| [29] |
Knox C, Law V, Jewison T, et al. DrugBank 3.0: a comprehensive resource for 'omics' research on drugs[J].
Nucleic Acids Res,2011, 39 (Database issue) : D1035-41.
( 0) 0)
|
| [30] |
Frijters R, Van Vugt M, Smeets R, et al. Literature mining for the discovery of hidden connections between drugs, genes and diseases[J].
PLoS Comput Biol,2010, 6 (9) : 655-64.
( 0) 0)
|
| [31] |
Chen MS, Han JW, Yu PS. Data mining: An overview from a database perspective[J].
IEEE Trans Knowl Data Eng,1996, 8 (6) : 866-83.
DOI: 10.1109/69.553155. ( 0) 0)
|
| [32] |
Brin S, Motwani R, Silverstein C. Beyond market baskets: generalizing association rules to correlations[J].
Proc Acm Sigmod,1997, 26 (1) : 265-76.
( 0) 0)
|
| [33] |
Ihaka R, Gentleman RR. A language for data analysis and graphics[J].
J Compu Graph Stat,1996, 5 (3) : 299-314.
( 0) 0)
|
| [34] |
Hanley JA, Mcneil BJ. The meaning and use of the area under a receiver operating characteristic(ROC) curve[J].
Radiology,1982, 143 (1) : 29-36.
DOI: 10.1148/radiology.143.1.7063747. ( 0) 0)
|
| [35] |
Lee TH, Choi W, Ji YS, et al. Comparison of ketorolac 0.45% versus diclofenac 0.1% for macular thickness and volume after uncomplicated cataract surgery[J].
Acta Ophthalmol (Copenh),2015 : 1-6.
( 0) 0)
|
| [36] |
Forget P, Machiels JP, Coulie PG, et al. Neutrophil: lymphocyte ratio and intraoperative use of ketorolac or diclofenac are prognostic factors in different cohorts of patients undergoing breast, lung, and kidney cancer surgery[J].
Ann Surg Oncol,2013, 20 (Suppl 3) : S650-60.
( 0) 0)
|
| [37] |
Ribas A, Puzanov I, Dummer R, et al. Pembrolizumab versus investigator-choice chemotherapy for ipilimumab-refractory melanoma (KEYNOTE-002): a randomised, controlled, phase 2 trial[J].
Lancet Oncol,2015, 16 (8) : 908-18.
DOI: 10.1016/S1470-2045(15)00083-2. ( 0) 0)
|
| [38] |
Siampalioti A, Karavias D, Zotou A, et al. Anesthesia management for the super obese: is sevoflurane superior to propofol as a sole anesthetic agent? A double-blind randomized controlled trial[J].
Eur Rev Med Pharmacol Sci,2015, 19 (13) : 2493-500.
( 0) 0)
|
| [39] |
Hirota T, Ieiri I. Drug-drug interactions that interfere with statin metabolism[J].
Expert Opin Drug Metab Toxicol,2015, 11 (9) : 1435-47.
DOI: 10.1517/17425255.2015.1056149. ( 0) 0)
|
| [40] |
Xing K, Gu B, Zhang P, et al. Dexamethasone enhances programmed cell death 1 (PD-1) expression during T cell activation: an insight into the optimum application of glucocorticoids in anti-cancer therapy[J].
BMC Immunol,2015, 16 : 39.
DOI: 10.1186/s12865-015-0103-2. ( 0) 0)
|
| [41] |
张闻.
英汉人类基因词典[M]. 北京: 人民卫生出版社, 2011 .
( 0) 0)
|
| [42] |
Wang H, Wang M, Lian G. Expression of enhancer of zeste homolog 2 in esophageal squamous cell carcinoma and its prognostic value in postoperative patients[J].
J South Med Univ,2013, 33 (1) : 99-102.
( 0) 0)
|
| [43] |
Wells RJ, Adams MT, Alonzo TA, et al. Mitoxantrone and cytarabine induction, high-dose cytarabine, and etoposide intensification for pediatric patients with relapsed or refractory acute myeloid leukemia: Children's Cancer Group study 2951[J].
J Clin Oncol,2003, 21 (15) : 2940-7.
DOI: 10.1200/JCO.2003.06.128. ( 0) 0)
|
| [44] |
Kim S, Liu H, Yeganova L, et al. Extracting drug-drug interactions from literature using a rich feature-based linear kernel approach[J].
J Biomed Inform,2015, 55 : 23-30.
DOI: 10.1016/j.jbi.2015.03.002. ( 0) 0)
|
| [45] |
Xu R, Wang Q. Large-scale automatic extraction of side effects associated with targeted anticancer drugs from full-text oncological articles[J].
J Biomed Inform,2015, 55 : 64-72.
DOI: 10.1016/j.jbi.2015.03.009. ( 0) 0)
|
| [46] |
Gonzalez GH, Tahsin T, Goodale BC, et al. Recent advances and emerging applications in text and data mining for biomedical discovery[J].
Brief Bioinform,2015, 9 : 1-10.
( 0) 0)
|
| [47] |
Rayavarapu RR, Heiden B, Pagani N, et al. The role of multicellular aggregation in the survival of ErbB2-positive breast cancer cells during extracellular matrix detachment[J].
J Biol Chem,2015, 290 (14) : 8722-33.
DOI: 10.1074/jbc.M114.612754. ( 0) 0)
|
| [48] |
Tafe LJ, Steinmetz HB, Allen SF, et al. Rapid fluorescence in situ hybridisation (FISH) for HER2 (ERBB2) assessment in breast and gastro-oesophageal cancer[J].
J Clin Pathol,2015, 68 (4) : 306-8.
DOI: 10.1136/jclinpath-2014-202787. ( 0) 0)
|
| [49] |
Li ZD, Wang K, Yang XW, et al. Expression of aryl hydrocarbon receptor in relation to p53 status and clinicopathological parameters in breast cancer[J].
Int J Clin Exp Pathol,2014, 7 (11) : 7931-7.
( 0) 0)
|
| [50] |
Wilson TR, Xiao Y, Spoerke JM, et al. Development of a robust RNA-based classifier to accurately determine ER, PR, and HER2 status in breast cancer clinical samples[J].
Breast Cancer Res Treat,2014, 148 (2) : 315-25.
DOI: 10.1007/s10549-014-3163-8. ( 0) 0)
|
| [51] |
Miron A, Varadi M, Carrasco D, et al. PIK3CA mutations in in situ and invasive breast carcinomas[J].
Cancer Res,2010, 70 (14) : 5674-8.
DOI: 10.1158/0008-5472.CAN-08-2660. ( 0) 0)
|
| [52] |
Wang L, Zhang Q, Zhang J, et al. PI3K pathway activation results in low efficacy of both trastuzumab and lapatinib[J].
BMC Cancer,2011, 11 (1) : 248.
DOI: 10.1186/1471-2407-11-248. ( 0) 0)
|
| [53] |
Jenssen TK, Laegreid A, Komorowski J, et al. A literature network of human genes for high-throughput analysis of gene expression[J].
Nat Genet,2001, 28 (1) : 21-8.
( 0) 0)
|
 2016, Vol. 36
2016, Vol. 36
