2. 上海交通大学 生物医学工程学院 Bio-ID研究中心,上海 200240 ;
3. 西北大学生命科学学院,陕西 西安 710069
2. School of Biomedicine Engineering and Bio-ID Research Center, Shanghai Jiaotong University, Shanghai 200240, China ;
3. School of Life Science, Northwestern University, Xi'an 710069, China
选择性多聚腺苷化(APA)是一类很重要的基因转录后表达调控方式,通过调节转录本3’非翻译区(3’ untranslated region, UTR)的长度来影响包括mRNA的稳定性、亚细胞定位和翻译效率等在内的多个生物学过程[1-3]。APA在生物体生长发育和疾病发生发展过程中受到高度有序的调控[4-5]。在发育生物学领域,全基因组范围内的APA谱表现出细胞类型特异性:在多能性的胚胎干细胞中,基因倾向于选取近端的多聚腺苷酸位点(PAS)而产生更短的3’UTR来维持干细胞特性;而终端分化细胞更多取用远端PAS[6-7];由APA调控的3’UTR的长短也呈现发育阶段性:小鼠胚胎发育伴随着全局3’ UTR的逐步增长[4];果蝇胚胎发育过程中的3’UTR变化趋势与之相似[8];而斑马鱼的胚胎发育中mRNA的3’ UTR呈现“V”字形的动态变化,在合子形成后基因组被活化表达的短时间内3’UTR缩短,之后迅速变长,且伴随着3’UTR多样性的由丰到简[9]。
小鼠雌性生殖干细胞(mFGSCs)是一类单能性成体干细胞,能在保持自我更新同时定向分化为卵细胞[10-12],是生殖医学和干细胞研究领域的重要研究对象。虽然从小鼠体内成功分离并建立细胞系[10],但FGSCs的分子生物学研究目前仍处于起步阶段,其分子水平上的调控机理至今尚不清晰。APA所产生的不同3’UTR作为一种重要的表观遗传调控手段在生殖干细胞中所发挥的作用也未被深入研究。本文应用本实验室建立的基于高通量测序的3’端poly(A)位点捕获技术3T-seq[13],在全基因组范围内分别对小鼠雌性生殖干细胞和胚胎干细胞(ESCs)APA谱图进行了分析比较,以探讨该转录后修饰对FGSCs特性的影响,为进一步丰富和完善生殖干细胞分子生物学研究提供新的内容。
1 材料和方法 1.1 细胞培养小鼠雌性生殖干细胞(由上海交通大学吴际教授惠赠),小鼠胚胎干细胞J1均培养于用0.1%明胶包被的6孔板培养皿(Corning),FGSC培养基为MEM-α(Gibco),含有终浓度为10%胎牛血清(Gibco),1 mmol/L丙酮酸钠,1 mmol/L非必需氨基酸(Gibco),1 mmol/L L-谷氨酰胺,0.1 mmol/L β-巯基乙醇,20 ng/mL LIF(Peprotech),10 ng/mL的EGF(Peprotech),10 ng/mL GDNF(Peprotech),10 ng/mL bFGF(Peprotech);ESC培养基为:高糖型DMEM(Gibco)、15%胎牛血清、100 U/mL青链霉素(Gibco)、1000 U/mL LIF(Chemicon)、0.1 mmol/L β-巯基乙醇、1%非必需氨基酸;新鲜配制的培养基用0.22 μm的滤器(Millipore)过滤后4 ℃保存。细胞培养置于5% CO2,37 ℃培养箱培养,每2 d更换培养基。细胞生长至80%~90%密度时,以1:2~3的比例传代,经3~4代收集细胞用于提取总RNA。
1.2 细胞总RNA的提取将生长状况良好的细胞弃去培养基,用PBS洗1次,每个6孔板加入1 mL Trizol regen(t Invitrogen)收集细胞,反复吹打使贴壁细胞破碎溶于Trizol后转移至新的RNase-free的1.5 mL离心管中,用于立即抽提总RNA或者冻于-80 ℃保存。总RNA抽提参照试剂盒说明书,RNA沉淀溶于RNase Free水(Invitrogen),用Nanodrop 2000(Thermo)定量并在1.5%琼脂糖凝胶中检验RNA完整度。
1.3 3T-seq文库构建文库构建参照本实验室之前报道的方法[13],简单来说:50 μg总RNA与预先包被了带生物素(Biotin)修饰的反转录引物GsuI-Oligo(dT)20的链霉亲和素磁珠M-280(Invitrogen)孵育,在Super Script Ⅲ(Invitrogen)反转录酶的作用下,用含有5-methylated-dCTP的dNTP Mix合成第1链cDNA,5m-dCTP封闭新合成的一链cDNA内部GsuⅠ的识别和切割位点;经过二链合成后在磁珠上将双链cDNA用片段化酶(NEB)随机打断为200~400 bp,只留下与磁珠相连的近3’端的DNA标签片段,用Gsu(I Fermentas)来酶切释放。纯化后将3’UTR标签文库末端补平和3’端加A,连接Illumina公司的P5/ P7测序接头,高保真酶PCR放大,筛选200~500 bp大小DNA作为测序文库经Illumina Hiseq 2500进行高通量测序,测序数据已上传至EMBL-EBI ArrayExpres数据库(accession number: E-MTAB-4038)。
1.4 数据分析数据处理方法参照之前的报道[14],将原始数据经过C++脚本的过滤和筛选得到的可用有效的数据,应用bowtie2软件比对到UCSC的小鼠参考基因组mm10,APA位点由迭代聚类方法鉴定。两种细胞系之间的3’UTR的变化用线性趋势分析方法进行比较。FDR由R软件估计,基因功能分析Gene Ontology由DAVID(https://david.ncifcrf.gov)完成。
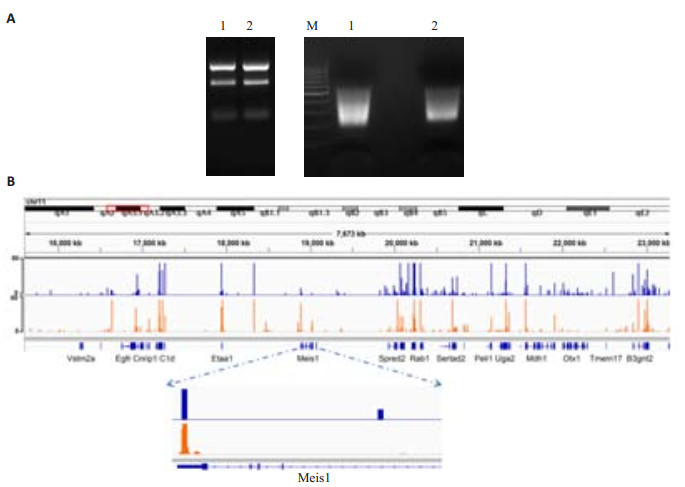
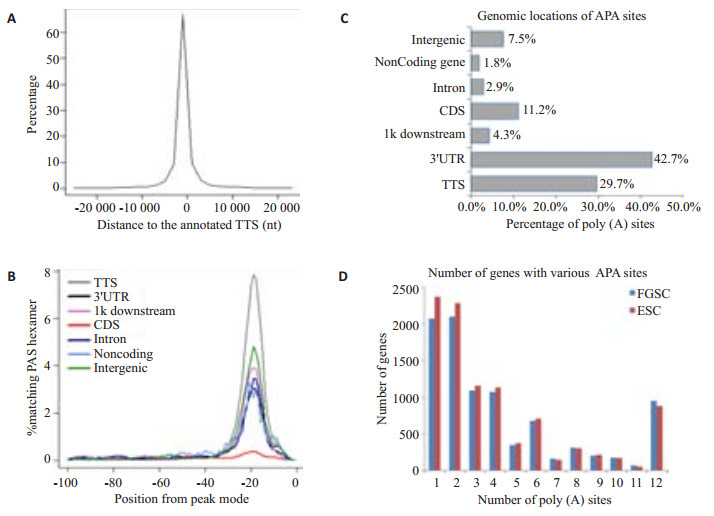
2 结果 2.1 FGSCs和ESCs细胞内基于3T-seq的全基因组范围APA位点的定位高质量的总RNA提取之后按照我们最近报道的方法[13]构建3T-seq文库(图 1A)并进行高通量测序。对测序数据进行生物信息学处理,两个样品共得到约9.2 M reads作为clean data,其中7.7 M(约84%)reads能够比对到基因组上,包含3.88 M特异性reads(占50%)。基因Poly(A)位点下游20 nt序列若出现连续8个A或含有12个或以上的A,则作为Internal Priming被滤掉[15]以减少对真实APA位点鉴定的影响,最终过滤得到3.1 M有效reads,这些reads有接近89.7%(曲线下方的面积)集中在已注释的转录终止位点(Transcription Terminal site, TTS)附近,如图 2A所示。APA位点鉴定方法根据之前报道[14],最后共确定50 243个APA位点:FGSCs细胞系25 431个,ESCs细胞系24 812个。在全基因组范围内位点如图 1B所示,具体统计数据见表 1。在APA位点上游20~30 nt范围内搜索以AAUAAA为序列的PAS六聚体,如图 2B所示,除了CDS区的APA位点外均能找到该motif,表明定义APA位点的高效性。APA位点在基因组中的具体分布如图 2C,42.7%位于已知3’UTR区,29.7%落在转录终止位点(TTS),剩余的分别比对到TTS下游1000 nt:4.3%,编码区(CDS):11.2%,内含子区(Intron):2.9%,非编码区(Noncoding gene):1.8%和基因间隔(Intergenic):7.5%。
|
图 1 FGSCs和ESCs中APA位点的研究 Figure 1 Identification of APA sites in FGSCs and ESCs. A: Gel electrophoresis of the total RNA and 3T-seq library for NGS. Lane 1: FGSCs; Lane 2: ESCs. M: 100 bp DNA Ladder; B: A genomic view of APA sites defined by 3T-seq in IGV genome browser. Blue track: ESCs; Yellow track: FGSCs. |
| 表 1 高通量测序数据处理和比对结果汇总 Table 1 Summary statistics of sequencing data from Illumina Hiseq2500 |
|
图 2 APA位点的鉴定和比对分析 Figure 2 Characterization and comparative analysis of APA sites in FGSCs and ESCs. A: Distribution of 3T-seq reads across the gene body; B: Position-specific distributions of PAS signal hexamer for poly(A) sites; C: Genomic locations of poly(A) sites; D: Statistics of genes with various number of detected poly(A) sites. |
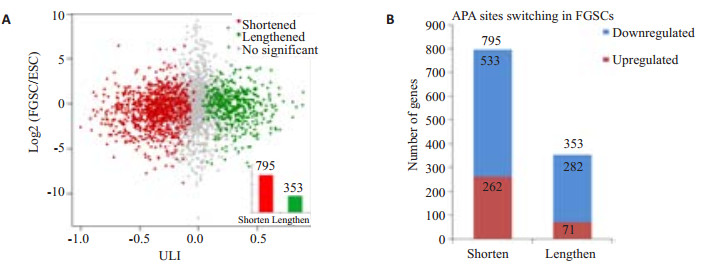
为了方便进行比较,我们采用线性趋势分析(Test of linear trend)方法[14]对含有多个APA位点的基因均一化处理后引入3’UTR长度指数(3’UTR length index,ULI)来定量指征同一个基因3’UTR的长度变化,若ULI为正值,则表示FGSC细胞系的3’UTR相对于ESC的变长,反之变短。结果有1 148个基因3’UTR长度发生明显变化,其中ULI为负795个(66%),ULI为正353个(34%)。整体的变化趋势如图 3A所示。拥有不同APA位点的基因个数代表着细胞APA的多样性,图 2D展示了这一分布情况。在FGSCs和ESCs中拥有1或2个APA位点的基因占最大比例,总和占总体45.2%,其次是3个和4个,总和为23.4%,拥有多至12个位点的基因也占10%,整体上看,两者在APA丰度上的差异并不显著。
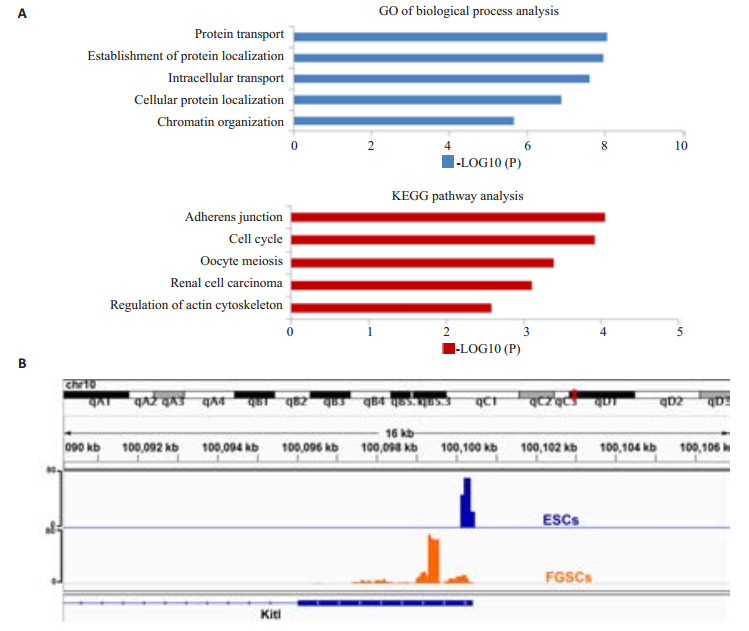
2.3 3’UTR变化基因的功能分析如上文所述,共有795个基因(图 3)在FGSCs中呈现出相比于ESCs的3’UTR变短的趋势,应用DAVID(https://david.ncifcrf.gov)的Gene Ontology和KEGG对基因进行功能和信号通路分析,这些基因多富集在蛋白质转运,定位,胞内运输等生物学过程,伴随着一些胚胎发育后期出现的与中胚层发育和原肠胚形成的基因富集条目;在细胞内的定位主要位于胞内腔(Intracellular lumen),细胞器内腔,核内腔和核质中,以及非膜结合细胞器;从分子功能上主要作用在与核苷酸结合,主要是嘌呤中的腺嘌呤核苷酸以及一部分与核糖核苷酸和ATP的结合。在通路分析方面,这些基因集中于粘着链接,细胞周期以及与卵细胞减数分裂相关的信号通路。GO分析条目如图 4A所示。
|
图 3 FGSC和ESC中3’UTR长度变化趋势 Figure 3 APA sites variations in FGSCs compared to ESCs. A: Scatter diagram of genes with differential APA defined by ULI; B: Composition of down-regulated and upregulated genes in FGSCs. |
|
图 4 APA差异位点基因功能分析 Figure 4 Gene function analysis of differential 3'UTR genes. A: GO and KEGG pathway analysis using DAVID; B: 3'UTR shortening in kitl gene shown in IGV genome browse. |
3’UTR变短的基因,因为其较短的3’UTR增加了mRNA本身的稳定性,减少了能与转录后调控的反式作用因子(如RNA结合蛋白)或者microRNA相互结合的顺式作用元件,更有可能逃逸细胞中microRNA介导的基因沉默,导致更高的表达水平[3],对于在FGSCs中的3’UTR变短伴随着表达量上升的262个基因,如图 3B中所示,在蛋白质转运和定位等生物学过程上有着富集,深入分析发现在FGSCs中,与生殖细胞发育密切相关的基因kitl发生显著的3’UTR缩短。
Kitl基因编码的KITL是一个干细胞因子(也被称为SCF),作为配体与跨膜酪氨酸激酶受体KIT结合,两者形成kit/kitl信号系统共同调控着出生前后卵巢中卵细胞和卵泡的发育,也有很多报道在精子发生中,kitl会促进A型精原细胞的DNA合成从而促进细胞增殖[16-17]。它在FGSCs中存在着明显的3’UTR缩短现象,在距离TTS位点更近的地方存在APA位点,如图 4B所示,对近端APA位点的取用伴随着该基因表达量的升高,表明kitl基因作为一个参与生殖细胞发育的基因在FGSCs中有特异的表达模式。
3 讨论生殖细胞的发育是一个复杂而高度有序的过程,受基因调控网络精确和稳定的调控[18]。雌性生殖干细胞作为一种单能性的成体干细胞,处于生殖发育进程中一个很特殊的环节:它们处于原始生殖细胞(PGC)发育下游,负责分化成成熟的生殖细胞。近年来越来越多的研究显示APA在发育过程中发挥重要的作用,比如多能性的胚胎干细胞更倾向于采用更短的3’UTR,使细胞能维持自身的干细胞特性,保持自我更新和较快的增殖[4, 7]。而对于生殖干细胞的转录后调控的研究仍处于空白,在此我们利用基于高通量测序的组学研究方法获得了小鼠FGSCs在全基因组范围内的APA谱,使我们能对生殖干细胞中的APA取用模式有了更全面的认识,我们的工作填补了生殖细胞领域转录后调控机制研究的空白,为今后对某些特定基因APA的深入研究提供了重要参考。
通过与小鼠ESCs中APA比较分析,发现FGSCs有着与ESCs显著区别的APA谱,具有生殖干细胞的特异性:在我们的数据中发现有1 148个基因相对于ESCs发生了3’UTR的变化,其中34%的基因存在3’UTR变长现象,66%的基因3’UTR缩短,如图 3A所示。对于3’UTR缩短的基因,在基因功能上多富集在与蛋白质转运和定位等生物学功能上,这与APA所介导的与蛋白质细胞定位相关功能相一致,在一些胚胎发育后期出现的与中胚层发育相关基因条目也有富集(图 4A),表明这些在胚胎发育阶段发挥作用的基因可能在生殖干细胞里也发挥一定的作用。
变短的3’UTR能导致更高的转录活性,其转录激活是通过顺式作用元件的缺失,逃逸microRNA介导的负调控来实现[3]。针对雌性生殖干细胞中3’UTR缩短的基因我们展开进一步分析,发现其中包含一些与生殖发育密切相关的基因,比如kitl基因(图 4B),该基因编码蛋白作为III型跨膜酪氨酸激酶受体KIT蛋白的配体[16],参与生殖发育调控中多个重要的信号通路[16-17, 19]。在FGSCs中,该基因产生了APA介导的3’UTR缩短转录子,而且表达量较高,推测缩短的3’UTR导致该基因的转录激活而参与生殖发育相关的生物学活动。
尽管3’UTR缩短导致转录激活,我们在FGSCs中也发现3’UTR变短的基因并不总是伴随着高表达量:795个3’UTR缩短的基因中,其表达量较ESCs上升的只有262个,剩余的533个基因没有明显上调甚至表现出表达下调的趋势,如图 3B所示,这提示3’UTR的长短与表达量不仅仅是简单的负相关。除了我们的结果外,之前的一些报道也发现类似的现象[14, 20-21],我们推测对于那些3’UTR缩短但是表达量没有升高的基因来说,可能是细胞内的microRNA没有表达,从而不能有效的通过与3’UTR靶区域结合抑制靶基因的表达所致。
综上所述,本文首次详细描绘了小鼠FGSCs在全基因组范围内的APA谱,通过和小鼠胚胎干细胞进行系统性的比较,为从转录后调控水平更深入地了解生殖干细胞的基因调控机制提供了基础,从而进一步丰富了生殖干细胞生物学研究内容。
| [1] |
Vinciguerra P, Stutz F. mRNA export: an assembly line from genes to nuclear pores[J].
Curr Opin Cell Biol,2004, 16 (3) : 285-92.
DOI: 10.1016/j.ceb.2004.03.013. ( 0) 0)
|
| [2] |
Berkovits BD, Mayr C. Alternative 3' UTRs act as scaffolds to regulate membrane protein localization[J].
Nature,2015, 522 (7556) : 363-7.
DOI: 10.1038/nature14321. ( 0) 0)
|
| [3] |
Sandberg R, Neilson JR, Sarma A, et al. Proliferating cells Express mRNAs with shortened 3' untranslated regions and fewer microRNA target sites[J].
Science,2008, 320 (5883) : 1643-7.
DOI: 10.1126/science.1155390. ( 0) 0)
|
| [4] |
Ji Z, Lee JY, Pan Z, et al. Progressive lengthening of 3' untranslated regions of mRNAs by alternative polyadenylation during mouse embryonic development[J].
Proc Natl Acad Sci USA,2009, 106 (17) : 7028-33.
DOI: 10.1073/pnas.0900028106. ( 0) 0)
|
| [5] |
Mayr C, Bartel DP. Widespread shortening of 3'UTRs by alternative cleavage and polyadenylation activates oncogenes in cancer cells[J].
Cell,2009, 138 (4) : 673-84.
( 0) 0)
|
| [6] |
Lackford B, Yao C, Charles GM, et al. Fip1 regulates mRNA alternative polyadenylation to promote stem cell self-renewal[J].
EMBO J,2014, 33 (8) : 878-89.
DOI: 10.1002/embj.201386537. ( 0) 0)
|
| [7] |
Ji Z, Tian B. Reprogramming of 3' untranslated regions of mRNAs by alternative polyadenylation in Generation of pluripotent stem cells from different cell types[J].
PLoS One,2009, 4 (12) : e8419.
DOI: 10.1371/journal.pone.0008419. ( 0) 0)
|
| [8] |
Hilgers V, Perry MW, Hendrix D, et al. Neural-specific elongation of 3' UTRs during Drosophila development[J].
Proc Natl Acad Sci USA,2011, 108 (38) : 15864-9.
DOI: 10.1073/pnas.1112672108. ( 0) 0)
|
| [9] |
Li Y, Sun Y, Fu Y, et al. Dynamic landscape of tandem 3' UTRs during zebrafish development[J].
Genome Res,2012, 22 (10) : 1899-906.
DOI: 10.1101/gr.128488.111. ( 0) 0)
|
| [10] |
Zou K, Yuan Z, Yang Z, et al. Production of offspring from a germline stem cell line derived from neonatal ovaries[J].
Nat Cell Biol,2009, 11 (5) : 631-6.
DOI: 10.1038/ncb1869. ( 0) 0)
|
| [11] |
Johnson J, Canning J, Kaneko T, et al. Germline stem cells and follicular renewal in the postnatal mammalian ovary[J].
Nature,2004, 428 (6979) : 145-50.
DOI: 10.1038/nature02316. ( 0) 0)
|
| [12] |
Johnson J, Bagley J, Skaznik-Wikiel M, et al. Oocyte Generation in adult mammalian ovaries by putative germ cells in bone marrow and peripheral blood[J].
Cell,2005, 122 (2) : 303-15.
DOI: 10.1016/j.cell.2005.06.031. ( 0) 0)
|
| [13] |
Lai DP, Tan S, Kang YN, et al. Genome-wide profiling of polyadenylation sites reveals a Link between selective polyadenylation and cancer metastasis[J].
Hum Mol Genet,2015, 24 (12) : 3410-7.
DOI: 10.1093/hmg/ddv089. ( 0) 0)
|
| [14] |
Fu Y, Sun Y, Li Y, et al. Differential genome-wide profiling of tandem 3' UTRs among human breast cancer and normal cells by high-throughput sequencing[J].
Genome Res,2011, 21 (5) : 741-7.
DOI: 10.1101/gr.115295.110. ( 0) 0)
|
| [15] |
Beaudoing E, Freier S, Wyatt JR, et al. Patterns of variant polyadenylation signal usage in human genes[J].
Genome Res,2000, 10 (7) : 1001-10.
DOI: 10.1101/gr.10.7.1001. ( 0) 0)
|
| [16] |
Driancourt MA, Reynaud K, Cortvrindt R, et al. Roles of KIT and KIT LIGAND in ovarian function[J].
Rev Reprod,2000, 5 (3) : 143-52.
DOI: 10.1530/ror.0.0050143. ( 0) 0)
|
| [17] |
Hutt KJ, Mclaughlin EA, Holland MK. Kit ligand and c-Kit have diverse roles during mammalian oogenesis and folliculogenesis[J].
Mol Hum Reprod,2006, 12 (2) : 61-9.
DOI: 10.1093/molehr/gal010. ( 0) 0)
|
| [18] |
Lesch BJ, Page DC. Genetics of germ cell development[J].
Nat Rev Genet,2012, 13 (11) : 781-94.
DOI: 10.1038/nrg3294. ( 0) 0)
|
| [19] |
Mithraprabhu S, Loveland KL. Control of KIT signalling in male germ cells: what can we learn from other systems[J].
Reproduction,2009, 138 (5) : 743-57.
DOI: 10.1530/REP-08-0537. ( 0) 0)
|
| [20] |
Wu CI, Shen Y, Tang T. Evolution under canalization and the dual roles of microRNAs: a hypothesis[J].
Genome Res,2009, 19 (5) : 734-43.
DOI: 10.1101/gr.084640.108. ( 0) 0)
|
| [21] |
Morris AR, Bos A, Diosdado B, et al. Alternative cleavage and polyadenylation during colorectal cancer development[J].
Clin Cancer Res,2012, 18 (19) : 5256-66.
DOI: 10.1158/1078-0432.CCR-12-0543. ( 0) 0)
|
 2016, Vol. 36
2016, Vol. 36
