2. 生殖医学科,广东 广州 510080
2. Reproductive Medicine Center, Guangdong General Hospital, Guangdong Academy of Medical Science, Guangzhou, Guangzhou 510080, China
限制性标记基因组扫描(restriction landmarkgenomic scanning,RLGS)是一种有效的基因扫描技术,能鉴别DNA的放大效应和异常的DNA甲基化[1-2]。前列腺癌新发病例已超越肺癌成为男性恶性肿瘤发病的第一位[3]。目前国内外依然使用前列腺特异性抗原(PSA)和直肠指检对前列腺癌进行筛查,结合B超、CT、MRI、PET/CT 等影像学检查辅助诊断[4]。但PSA 用于诊断前列腺腺癌的特异性较低[5]。近年来的研究发现前列腺癌的发生和发展常伴随着DNA甲基化水平的改变,甚至有非常高的前列腺癌特异性,有望成为前列腺癌诊断的新肿瘤标志[6-8]。根据甲基化研究位点数量的不同,甲基化研究技术可分为特定片段/位点甲基化分析及全基因组甲基化分析。在过去的研究中,主要以特定片段/位点甲基化分析为主,如英国学者采用亚硫酸氢钠测序法(BSP)对前列腺癌和前列腺增生组织中GSTP1、CDH1和P16基因CPG岛甲基化进行检测,发现前列腺腺癌组织中GSTP1和CDH1甲基化与前列腺增生的差异具有统计学意义[9-10],但实验中反复用PCR扩增会使肿瘤特异性存疑。本研究采用的RLGS不需要依赖PCR和杂交动力学就可以全面检测前列腺腺癌腺体细胞及良性前列腺腺体细胞基因组CpG 岛的甲基化状态,获取具有高敏感性和特异性的前列腺腺癌标志物,可望提高前列腺腺癌的早期筛选水平,并为前列腺腺癌的表观治疗提供新靶点。
1 资料和方法 1.1 临床资料本组38例组织标本均来自广东省人民医院泌尿外科2012年1月~2013年12月前列腺癌根治术患者及经尿道前列腺等离子电切术患者的前列腺组织。前列腺癌患者平均年龄为68.3岁(63~78岁),良性前列腺增生组平均年龄为68.6岁(63~73岁)。前列腺腺癌组按照AJCC的TNM分期标准Ⅲ期17 例,Ⅳ期3 例,Gleason评分均为7分,前列腺腺癌患者术前均行前列腺穿刺活检术确诊。良性前列腺增生患者术前PSA均≤4 ng/mL,术后病理证实为良性前列腺增生。入选标本在进行显微切割前后均由2位工作经验大于5年的病理科医生再次阅片确认标本的病理诊断。所有患者均签署研究知情同意书,研究方案经医院伦理委员会审核批准。
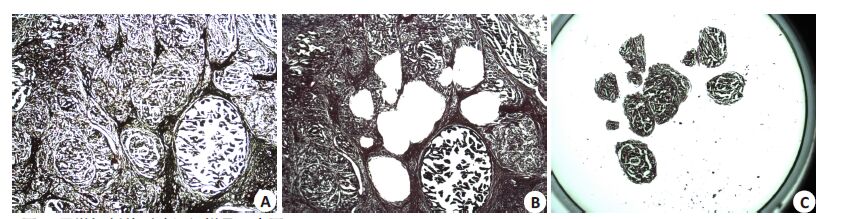
1.2 标本保存、包埋、HE染色、显微切割及DNA提取术中前列腺组织离体后立即在无菌条件下剖开前列腺组织,直视下切取前列腺腺癌组织或直接获取良性前列腺增生组织,切取组织大小为1.0 cm×1.0 cm×1.0 cm,以生理盐水清洗净,立即分装在2.5 mL冻存管中,并迅速予以液氮暂存后转移到-80 ℃冰箱。该过程在前列腺腺癌组织或良性前列腺增生组织离体后10~20 min内完成。收集好组织标本从-80 ℃冰箱取出后,立即移入温度设置为-23 ℃的冰冻切片机中,迅速用O.C.T.包埋,切片厚度8 μm,平铺于Membrane-Slides上,并在室温下行快速染色,具体步骤为:苏木素10 s→自来水洗1~2 s→1%盐酸酒精分化(1 s)→自来水洗1~2 s→伊红2 s→自来水洗1~2 s→80%酒精1 s→90%酒精1 s→100%酒精1 s,染色完成后迅速将玻片放入电热恒温鼓风箱中烘干约2~3 s,室温下开始用激光捕获显微切割仪(LMD6000,Leica,Gemany)进行显微切割(图 1)。组织样本总DNA 提取按QIAGEN试剂盒(产品编号:51106)。
|
图 1 显微切割前列腺组织样品示意图 Figure 1 Laser capture microdissection of prostate samples. A: Tumor lesions marked before microdissection; B: The same slide after microdissection; C: Resected tumor tissues. |
样本DNA以甲基敏感的NotⅠ(GC↓GGCCGC)酶进行消化。低频切割识别序列包含2 个CpG位点。以32 P-dCTP和32P-dGTP标记末端。再以EcoR V消化,产生较短的NotⅠ-EcoR V片段,电泳展开。然后用HinfⅠ于凝胶内消化,产生更短的NotⅠ-HinfⅠ片段。将凝胶旋转90°第2次电泳显影成RLGS图谱。
1.4 焦磷酸测序在4种酶(DNA聚合酶(DNA polymerase)、ATP硫酸化酶(ATP sulfurytase)、荧光素酶(luciferase)和三磷酸腺苷双磷酸酶(Apyrase)的催化下,每一轮测序反应体系中只加入一种脱氧核苷酸三磷酸(dNTP)模板配对,配对成功后在DNA聚合酶的作用下,添加到测序引物的3'末端,同时释放出一个分子的焦磷酸(PPi)。ATP硫酸化酶催化PPi与5'-磷酰硫酸(APS)结合形成ATP,然后在荧光素酶的催化作用下,生成的ATP和荧光素结合形成氧化荧光素,同时产生可见光。CCD感应器检测到光信号后,Pyrogram转化为检测峰,每个峰的高度(光信号)与掺入的核苷酸数目呈正比。Apyrase不断降解未掺入的核苷酸和ATP,淬灭光信号,再生反应体系。
1.5 分析方法用Roche-NimbleGen寻找特定参数甲基化富集峰来判断CpG岛甲基化情况,筛选前列腺癌组CpG岛甲基化而良性前列腺增生组CpG岛不发生甲基化的区域及其对应的基因(高甲基化基因)和前列腺癌组CpG岛不发生甲基化而良性前列腺增生组CpG岛甲基化区域及其对应的基因(去甲基化基因),获得一组前列腺腺癌和良性前列腺增生明显差异甲基化的基因谱。根据差异甲基化基因的PeakScore值(基因甲基化可信度/甲基化的显著性)的大小,筛选出CpG岛区甲基化最显著的7个基因。上传至NIH-DAVID数据库[11]进行差异基因的GO分析,获取差异甲基化基因的分子功能。焦磷酸测序结果统计方法采用选取检测位点甲基化频率的平均值。
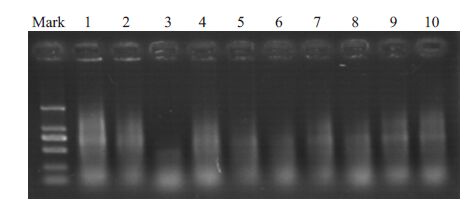
2 结果 2.1 样品DNA完整性检测本实验研究样本均使用激光捕获显微切割捕获单一同质的前列腺腺癌腺体细胞及良性前列腺增生腺体细胞(图 1),将前列腺腺癌组和良性前列腺增生组提取的总DNA用NanodropND-1000 检测,A260/A280值均大于1.8,琼脂糖凝胶电泳(选取其中10份DNA样品见图 2),样本总DNA条带清晰,完整度好。实验证明,激光捕获显微切割技术获取前列腺腺癌和良性前列腺增生腺体细胞可行有效。
|
图 2 组织样品DNA条带 Figure 2 DNA extracted from the tissue samples. 1-5: DNA from prostatic adenocarcinoma; 6-10: DNA from benign prostatic hyperplasia. |
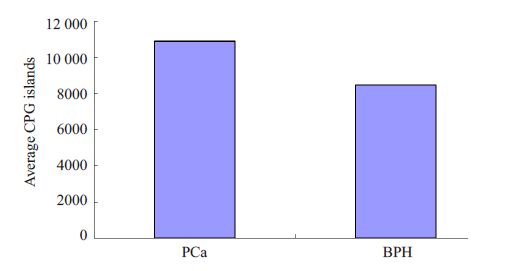
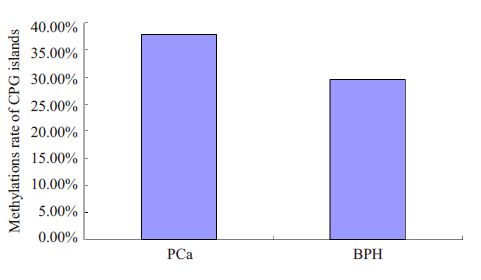
在总共28752个CpG岛中,前列腺腺癌样本平均有10878个CpG岛发生甲基化,基因组CpG岛甲基化发生率为37.83%。良性前列腺增生样本平均有8470 个CpG 岛发生甲基化,基因组CpG 岛甲基化发生率为29.46%,前列腺腺癌基因组CpG岛甲基化位点数量较良性前列腺增生基因组CpG岛甲基化位点平均多2408个。实验证明前列腺腺癌基因组多CpG位点甲基化异常改变的结果(图3、4)。
|
图 3 两组样品平均CpG岛发生甲基化对比 Figure 3 Comparison of average CpG island methylations in the two samples. |
|
图 4 两组样品CpG岛甲基化发生率对比 Figure 4 Comparison of methylation rate of the CpG islands in the two samples. |
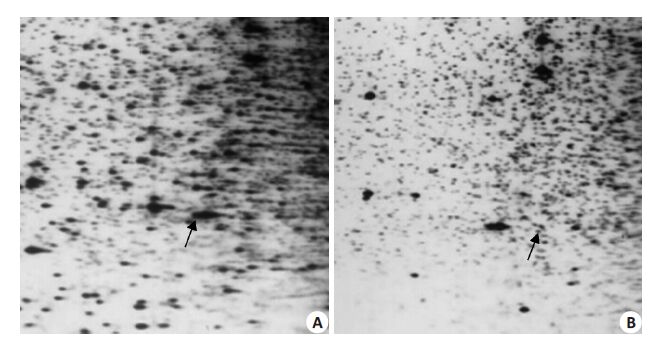
为了辨别前列腺腺癌CpG岛的甲基化程度,我们完成了38例前列腺组织的RLGS。在分析前列腺腺癌的RLGS分布与前列腺增生分布对比时,有较多的二倍体位点的丢失被识别。肿瘤分布中任何丢失的点或者在强度上明显减少的点都被认为是丢失。在38例对比研究中发现前列腺腺癌组与前列腺增生组样本的对比存在明显的差异(图 5A、B)。这些二倍体点丢失的原因包括NotI位点的超甲基化、基因位点的纯合子缺失、用于RLGS分析的3个酵素识别位点中任意一个发生点突变或NotⅠ/EcoR V碎片内的染色体重排断点等等。
|
图 5 二倍体位点 Figure 5 Diploid site (arrows) in BPH (A) and prostate cancer tissues (B) in RLGS distribution. |
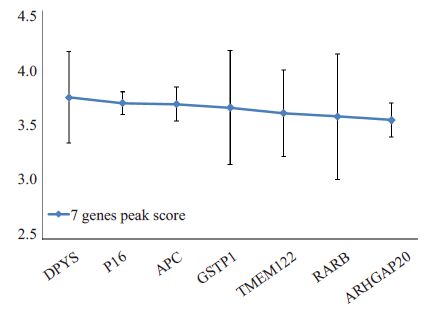
根据差异甲基化区域的PeakScore 值从大到小排列,获取启动子区高甲基化最显著的7个区域及其对应的基因。从大到小分别为DPYS、P16、APC、GSTP1、TMEM122、RARB、ARHGAP20(图 6)。
|
图 6 前列腺腺癌基因组CpG岛高甲基化最显著的7个基因Peak Score值 Figure 6 Peak scores of the 7 genes with the most obvious hypermethylation of the CpG islands in prostatic adenocarcinoma genome. |
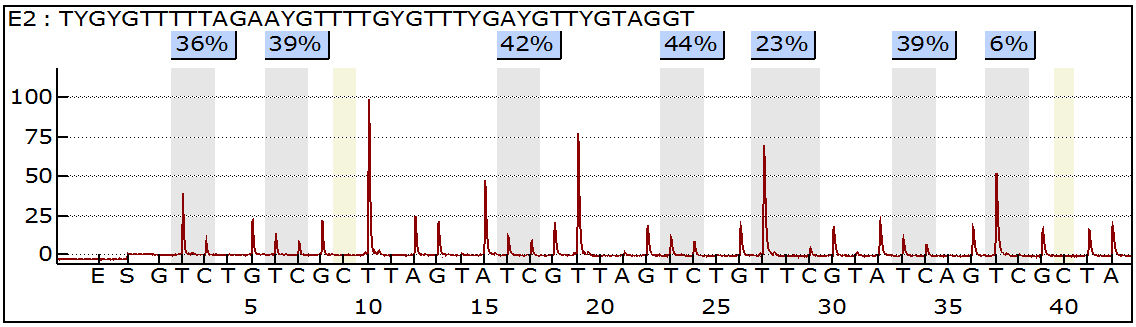
对筛选出来的7个基因进行二代甲基化测序,分别得到对应的甲基化值,从大到小分别为DPYS、APC、RARB、TMEM122、ARHGAP20、P16、GSTP1(表 1)。甲基化频率最高的DPYS测序结果(图 7)。
| 表 1 7个基因CPG岛高甲基化频率检测结果 Table 1 Hypermethylation rate of the CpG islands of the 7 genes |
|
图 7 DPYS焦磷酸测序结果 Figure 7 Sequencing of DPYS PRPP. |
生物信息学分析,可知以上7个基因参与了包括非跨膜蛋白酪氨酸激酶激活、转录共激活因子激活、甲基巴豆酰-CoA羧化酶激活、蛋白结合等分子功能;转录因子活性的正调控、亮氨酸分解代谢、T细胞分化正调控、阳性胸腺T细胞的选择、DNA依赖转录调控等生物过程。其中DPYS参与了13个GO注释的生物功能,50种疾病的发生发展和47个蛋白间的相互作用。
3 讨论在前列腺癌的发生发展中,越来越多研究者认识到表观遗传学改变参与了前列腺癌的早期阶段,甚至癌前病变[12]。其中DNA甲基化是最重要的表观遗传学内容,DNA甲基化异常已被证实与前列腺腺癌密切相关,且在前列腺腺癌的不同阶段均可检测[13]。因此,在早期诊断前列腺癌方面具有巨大的研究价值。RLGS技术的优点包括:①与甲基化特异性PCR或核酸分子杂交技术不同,RLGS不需要预先设计引物或探针,避免了先入为主的偏差和盲目性,覆盖性好,可较完整描绘甲基化谱;②较客观地为下一步甲基化肿瘤标志物的筛选提供目的基因;③ 对未知序列DNA片断的高甲基化亦可检出[14],可为新的肿瘤相关基因的克隆和鉴定提供线索。本研究采用RLGS技术检测前列腺癌组织和正常前列腺组织中5'CpG岛甲基化状态,以期构建前列腺癌组织基因组范围内的5'CpG岛甲基化谱,得到一组在前列腺腺癌基因组中高甲基化的基因。与正常对照相比,样本中缺失或信号减弱的点表示高甲基化的CpG岛。相反,新出现或信号增强的点则表示低甲基化的CpG岛。对可能存在的前列腺癌相关但未测定序列进行测序和功能检测,为新的前列腺癌相关基因的克隆、定位和鉴定提供线索,筛选出特异性甲基化标志物可以有效补充目前前列腺癌早期诊断的不足。本实验选用富含CG的酶切位点作为路标,用可识别CpG岛甲基化位点的限制酶将基因组DNA切割成有甲基化和没有甲基化的两种DNA片断。酶切片段末端用同位素标记,然后通过二维电泳(图 5),放射自显影将上述两种片断分开;再参照特殊的内切酶DNA片断序列文库,得到相应的DNA片断序列,比对人类基因组DNA的NotI/EcoRV边缘文库。当一个孔板中的复制品与基因组DNA混合时,对于这个特定的孔板来说,合成的RLGS凝胶就是一个由一系列确定的点来显示增加强度的标准分布。这一现象是在一系列的32块孔板的行和列中混合操作的。因此,我们在这一文库中绘制了确切的孔板、行和列各自的RLGS点,并且由此得到了RLGS点克隆的快速访问,对比分析得出前列腺腺癌CPG岛发生差异性的甲基化位点比良性前列腺增生组平均多1587个,基因组总体甲基化发生率两组间具有明显的统计学差异(图 4)。
激光捕获显微切割技术(LCM)[15]进行前列腺腺体细胞的获取能有效的避免了腺体细胞外的其他细胞如肌肉及纤维等细胞的影响,保证了实验结果的准确,使疾病相关基因的筛查更具特异性。本实验通过对所有样本的甲基化谱进行差异甲基化分析,发现只在前列腺腺癌发生甲基化而在良性前列腺增生不发生甲基化的CpG岛有366个;只在良性前列腺增生发生甲基化而在前列腺腺癌不发生甲基化的CpG岛有193个。由此可见,前列腺腺癌的发生可能是基因组多位点的甲基化异常改变并通过复杂生物过程相互作用的结果。DNA甲基化抑制基因转录主要通过染色体重塑。当启动子区CpG岛发生甲基化时,甲基化胞嘧啶结合蛋白以及各种染色体重塑相关的复合物,包括组蛋白去乙酰化酶,会被吸引到DNA甲基化区域。组蛋白去乙酰化酶可以使组蛋白去乙酰化并有助于维持核小体紧密连接,保持转录沉默状态。同样,当DNA发生去甲基化时,则不会吸引组蛋白去乙酰化酶,组蛋白将继续保持乙酰化并促进基因转录[16]。许多基因已被确定在前列腺和其他癌症中异常甲基化,这些基因主要包括DNA损伤修复基因、激素应答基因、细胞周期调控基因、肿瘤抑制基因、凋亡基因、肿瘤细胞浸润和转移基因等[10, 17]。本研究利用Roche-NimbleGen寻找特定参数甲基化富集峰来判断CpG岛甲基化,通过PeakScore值的大小筛得7个基因,这些基因均为高密度CpG岛甲基化基因。通过生物信息学分析,这些基因参与了多个生物功能作用途径,可能在前列腺腺癌的发病中发挥重要的作用。DPYS参与了13个GO注释的生物功能,50种疾病的发生发展和47个蛋白间的相互作用,其高甲基化改变可能在前列腺腺癌中也存在重要的意义,DPYS基因CPG岛7位点焦磷酸测序显示甲基化频率为32.7%,是我们后续研究的重点。DPYS基因位于8号染色体q11,由DPYS的注释生物功能我们了解到,它参与了免疫应答,信号转导等生物过程,在自然杀伤细胞介导的细胞毒作用和T细胞受体信号通路中扮演重要的角色。目前围绕DPYS在前列腺癌领域的研究屈指可数,英国学者通过对367例前列腺癌组织标本甲基化检测中发现,DPYS甲基化与PSA、GS评分一样,可作为一个独立的指标预测前列腺癌术后的生存情况[18]。在本组样品RLGS中发现DPYS基因CPG岛甲基化在前列腺腺癌中差异明显。通过二代焦磷酸测序发现DPYS甲基化的各个位点均存在明显的甲基化(图 7),在前列腺腺癌的发生中存在明显的抑制作用,当DPYS基因发生高甲基化改变,DPYS转录抑制,APC介导的免疫应答过程失调,前列腺腺癌免疫抑制功能丧失,最终导致前列腺腺癌发生。
目前,临床上晚期前列腺腺癌多以内分泌治疗为主,开始效疗好,后逐渐发展成为去势抵抗性前列腺腺癌(HRPC)[19]。HRPC除了选用多西他赛,还可选用两类新药:新型激素药物阿比特龙、MDV3100和非激素药物Sipuleucel-T、卡巴他赛[20]。在各种新型药物中,Sipuleucel-T 副作用较轻,是一个免疫治疗药物,并于2010年4月获美国FDA批准用于去势抵抗性前列腺腺癌的治疗,是第一个治疗性肿瘤疫苗。以抗原提呈细胞介导的免疫反应作为基础,不仅有效延长患者生存期,且副作用较轻[21]。我们通过RLGS分析发现前列腺癌基因组CPG岛存在高甲基化,并通过生物信息学分析所得的Peak Score值成功筛选出7个基因,焦磷酸测序发现DPYS基因甲基化最明显,与前列腺腺癌免疫抑紧密相关,可作为候选基因在前列腺腺癌免疫治疗领域做进一步研究。
| [1] | Hatada I, Hayashizaki Y, Hirotsune S, et al. A genomic scanning method for higher organisms using restriction sites as landmarks[J]. Proc Natl Aca Sci USA, 1991, 88 (21): 9523-7. DOI: 10.1073/pnas.88.21.9523. |
| [2] | Hayashizaki Y, Hirotsune S, Okazaki Y, et al. Restriction landmark genomic scanning method and its various applications[J]. Electrophoresis, 1993, 14 (1): 251-8. DOI: 10.1002/(ISSN)1522-2683. |
| [3] | Siegel R, Naishadham D, Jemal A. Cancer statistics, 2013[J]. CA Cancer J Clin, 2013, 63 (1): 11-30. DOI: 10.3322/caac.v63.1. |
| [4] | Heidenreich A, Bastian PJ, Bellmunt JA, et al. EAU guidelines on prostate cancer. part 1: screening, diagnosis, and local treatment with curative Intent-Update 2013[J]. Eur Urol, 2014, 65 (1): 124-37. DOI: 10.1016/j.eururo.2013.09.046. |
| [5] | Thompson IM, Pauler DK, Goodman PJ, et al. Prevalence of prostatecancer among men with a prostate-specific antigen level≤4.0 ng permilliliter[J]. N Engl J Med, 2004, 350 (22): 2239-46. DOI: 10.1056/NEJMoa031918. |
| [6] | Delpu Y, Cordelier P, Cho WC, et al. DNA methylation and cancer diagnosis[J]. Int J Mol Sci, 2013, 14 (7): 15029-58. DOI: 10.3390/ijms140715029. |
| [7] | Formosa A, Lena AM, Markert EK, et al. DNA methylation silencesmiR-132 in prostate cancer[J]. Oncogene, 2013, 32 (1): 127-34. DOI: 10.1038/onc.2012.14. |
| [8] | Nelson WG, Haffner MC, De Marzo AM, et al. Epigenetic changes inProstate cancer[M]. Prostate Cancer: A Comprehensive Perspective, 2013: 169-79. |
| [9] | Miyake T, Nakayama T, Naoi Y, et al. GSTP1 expression predicts poor pathological complete response to neoadjuvant chemotherapy in ER-negative breast cancer[J]. Cancer Sci, 2012, 103 (5): 913-20. DOI: 10.1111/cas.2012.103.issue-5. |
| [10] | Vasiljević N, Wu K, Brentnall AR, et al. Absolute quantitation of DNA methylation of 28 candidate genes in prostate cancer using pyrosequencing[J]. Dis Markers, 2011, 30 (4): 151-61. DOI: 10.1155/2011/157829. |
| [11] | Huang DW, Sherman BT, Lempicki RA. Systematic and integrative analysis of large gene lists using David bioinformatics resources[J]. Nat Protoc, 2009, 4 (1): 44-57. |
| [12] | Truong M, Yang B, Livermore A, et al. Using the epigenetic field defectto detect prostate cancer in biopsy negative patients[J]. J Urol, 2013, 189 (6): 2335-41. DOI: 10.1016/j.juro.2012.11.074. |
| [13] | Majumdar S, Buckles E, Estrada J, et al. Aberrant DNA methylation andprostate cancer[J]. Curr Genomics, 2011, 12 (7): 486. DOI: 10.2174/138920211797904061. |
| [14] | Okuizumi H, Takamiya T, Okazaki Y, et al. Restriction landmark genome scanning[J]. Methods Mol Biol, 2011, 791 : 101-12. DOI: 10.1007/978-1-61779-316-5. |
| [15] | Jensen E. Laser-Capture microdissection[J]. Anat Rec, 2013, 296 (11): 1683-7. DOI: 10.1002/ar.v296.11. |
| [16] | Jones PL, Veenstra GJ, Wade PA, et al. Methylated DNA and MeCP2recruit histone deacetylase to repress transcription[J]. Nat Genet, 1998, 19 (2): 187-91. DOI: 10.1038/561. |
| [17] | Chung W, Kwabi-Addo B, Ittmann M, et al. Identification of novel tumor markers in prostate, colon and breast cancer by unbiased methylation profiling[J]. PLoS One, 2008, 3 (4): e2079. DOI: 10.1371/journal.pone.0002079. |
| [18] | Vasiljević N, Ahmad AS, Thorat MA, et al. DNA methylation gene-based models indicating Independent poor outcome in prostate cancer[J]. BMC Cancer, 2014, 14 (1): 655. DOI: 10.1186/1471-2407-14-655. |
| [19] | Pienta KJ, Walia G, Simons JW, et al. Beyond the androgen receptor: New approaches to treating metastatic prostate cancer. Report of the 2013Prouts Neck Prostate Cancer Meeting[J]. Prostate, 2014, 74 (3): 314-20. DOI: 10.1002/pros.22753. |
| [20] | Heidenreich A, Bastian PJ, Bellmunt J, et al. EAU guidelines onprostate cancer. Part II: treatment of advanced, relapsing, andcastration-resistant prostate cance[J]. Eur Urol, 2014, 65 (2): 467-79. DOI: 10.1016/j.eururo.2013.11.002. |
| [21] | Higano CS, Small EJ, Schellhammer P, et al. Sipuleucel-T[J]. Nat Rev Drug Discov, 2010, 9 (7): 513-4. DOI: 10.1038/nrd3220. |
 2016, Vol. 36
2016, Vol. 36

