肝移植受体需长期终生服用免疫抑制剂,而免疫抑制剂带来的机会性感染和其他毒副作用反过来成为阻碍移植受体长期生存的重要因素之一,因此诱导免疫耐受成为可有效提高移植受体长期生存率的方法[1]。目前相应动物模型已成功建立,但在临床上顺利开展该方案±然面对很大的挑战[1]。这种通过诱导产生的免疫耐受又称为临床操作性免疫耐受,是指移植受体在医生指导下或自行撤除免疫抑制药物后维持稳定的免疫耐受状态[2-7]。研究表明20%~30%的肝移植术后长期稳定生存者经医生指导撤除免疫移植药物后可产生临床操作性免疫耐受[5, 8]。与产生急性排斥反应者相比,这些移植受体的部分免疫细胞除了表现出数量及比例的差异,同时可有部分基因出现特异表达[9]。目前未有相关调节机制的完整报道。
miRNAs是一类小分子单链非编码RNA,通过结合靶mRNA 的3'-UTR 端抑制靶蛋白表达或促使靶 mRNA降解[10]。到目前为止,已报道的与免疫细胞相关的miRNA已超过100种,并发现在下列疾病(包括肾移植和小肠移植术后排斥反应、类风湿性关节炎和红斑狼疮性肾炎)中存在潜在miRNA作为生物标记物[12-17]。然而,miRNA能否调控移植受体特异性基因的表达并作为生物标记物有效区分肝移植术后长期稳定生存者与急性排斥反应者尚未可知。
本研究中,我们用microarray芯片检测比较肝移植受体与正常人群外周单个核细胞miRNA表达谱,发现与移植术后急性排斥反应者相比,稳定肝移植受体有13个miRNAs出现下调表达,我们进一步用实时定量PCR方法验证其中3个miRNAs包括miR-340、miR-106b以及miR-18b 的表达。功能预测研究表明miR-340 和 miR-106b与TGF-β信号通路相关。研究结果提示这些差异表达miRNAs可能在肝移植者免疫调节中发挥重要作用,并可作为诊断免疫耐受的长期生存移植受体的潜在生物标记物。
1 对象和方法 1.1 研究对象本实验选取29位肝移植术后稳定患者(STA 组),及10位经多次组织活检证明肝移植术后排斥反应患者(RJ组),分别抽取其外周血样本供实验研究;另选取17名广州输血中心的年龄匹配无既往肝病史及自体免疫疾病史的健康人(HC组),抽取其血液作为对照。参照 Marc Martinez-Llordella 等[5]人的研究,本实验STA组的入组标准为:(1)肝移植术后存活至少3年以上;(2)服用单种抗排斥药物;(3)移植术后12个月内未出现急性排斥反应;(4)肝功能正常;(5)病史中无自身免疫性肝脏疾病。STA组进一步分为两组,其中STA3Y年组(术后存活时间多于3年且少于5年)患者10例;STA5Y年组(术后存活时间多于5年)患者19例。RJ组包括10名经至少两项组织活检证明有急性排斥反应的移植患者。STA组患者服用单种抗排斥药物至少1年后采集血液样本;RJ组患者一经诊断为急性排斥反应,在其接受类固醇注射或改变抗排斥治疗前应采集其血液样本。病人组与对照组患者的一般情况及临床资料见表一。本研究遵循赫尔辛基宣言(1975),并获本医院伦理委员会(中山大学第一附属医院医学伦理委员会)批准([2011]175),入组病人均签署知情同意书。
1.2 方法 1.2.1 提取PBMC总RNA血液样本收集于EDTA采血管中,用淋巴细胞密度梯度分离法(Ficoll)收集外周血单个核细胞(PBMC),具体操作参照试剂说明书(GE Healthcare,USA)。参照Invitrogen 公司和QIAGEN公司的TRIzol 试剂盒和miRNeasy mini kit 试剂盒的方法提取总RNA。在nanodrop(ND-1000,Nanodrop Technologies)进行RNA浓度和纯度的测定,跑电泳观察总RNA的完整性。
1.2.2 RNA 印记及芯片杂交提取总RNA 后,用 miRCURYTM Hy3TM/Hy5TM Power labeling kit(Exiqon,Vedbaek,Denmark)按厂家指引进行RNA印迹。标记结束后,带有Hy3TM荧光标记的样本在miRCURYTM LNA(v.16.0)(Exiqon)芯片上完成杂交。
1.2.3 数据分析将扫描图片导入GenePix Pro 6.0 software(Axon)软件,获取数据并进行分析。每个 miRNAs 取3 次反应值的平均值,全部样本中亮度值>50 的miRNAs将进行归一化处理。miRNAs的表达值应用中位数标准化的方式进行计算,在标准化表达值后,在MEV software软件中(v4.6,TIGR).用Volcano Plot filtering Volcano Plot filtering. Hierarchical clustering的方法计算得出各组间有差异的miRNAs。1.2.4 qRT-PCR 从新鲜外周血样本中收集PBMCs,用 mir-Vana miRNA 提取试剂盒(Ambion,Austin,TX,USA)提取其总RNA。
芯片杂交显示PBMCs中差异表达的miRNA,用 qRT-PCR 对其进行二次验证。用TaqMan MicroRNA逆转录试剂盒(Applied Biosystems)对10 ng总RNA进行逆转录。qRT-PCR 所用特异性引物详见表S1,反应在Real-time PCR 仪(Applied Biosystems 7900HT Realtime PCR System)上完成,将U6 基因作为内参。 miRNAs的表达值用中位数标准化的方式进行计算,结果用2-ΔΔCt表示,用惠-曼特尼U检验计算其P值。
2 结果 2.1 肝移植受体的一般临床特征入选的39 例肝移植受体中,36 例为术前HBV感染,余3例分别为多囊肝、布加综合征和乙肝阴性的原发性肝细胞癌(HCC)。STA 3Y组中原发性肝细胞癌发生比例(2/8)低于STA 5Y组(8/19)和RJ组(4/10),其他基本特征(包括年龄、术前MELD评分、供肝冷缺血时间、手术时间、无肝期、术中红细胞输注量、ICU滞留时间、分析时外周血白细胞计数)3组间无显著差异。肝功能相关指标包括ALT和AST的表达水平在STA 3Y和 STA 5Y组间无显著差异,而RJ组肝功能水平则低于其他两组。32例肝移植受体服用CNI免疫抑制剂(FK506或环孢素A)作为免疫移植维持方案(其中STA 3Y组8例,STA 5Y组15例,RJ组9例)。肝移植受体的一般临床特征如表 1所示。
| 表 1 各组肝移植受体及健康对照组一般临床特征 Table 1 Characteristics of liver transplant recipients and healthy volunteers |
本研究采用microarray芯片检测肝移植术后长期稳定生存组与急性排斥反应组外周单个核细胞 miRNAs表达,发现有13个miRNAs出现下调表达(表 2)。其中,4个miRNAs(包括miRNA-23b、miRNA- 378、 miRNA-454 和miRNA-18b)表达水平有显著性差异(P<0.01),其余9个miRNAs(包括miRNA-340、miRNA-423-3p、miRNA-138-1*、miRNA-4289、miRNA-378/378c/378d、miRNA-15a、miRNA-505*、miRNA-106b和 miRNA-15b)表达水平有明显差异(P<0.05)。没有发现比急性排斥反应组表达升高的miRNAs。
2.3 肝移植术后长期稳定生存组比健康对照组外周单个核细胞低表达的miRN通过比较肝移植术后长期稳定生存组与健康对照组间外周单个核细胞miRNA的表达谱,发现肝移植术后稳定生存组中,5个miRNAs(包括miRNA-27a、 miRNA-138-1*、miRNA-197、miRNA-378 和miRNAJ3149)表达水平显著低于健康对照组(P<0.01),miRNA-1290 表达量则明显低于健康对照组(P<0.05)。同样的,本研究没有发现比健康对照组表达升高的miRNAs(表 3)。
| 表 2 稳定肝移植受体组比急性排斥反应组下调的miRNAs Table 2 Down-regulated miRNAs in STA recipients compared with RJ recipients |
| 表 3 稳定肝移植受体组比健康对照组下调的miRNAs Table 3 Down-regulated miRNAs in STA recipients compared with HC individuals |
肝移植术后急性排斥反应组外周单个核细胞 miRNA-155 表达量明显高于健康对照组(P<0.05),而 miRNA-1290则出现低表达(P<0.05,表 4)。
| 表 4 肝移植急性排斥反应组与健康对照组差异表达miRNAs Table 4 MicroRNAs different expression in PBMC distinguishes rejection reicipient from healthy control |
根据PCR验证标准,即miRNAs差异倍数>2且P<0.01,我们选择了芯片结果中表达下调的miRNA-340、 miRNA-106b 和miRNA-18b 进行验证(图 1)。实时定量PCR结果表明,这3个miRNAs在稳定肝移植受体组和健康对照组中的表达水平均低于急性排斥组。其中,STA 5Y组与急性排斥组相比,miRNA-106b表达量下(CREBBP)、调了0.58 倍(P<0.01);miRNA-340 表达量下调了0.51倍(P<0.01),miRNA-18b 表达量下调了0.61 倍(P<0.01)。同时,健康对照组中miRNA-18b的表达量比急性排斥反应组下调了0.45倍(P<0.01)。
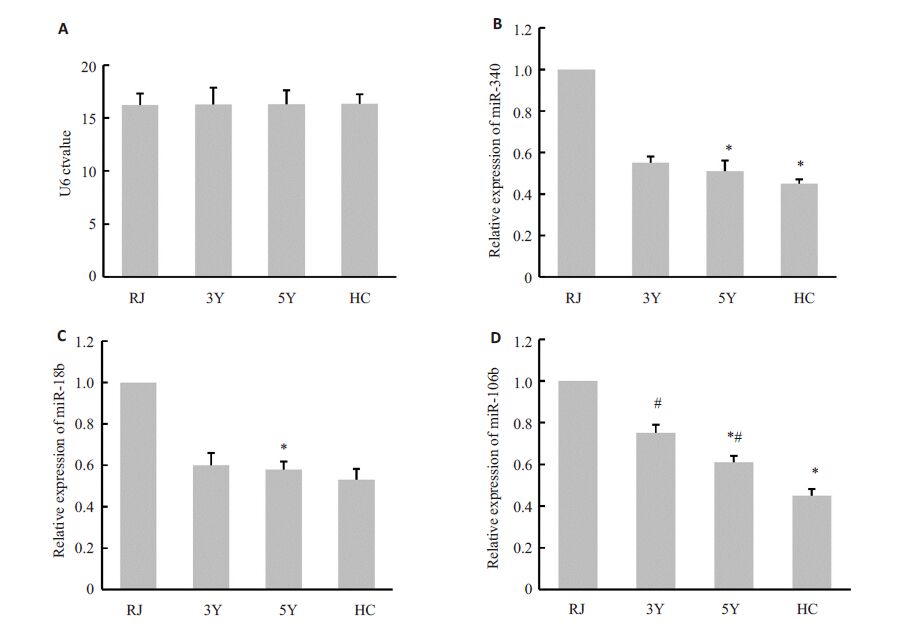
|
图 1 STA 5Y受体组、急性排斥反应组与健康对照组外周单个核细胞差异miRNAs表达量验证 Figure 1 Validation of differential expression of microRNAs inPBMCs from STA 5Y recipients,RJ recipients and healthy subjects. RJ: Groups with acute rejection ; 3Y: Stable group of 3-5 years; 5Y: Stable group over 5 years; HC: healthy control group. A: Expression of U6 as a reference that showed no significant differences between the groups (P>0.05) . B: Compared with RJ and HC groups,the expression of miR-340 was significantly reduced in STA5Y group (*P<0.01 vs RJ group). C: Compared with RJ group,the expression of miR-18b was significantly reduced in STA 5Y group (*P<0.01) ; D: Compared with RJ and HC groups,the expression of miR-106b was significantly reduced in STA 5Y group (*P<0.01 vs RJ group; #P<0.01 vs HC group). Data are expressed as fold changes and the P value was calculated by Mann-Whitney test. |
我们运用DIANA-mirPath 软件预测差异表达 miRNAs(miRNA-106b、miRNA-340和miRNA-18b)可能调控的分子信号通路。结果显示miRNA-340 和 miRNA-106b 可参与调节TGF-β信号通路中一系列分子。其中,miRNA-340可参与调控的该信号通路中的分子包括骨形态发生蛋白4(BMP4)、Noggin 基因(NOG)、SMAD家族蛋白4(SMAD4)、DNA结合蛋白抑制剂(ID2)、DNA结合蛋白抑制剂3(ID3)、促分裂原活化蛋白1(MAPK1)、SMAD家族蛋白7(SMAD7)、 Smad泛素化调控因子1(SMURF1)、活化素A受体1C型(ACVR1C)、SMAD家族蛋白4(SMAD4)、视网膜母细胞瘤类蛋白2(RBL2)、锌指蛋白(Zinc finger)、FYVE结构域蛋白16(ZFYVE16)、CREB 结合蛋白活化素A受体2B型(ACVR2B)、RAS同系家族蛋白A(RHOA)、SMAD家族蛋白6(SMAD6)以及配对同源异性域蛋白2(PITX2);同时,miRNA-106b可参与调控的该信号通路中的分子包括骨形态发生蛋白受体Ⅱ 型(BMPR2),SMAD 家族蛋白5(SMAD5)、 SMURF1、转化生长因子β受体Ⅱ(TGFBR2),SMAD2,视网膜母细胞瘤类蛋白1(RBL1),E2F 转录因子(E2F5),ZFYVE16,磷酸化蛋白2 催化亚基α同工酶(PPP2CA),活化素受体2(ActivinR2)和SMAD7。
TGF-β信号通路中不包含miRNA-18b 的靶基因,但是p53 信号通路中则包含部分miRNA-18b 的靶基因,包括毛细血管扩张共济失调突变基因(ATM)、周期蛋白±赖性激酶2(CDK2)、周期蛋白D2(CCND2)和胰岛素生长因子。同时,受泛素调节的蛋白酶解作用相关的信号通路上也有miRNA-18b靶基因,包括泛素辍合酶E2G1(UBE2G1),泛素辍合酶E2Z(UBE2Z),促分裂原活化蛋白1 E3 泛素化蛋白连接酶(MAP3K1),活化的STAT3蛋白抑制剂(PIAS3)和神经前体细胞表达的发育衰减蛋白4(NEDD4)。
3 讨论寻找及鉴定肝移植术后长期稳定生存者外周血中有意义的生物学标志物,对于肝移植术后患者免疫耐受状态评估及预测具有重大意义,并有助于我们进一步了解移植免疫耐受机制,为临床指导抗排斥治疗提供了重要依据。
MicroRNAs(miRNAs)是一类小分子单链非编码 RNA,它们可直接与靶mRNA 的3' 端非转录区(3'-UTRs)相结合,从而抑制蛋白的表达或促进靶 mRNA的降解[10]。在免疫细胞中目前已发现超过100种miRNAs[10-11],且已有大量证据证明miRNAs可作为肾移植排斥反应[12],小肠移植排斥反应[13],类风湿关节炎[14],败血症[15],狼疮肾炎[16]及多种癌症的潜在生物标志物。然而,miRNA的表达谱是否可用于肝移植患者的免疫耐受状态评估还有待验证。
本研究中我们选取了29 名肝移植术后稳定生存者,通过对其PBMCs进行芯片杂交及Real-time PCR以发现及鉴定潜在的肝移植术后稳定生物标志物。芯片杂交结果显示肝移植术后稳定生存者PBMCs中有13 种MicroRNAs表达下调,其中miR-340,miR-106b,miR-18b经Real-time PCR验证其表达下调。
芯片杂交显示表达下调的13种miRNAs中,部分经先前报道证实与免疫反应相关,并参与了自体免疫疾病的发病机制。近期一项研究证实miR-378 可直接靶向结合颗粒酶B,进而负向调控人NK细胞的细胞毒性[18]。miR-23b被证实在狼疮、类风湿性关节炎及多发性硬化等自体免疫疾病中表达下调,提示它在自体免疫疾病中发挥了一定的抑制作用[19]。miR-15a及 miR-16-1被发现与血液系统及实体的恶性肿瘤相关,已有研究证实它们可以调控CD4阳性T细胞中BCL2的表达,并在复发-缓解型多发性硬化中起到了一定的调控作用[20]。此外,在本研究中表达下调的13 种 miRNAs中,一些已被认为是多种自体免疫疾病的潜在生物标志物。对多发性硬化症而言,环状miR-454可作为一种方便易得的生物标志物,并可依据其进行疾病分期[21]。另有研究显示环状miR-505*在克罗恩病及溃疡性结肠炎中表达下调,提示其可作为急性克罗恩病及溃疡性结肠炎的诊断标志之一[22]。
本研究中13种表达下调的miRNAs中,miR-340,miR-106b 及miR-18b 经Real-time PCR二次验证证明其表达水平下调,进一步利用DIANA-mirPath软件对它们进行分析发现,miR-340和106b预测的靶基因通路与TGF-β明显相关,miR-18b预测的靶基因则与p53信号转导通路、泛素介导的蛋白水解及细胞周期相关。以上结果提示,这3种microRNAs可能在肝移植术后免疫中发挥着重要作用。
miR-106b已经被多个研究证实可调控免疫反应。研究发现在多发性硬化患者外周血的调节性T细胞(简称Treg)中,miR-106b表达上调,且经证实其可抑制免疫调节关键通路——TGF-β通路的激活[23-24]。TGF-β一直被认为是负向免疫调节的关键因子,相关研究证明 TGF-β可抑制T细胞反应并产生免疫耐受[25-26],这种抑制作用通常被认为归结于TGF-β可促进Tregs 的增殖及分化[27-30]。然而另有研究显示在IL-6 或IL-21 共刺激的情况下,TGF-β同样可以促进Th17 的增殖及分化[29, 32-34]。Th17是一种新发现的已被证实可介导自身免疫疾病的效应T细胞[31-32],这提示在某些特定情况下 TGF-β也可促进自身免疫。综上信息提示,本研究中下调表达的miR-106b作为TGF-β通路的抑制因子,也可调控Tregs与Th17之间的动态平衡。miR-106b如何调控二者的平衡进而调节免疫状态,其机制还有待进一步的研究。
本研究中,miR-340在肝移植稳定生存者中同样表达下调,其预测靶基因也与TGF-β通路明显相关。有研究发现,miR-340在多发性硬化症患者的CD4阳性记忆 T细胞中高表达,并可抑制Th2细胞的分化,但可通过降低GATA3 的表达水平来提高促炎性Th1 细胞的反应性[35]。记忆T细胞一直是人实体器官移植术后形成免疫耐受的主要障碍之一[36]。本研究中下调的miR-340,是否是通过调节记忆T细胞的功能来促进免疫耐受,其机制也有待于更深入详尽的研究。
目前关于miR-18的研究,大部分集中于其在恶性肿瘤中所起的作用[37-38],其在免疫反应中的作用则很少被关注。本研究中对miR-18的靶基因预测分析显示,一些靶基因与p53信号转导通路相关。p53是诱导细胞凋亡及细胞周期停滞中的关键调控因子,它在炎症反应及自体免疫反应中也起着重要作用。经证实p53可抑制炎症反应,并且是多种自体免疫疾病如类风湿性关节炎及实验性自身免疫性脑脊髓炎中的保护因子[39-42]。然而另有研究证实,p53也可以通过促进产生自身抗体和促炎症因子,及促进淋巴细胞聚集,从而加剧免疫反应[43-44]。因此,p53在自体免疫中所起的作用究竟为何目前仍不明确。本研究所发现肝移植稳定生存者下调表达的miR-18部分靶基因参与了p53信号转导,这为明确p53在自体免疫中所起的作用提供了参考信息。
值得一提的是,miR-18b的预测靶基因中一些分子与泛素介导的蛋白水解信号途径有关。先前已有实验证实,参与泛素介导的蛋白水解过程的多种泛素连接酶,在负向调控抗原受体信号转导中发挥了重要作用[45-46],并可促进诱导淋巴细胞成为无能细胞[47-48]。以上信息提示泛素介导的蛋白水解过程在各类免疫反应(包括器官移植后的排斥反应)中都发挥了重要作用。因此,进一步的研究miR-18在泛素介导的蛋白水解过程中发挥的作用,有助于弄清泛素介导的蛋白水解在肝移植免疫反应中的作用。
综上所述,本研究发现肝移植术后稳定生存者比有排斥反应者其miR-340,miR-106b 及miR-18b 表达明显下调。进一步分析发现,miR-340和106b预测的靶基因与TGF-β通路明显相关,miR-18b预测的靶基因则与 p53信号转导通路、泛素介导的蛋白水解相关,以上信息提示这些microRNAs在肝移植术后的免疫反应中发挥着重要调控作用。我们根据每个病人的综合情况,已指导部分病人临床减药或停药,几个月来这些病人暂未发生排斥反应。这些microRNAs在肝移植免疫中所起的作用,以及其能否作为临床评估肝移植患者免疫水平的生物标志物,还有待后续实验进一步验证。
| [1] | Turka LA, Wood K, Bluestone JA. Bringing transplantation tolerance into the clinic: lessons from the ITN and RISET for the Establishment of Tolerance consortia[J]. Curr Opin Organ Transplant,2010, 15 (4) : 441-8. DOI: 10.1097/MOT.0b013e32833bd371. |
| [2] | Lerut J, Sanchez-Fueyo A. An appraisal of tolerance in liver transplantation[J]. Am J Transplant,2006, 6 (8) : 1774-80. DOI: 10.1111/ajt.2006.6.issue-8. |
| [3] | Koshiba T, Li Y, Takemura M, et al. Clinical, immunological, and pat hological aspects of operational tolerance after pediatric living-donor liver transplantation[J]. Transpl Immunol,2007, 17 (2) : 94-7. DOI: 10.1016/j.trim.2006.10.004. |
| [4] | Martínez-Llordella M, Puig-Pey I, Orlando G, et al. Multiparameter immune profiling of operational tolerance in liver transplantation[J]. Am J Transplant,2007, 7 (2) : 309-19. DOI: 10.1111/ajt.2007.7.issue-2. |
| [5] | Martínez-Llordella M, Lozano JJ, Puig-Pey I, et al. Using transcriptional profiling to develop a diagnostic test of operational tolerance in liver transplant recipients[J]. J Clin Invest,2008, 118 (8) : 2845-57. |
| [6] | Tryphonopoulos P, Ruiz P, Weppler D, et al. Long-term follow-up of 23 operational tolerant liver transplant recipients[J]. Transpl- antation,2010, 90 (12) : 1556-61. DOI: 10.1097/TP.0b013e3182003db7. |
| [7] | Nafady-Hego H, Li Y, Ohe H, et al. The Generation of donor- specific CD4 + CD25 ++ CD45RA + naive regulatory T cells in opera- tionally tolerant patients after pediatric living-donor liver transpl- antation[J]. Transplantation,2010, 90 (12) : 1547-55. DOI: 10.1097/TP.0b013e3181f9960d. |
| [8] | Charlton MR, Wall WJ, Ojo AO, et al. Report of the first international liver transplantation society expert panel consensus conference on renal insufficiency in liver transplantation[J]. Liver Transpl,2009, 15 (11) : S1-34. DOI: 10.1002/lt.v15:11. |
| [9] | Turka LA, Lechler RI. Towards the identification of biomarkers of transplantation tolerance[J]. Nat Rev Immunol,2009, 9 (7) : 521-6. DOI: 10.1038/nri2568. |
| [10] | O'connell RM, Rao DS, Chaudhuri AA, et al. Physiological and pathological roles for microRNAs in the immune system[J]. Nat Rev Immunol,2010, 10 (2) : 111-22. DOI: 10.1038/nri2708. |
| [11] | Lodish HF, Zhou BY, Liu G, et al. Micromanagement of the immune system by microRNAs[J]. Nat Rev Immunol,2008, 8 (2) : 120-30. DOI: 10.1038/nri2252. |
| [12] | Anglicheau D, Sharma VK, Ding R, et al. MicroRNA expression profiles predictive of human renal allograft status[J]. Proc Natl Acad Sci USA,2009, 106 (13) : 5330-5. DOI: 10.1073/pnas.0813121106. |
| [13] | Sotolongo B, Asaoka T, Island E, et al. Gene expression profiling of MicroRNAs in small-bowel transplantation paraffin-embedded mucosal biopsy tissue[J]. Transplant Proc,2010, 42 (1) : 62-5. DOI: 10.1016/j.transproceed.2009.12.018. |
| [14] | Pauley KM, Satoh M, Chan AL, et al. Upregulated miR-146a expression in peripheral blood mononuclear cells from rheumatoid arthritis patient[J]. Arthritis Res Ther,2008, 10 (4) : R101. DOI: 10.1186/ar2493. |
| [15] | Vasilescu C, Rossi S, Shimizu M, et al. MicroRNA fingerprints identify miR-150 as a plasma prognostic marker in patients with sepsi[J]. PLoS One,2009, 4 (10) : e7405. DOI: 10.1371/journal.pone.0007405. |
| [16] | Te JL, Dozmorov IM, Guthridge JM, et al. Identification of unique microRNA signature associated with lupus nephritis[J]. PLoS One,2010, 5 (5) : e10344. DOI: 10.1371/journal.pone.0010344. |
| [17] | Ji J, Shi J, Budhu A, et al. MicroRNA expression, survival, and response to interferon in liver Cance[J]. N Engl J Med,2009, 361 (15) : 1437-47. DOI: 10.1056/NEJMoa0901282. |
| [18] | Wang P, Gu Y, Zhang Q, et al. Identification of resting and type I IFN-activated human NK cell miRNomes reveals microRNA-378 and microRNA-30e as negative regulators of NK cell cytotoxicity[J]. J Immunol,2012, 189 (1) : 211-21. DOI: 10.4049/jimmunol.1200609. |
| [19] | Zhu S, Pan W, Song X, et al. The microRNA miR-23b suppresses IL-17-associated autoimmune inflammation by targeting TAB2, TAB3 and IKK-α[J]. Nat Med,2012, 18 (7) : 1077-86. DOI: 10.1038/nm.2815. |
| [20] | Lorenzi JC, Brum DG, Zanette DL, et al. miR-15a and 16-1 are downregulated in CD4+ T cells of multiple sclerosis relapsing pati- ent[J]. Int J Neurosci,2012, 122 (8) : 466-71. DOI: 10.3109/00207454.2012.678444. |
| [21] | Gandhi R, Healy B, Gholipour T, et al. Circulating microRNAs as biomarkers for disease staging in multiple sclerosis[J]. Ann Neurol,2013, 73 (6) : 729-40. DOI: 10.1002/ana.23880. |
| [22] | Wu F, Guo NJ, Tian H, et al. Peripheral blood microRNAs distinguish active ulcerative colitis and Crohn's disease[J]. Inflamm Bowel Dis,2011, 17 (1) : 241-50. DOI: 10.1002/ibd.21450. |
| [23] | De Santis G, Ferracin M, Biondani A, et al. Altered miRNA expression in T regulatory cells in course of multiple sclerosis[J]. J Neuroimmunol,2010, 226 (1/2) : 165-71. |
| [24] | Petrocca F, Vecchione A, Croc e CM. Emerging role of miR-106b- 25/miR-17-92 clusters in the control of transforming growth factor beta signaling[J]. Cancer Res,2008, 68 (20) : 8191-4. DOI: 10.1158/0008-5472.CAN-08-1768. |
| [25] | Bommireddy R, Engle SJ, Ormsby I, et al. Elimination of both CD4+ and CD8 + T cells but not B cells eliminates inflammation and pro- longs the survival of TGFbeta1-deficient mice[J]. Cell Immunol,2004, 232 (1/2) : 96-104. |
| [26] | Gorelik L, Flavell RA. Abrogation of TGFbeta signaling in T cells leads to spontaneous T cell differentiation and autoimmune disease[J]. Immunity,2000, 12 (2) : 171-81. DOI: 10.1016/S1074-7613(00)80170-3. |
| [27] | Yamagiwa S, Gray JD, Hashimoto S, et al. A role for TGF-beta in the Generation and expansion of CD4 + CD25 + regulatory T cells from human peripheral blood[J]. J Immunol,2001, 166 (12) : 7282-9. DOI: 10.4049/jimmunol.166.12.7282. |
| [28] | Zheng SG, Gray JD, Ohtsuka K, et al. Generation ex vivo of TGF- beta-producing regulatory T cells from CD4+ CD25- precursors[J]. J Immunol,2002, 169 (8) : 4183-9. DOI: 10.4049/jimmunol.169.8.4183. |
| [29] | Bettelli E, Carrier Y, Gao W, et al. Reciprocal developmental pathways for the Generation of pathogenic effector TH17 and regulatory T cell[J]. Nature,2006, 441 (790) : 235-8. |
| [30] | Zheng SG, Wang JH, Stohl W, et al. TGF-beta requires CTLA-4 early after T cell activation to induce FoxP3 and generate adaptive CD4+CD25+ regulatory cell[J]. J Immunol,2006, 176 (6) : 3321-9. DOI: 10.4049/jimmunol.176.6.3321. |
| [31] | Lubberts E, Koenders MI, Oppers-Walgreen B, et al. Treatment with a neutralizing anti-murine interleukin-17 antibody after the onset of collagen-induced arthritis reduces joint inflammation, cartilage destruction, and bone erosion[J]. Arthritis Rheum,2004, 50 (2) : 650-9. DOI: 10.1002/(ISSN)1529-0131. |
| [32] | Veldhoen M, Hocking RJ, Atkins CJ, et al. TGFbeta in the context of an inflammatory cytokine milieu supports de novo differentiation of IL-17-producing T cell[J]. Immunity,2006, 24 (2) : 179-89. DOI: 10.1016/j.immuni.2006.01.001. |
| [33] | Mangan PR, Harrington LE, O'quinn DB, et al. Transforming growth factor-beta ind uces development of the T(H)17 lineage[J]. Nature,2006, 441 (790) : 231-4. |
| [34] | Yang L, Anderson DE, Baecher-Allan C, et al. IL-21 and TGF-beta are required for differentiation of human T(H)17 cells[J]. Nature,2008, 454 (722) : 350-2. |
| [35] | Guerau-de-Arellano M, Smith KM, Godlewski J, et al. Micro-RNA dysregulation in multiple sclerosis favours pro-inflammatory T- cell-mediated autoimmunity[J]. Brain,2011, 134 (Pt 12) : 3578-89. |
| [36] | Jones ND. Memory T cells: how might they disrupt the induction of tolerance?[J]. Transplantation,2009, 87 (9 Suppl) : S74-7. |
| [37] | Dar AA, Majid S, Rittsteuer C, et al. The role of miR-18b in MDM2-p53 pathway signaling and melanoma progression[J]. J Natl Cancer Inst,2013, 105 (6) : 433-42. DOI: 10.1093/jnci/djt003. |
| [38] | Murakami Y, Tamori A, Itami S, et al. The expression level of miR- 18b in hepatocellular carcinoma is associated with the grade of malignancy and prognosi[J]. BMC Cancer,2013, 13 (2) : 99. |
| [39] | Simelyte E, Rosengren S, Boyle DL, et al. Regulation of arthritis by p53: critical role of adaptive immunity[J]. Arthritis Rheum,2005, 52 (6) : 1876-84. DOI: 10.1002/(ISSN)1529-0131. |
| [40] | Yamanishi Y, Boyle DL, Pinkoski MJ, et al. Regulation of joint destruction and inflammation by p53 in collagen-induced arthritis[J]. Am J Pathol,2002, 160 (1) : 123-30. DOI: 10.1016/S0002-9440(10)64356-8. |
| [41] | Okuda Y, Okuda M, Bernard CC. Regulatory role of p53 in experimental autoimmune encephalomyelitis[J]. J Neuroimmunol,2003, 135 (1/2) : 29-37. |
| [42] | Kuan AP, Cohen PL. p53 is required for spontaneous autoantibody production in B6/lpr lupus mice[J]. Eur J Immunol,2005, 35 (5) : 1653-60. DOI: 10.1002/(ISSN)1521-4141. |
| [43] | Campbell HG, Slatter TL, Jeffs A, et al. Does Δ133p53 isoform trigger inflammation and autoimmunity?[J]. Cell Cycle,2012, 11 (3) : 446-50. DOI: 10.4161/cc.11.3.19054. |
| [44] | Naramura M, Jang IK, Kole H, et al. c-Cbl and cbl-b regulate T cell responsiveness by promoting ligand-induced TCR down-modulation[J]. Nat Immunol,2002, 3 (12) : 1192-9. DOI: 10.1038/ni855. |
| [45] | Yokoi N, Komeda K, Wang HY, et al. Cblb is a major susceptibility gene for rat type 1 diabetes mellitus[J]. Nat Genet,2002, 31 (4) : 391-4. |
| [46] | Davis M, Ben-Neriah Y. Behind the scenes of anergy: a tale of three E3[J]. Nat Immunol,2004, 5 (3) : 238-40. DOI: 10.1038/ni0304-238. |
| [47] | Heissmeyer V, Macián F, Im SH, et al. Calcineurin imposes T cell unresponsiveness through targeted proteolysis of signaling proteins[J]. Nat Immunol,2004, 5 (3) : 255-65. DOI: 10.1038/ni1047. |
| [48] | Anandasabapathy N, Ford GS, Bloom D, et al. Grail: an E3 ubiquitin ligase that inhibits cytokine gene transcription is expr- essed in anergic CD4+ T cell[J]. Immunity,2003, 18 (4) : 535-47. DOI: 10.1016/S1074-7613(03)00084-0. |
 2015, Vol. 35
2015, Vol. 35

