2. 广州市刑事科学技术研究所//公安部法医病理学重点实验室 ;
3. 广东省法医遗传学重点实验室, 广东 广州 510000
2. Guangzhou Institute of Criminal Science and Technology/Key Laboratory of Forensic Pathology in Ministry of Public Security ;
3. Guangdong Province Key Laboratory of Forensic Genetics, Guangzhou 510000, China
水中尸体的溺死诊断是法医工作者常见的难题[1-3]。目前,国内外对溺死诊断方法主要靠尸体解剖和硅藻检验等综合分析[4]。硅藻检验方法中传统强酸消化法应用最为普遍,但其易污染、硅藻回收率低[5]。本课题组前期建立的微波消解-真空抽滤-电镜扫描法大大提高了硅藻检验的特异性及硅藻检出率,如对硅藻进行定量与定性检测还有助于推断溺死地点[5]。但该方法在强酸消化、微波消解的过程中破坏了蓝藻、绿藻等与溺死相关的藻类。有报道称绿藻[6]、蓝藻[7]等藻类也存在与溺死尸体脏器中。为给溺死诊断提供快速、灵敏的新方法,近年来一些法医学者报道使用PCR技术检测藻类特异性DNA片段来诊断溺死,如Tie 等[6]使用针对蓝藻的16SrDNA片段特异性引物诊断溺死,余政梁[8]等人使用检测硅藻的SSU基因特异性引物来诊断溺死。
目前,使用PCR技术检测藻类DNA特异性片段相关报道大多使用针对一种门类的藻类特异性引物来诊断溺死,不能同时扩增其它与溺死相关藻类(如硅藻、蓝藻、绿藻等)。为能同步检测多种随溺液进入体循环的溺死相关藻类,本研究使用针对蓝藻、硅藻、绿藻等多种藻类特异性引物,扩增藻类16SrDNA特异性片段,来检测溺死动物与水中尸体脏器样本中藻类16SrDNA特异性基因片段,旨在为溺死诊断提供新方法。
1 材料与方法 1.1 仪器与试剂PCR扩增仪9700(美国AB),PowerPac™电泳仪电源(美国Bio-Rad),Mini-Protean® Ⅱ电泳槽(美国Bio-Rad),3130 毛细管电泳仪(美国AB),恒温混匀仪(珠海博迈杰生物科技有限公司)。
三羟甲基氨基甲烷(Promega),硼酸(Promega),20 mg/mL蛋白酶K(Qiagen),2×Premix Tap(Takara),37%甲醛溶液(AR,广州化学试剂厂),甲醇(AR,广州化学试剂厂),无水碳酸钠(AR,广州化学试剂厂),硝酸银(AR,美国SIGMA),丙烯酰胺(美国SIGMA),N,N-亚甲基丙烯酰胺(美国SIGMA),过硫酸铵(美国Amresco),TEMED(美国Amresco),LIZ500(美国AB),甲酰胺(美国AB)。
30%丙烯酰胺(29∶1)、10%过硫酸铵、5×TBE在制胶前配制。无菌镊及眼科剪等器具使用前严格消毒。
1.2 实验动物分组及模型制作实验动物分组35只大白兔由广东省动物实验中心提供,雌雄不限,体质量2.4~3.4 kg,随机分成3组:生前入水组(n=15)、死后入水组(n=15)和对照组(n=5)。
模型制作在南沙虎门大桥处,将生前入水组实验兔置笼中,沉入海水下0.5 m 5 s 后提出水面10 s,再重新沉入相同海水深处,重复上述步骤直至实验兔溺死,死后在水下0.5 m 处浸泡24 h;死后入水组采用空气栓塞法处死后,置于相同水深浸泡24 h后取出;对照组采用空气栓塞法处死后不做任何处理。
样本收集:(1)取溺死地点水样500 mL,实验兔肺脏、肝脏、肾脏各3 g,-40 ℃冰箱保存;(2)微波消解-真空抽滤-电镜扫描检测过的20例水中尸体的肝脏样本20份由广州市刑事科学技术研究所提供(每例水中尸体采集1份肝脏样本,共20份肝脏样本,其中14份硅藻阳性)(3)硅藻(直链藻、菱形藻、针杆藻、舟形藻、小环藻)由中国科学院水生生物研究所提供,蓝藻(铜绿微囊藻)、绿藻(小球藻)等藻类由暨南大学赤潮与水生生物学研究中心提供。白色念珠菌(真菌),肉毒梭状芽孢杆菌(细菌)由广州市微生物研究所提供。
1.3 藻类,细菌及真菌DNA提取水样2 mL,12 000 r/min离心15 min,去上清;组织样本约500 mg剪碎加入PowerBead Tubes 中,同时加入10 μL PK(20 mg/mL),恒温混匀仪56 ℃,2 h。95 ℃,8 min[7]。使用PowerSoilTM DNA Isolation Kit试剂盒提取组织中藻类,7种已知藻类,细菌及真菌DNA[8]。
1.4 PCR扩增使用文献[9]报道的引物扩增藻类16SrDNA特异性片段,引物对序列如下:359F:5'-GGGGAATCTTCCGCAATGGG-3;781R(a):5'-GACTACAGGGGTATCTAATCCCATT-3',781R(b):5'-GACTACAGGGGTATCTAATCCCTTT-3'。正向引物5’端添加FAM荧光,由上海生工生物工程技术服务有限公司合成。
PCR 扩增总体系为25 μL,内含12.5 μL PremixTap,上下游引物各1 μL(10 μmol/L,下游引物即781R(a)与781R(b)等浓度、体积混合),9.5 μL无离子水,1 μLDNA模板。PCR热循环参数:94 ℃ 5 min;94 ℃ 30 s,60 ℃30 s,72 ℃40 s,共35个循环;最后72 ℃,延伸10 min。
1.5 PCR产物检测5μL 扩增产物经8%聚丙烯酰胺凝胶(C=3%)电泳后银染显带。本实验中使用毛细管电泳(CE)技术测量7种藻类DNA经PCR扩增后的产物大小。LIZ500内标0.3 μL,9 μL甲酰胺,1 μL PCR产物混合后作为毛细管电泳上样样品
1.6 产物测序随机选取溺死组实验兔肝脏阳性样本、本方法检测水中尸体肝脏(硅藻检测阳性)阳性样本各2份及硅藻阴性本方法检测阳性的样本由上海生工生物工程技术服务有限公司测序。结果与NCBI数据库(http://blast.ncbi.nlm.nih.gov/Blast.cgi)比对。
1.7 统计学分析采用SPSS 13.0统计软件进行统计分析。组间率的比较用χ2检验,P<0.05为差异具有统计学意义。
2 结果 2.1 7种藻类PCR产物电泳见图 1使用本文引物扩增7种藻类DNA后,PCR产物经PAGE电泳银染显示结果为阳性且产物片断大小相似,约420 bp(毛细管电泳结果)。
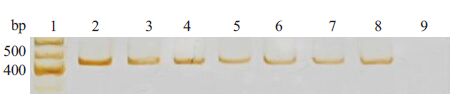
|
图 1 7种藻类PCR产物银染电泳图 Figure 1 Electrophoresis of the PCR product of 7 species of algaewith sliver staining. Lane 1: DL1000 Maker; Lanes 2-8:Microcystis,Melosira varians,Nitzschia sp,Synedra sp,Naviculasp,Chlorella,and Cyclotella meneghiniana,respectively. Lane 9:Blank control. |
本文引物扩增人基因组、兔基因组、真菌基因组、细菌基因组电泳银染结果为阴性,显示本文所用引物针对溺死相关藻类基因组具有较好的特异性(图 2)。

|
图 2 引物对人、实验兔、真菌、细菌基因组特异性验证 Figure 2 Verification of primer specificity for human,rabbit,fungus and bacteria genes. Lane 1: DL1000 Maker; Lanes 2-6:Positive control (Microcystis),human gene,rabbit gene,Monilia albica,and Clostridium acetobutylicum; Lane 7: Blankcontrol. |
生前入水组(n=15)、死后入水组(n=15)和对照组(n=5)肺、肝、肾的检材中提取的藻类DNA在相同条件下经本文引物PCR扩增,银染显带,藻类16SrDNA检出率结果见表 1。经χ2检验,生前入水组与死后入水组各种脏器间阳性率具有统计学差异(P<0.05,图 3,表 1)。
| 表 1 各组实验兔不同组织器官检出例数及阳性率 Table 1 Detection rate of algae in different organs of the rabbits[n(%)] |

|
图 3 生前入水组兔脏器及水样PCR产物银染图 Figure 3 Sliver staining of the PCR product of drowning rabbitorgans and water sample. Lane 1: DL1000 Maker; Lane 2:Positive control (Microcystis); Lane 3: Blank control; Lanes4-7: Kidney,liver,lung,and water sample of the drowningsite,respectively. |
20份样本中19份样本两种方法检测结果一致(表 2)。6份样本MD-VF-Auto SEM法检测硅藻结果为阴性中,1份肝脏样本硅藻阴性本方法为阳性(图 4)。
| 表 2 PCR法与MD-VF-Auto SEM法检测水中尸体脏器样本藻类检测结果 Table 2 Results of PCR and MD-VF-Auto SEM methods for algaedetection in the 20 samples from human corpses found in water |

|
图 4 6份水中尸体硅藻阴性样本PCR产物银染电泳图 Figure 4 Sliver staining of the PCR product of 6 diatom-negativesamples from human corpses found in water. Lane 1: DL1000Maker; Lane 2: Positive control (Microcystis); Lane 3: Blankcontrol; Lanes 4-9: 6 diatom-negative samples. |
溺死组实验兔2份肝脏阳性样本测序序列比对结果:1份与蓝藻同源性较高(铜绿微囊藻,同源性99%)。1份与硅藻同源性较高(直链藻,同源性99%)。2份水中尸体肝脏样本测序序列比对结果与硅藻同源性较高(直链藻,同源性为99%)。1 份水中尸体肝脏样本MD-VF-Auto SEM法检测硅藻为阴性本方法为阳性的样本中藻类与蓝藻同源性较高(铜绿微囊藻,同源性99%)。
3 讨论本文引物为文献报道针对在水环境中存在广泛的蓝藻、硅藻等藻类同时不能扩增出细菌及古生菌等菌类的16SrDNA特异性片段中V3,V4区引物,用于对藻类进行分类及系统发育关系研究[9]。但目前普遍认为16SrDNA是功能与结构上一保守基因,有时不能充分区分一些物种序列之间的差异性[10]。此外,16SrDNA还用于调查自然环境中藻类的存在以及其丰富度[11]。本对引物除能扩增出蓝藻、硅藻外,我们研究发现此对引物还可扩增绿藻等溺死相关藻类。上述藻类在水环境如江河、溪流、湖泊和海洋中分布极为普遍[12]。已有报道证实这些藻类存在溺死人体脏器中[6-8, 13]。因此,使用此对引物可检测水中尸体脏器中上述藻类存在进而作为溺死诊断的手段。
本研究在动物实验中,生前入水组实验兔肺脏阳性率100%,肝脏、肾脏阳性率分别为86%、86%,具有较高的阳性率。死后入水组仅2 例实验兔肺脏中发现藻类的存在,可能与水的压力作用使得水中藻类经呼吸道进入肺脏所致[14]。两组实验兔各器官阳性率存在显著差异。仅从图 1、图 2看出,本文所用引物可扩增出蓝藻、硅藻、绿藻等与溺死相关藻类,同时引物扩增实验兔基因组与人基因组,真菌(白色念珠菌)及在腐败物中存在的细菌(肉毒梭状芽孢杆菌)[15]基因组结果为阴性,样本组织0.5 g即可满足实验需求,因此本文中所用引物检测溺死脏器组织中溺死相关藻类具有较高阳性率,较好的藻类特异性与灵敏度。本文取材量低于参考文献[16]所需取材量,理论上如增加取材量,阳性率可能更高。
MD-VF-Auto SEM法是硅藻检验中一种非常可靠方法,具有较高的硅藻回收率,在溺死诊断中已发挥重要作用[5],但在强酸消化、微波消解的过程中破坏了一些与溺死相关藻类如蓝藻、绿藻等浮游生物。本文PCR法对MD-VF-Auto SEM法检测过的6份水中尸体肝脏硅藻结果为阴性的样本进行检测发现,1份样本使用本方法检测结果为阳性。本文PCR法扩增该份样本中藻类16SrDNA特异性片段产物序列比对结果显示与蓝藻(铜绿微囊藻)同源性较高,达99%。因此,在MDVF-Auto SEM法检测水中尸体脏器硅藻为阴性时,检测其它溺死相关藻类如蓝藻等浮游生物存在来推断溺死是必要的。
结果显示,本文引物克服了以往有关PCR扩增藻类用于诊断溺死的报道大多针对单一藻类特异性引物来检测单一门类藻类即不能同时检测其它与溺死相关藻类的局限性,即使用针对蓝藻、硅藻、绿藻等多种藻类同源性序列引物来检测溺死组织样本中多种溺死相关藻类具有较高的阳性率、灵敏度及特异性,操作简便、易于推广,在溺死诊断中有良好的应用前景。
| [1] | Piette MH. De letter E A.drowning: still a difficult autopsy diagnosis[J]. Forensic Sci Int,2006, 163 (1/2) : 1-9. |
| [2] | Aoyagi M, Iwadate K, Fukui K, et al. A novel method for the diagnosis of drowning by detection of Aeromonas sobria with PCR method[J]. Leg Med,2009, 11 (6) : 257-9. DOI: 10.1016/j.legalmed.2009.07.003. |
| [3] | Hu SL, Liu C, Wen JF, et al. Detection of diatoms in water and tissues by combination of microwave digestion, vacuum filtration and scanning electron microscopy[J]. Forensic Sci Int,2013, 226 (1/ 3) : e48-51. |
| [4] | Kane M, Fukunaga T, Maeda H, et al. The detection of picoplankton 16S rDNA in cases of drowning[J]. Int J Legal Med,1996, 108 (6) : 323-6. DOI: 10.1007/BF02432130. |
| [5] | Zhao J, Liu C, Hu SL, et al. Microwave Digestion-Vacuum Filtration-Automated scanning electron microscopy as a sensitive method for forensic diatom test[J]. Int J Legal Med,2013, 127 (2) : 459-63. DOI: 10.1007/s00414-012-0756-9. |
| [6] | Tie J, Uchigasaki S, Haseba T, et al. Direct and rapid PCR amplification using digested tissues for the diagnosis of drowning[J]. Electrophoresis,2010, 31 (4, SI) : 2411-5. |
| [7] | Kakizaki E, Ogura Y, Kozawa S, et al. Detection of diverse aquatic microbes in blood and organs of drowning victims: First metagenomic approach using high-throughput 454-pyrosequencing[J]. Forensic Sci Int,2012, 220 (1/3) : 135-46. |
| [8] | 余政梁, 刘超, 胡孙林, 等. PCR-DHPLC法检测硅藻SSU基因在溺 死鉴定中的应用[J]. 中国法医学杂志,2013 (6) : 457-60. |
| [9] | Nubel U, Garcia-Pichel F, Muyzer G. PCR primers to amplify 16S rRNA genes from cyanobacteria[J]. Appl Environ Microbiol,1997, 63 (8) : 3327-32. |
| [10] | Fox GE, Wisotzkey JD, Jurtshuk PJ. How close is close:16S rRNA sequence identity May not be sufficient to guarantee species identity[J]. Int J Syst Bacteriol,1992, 42 (1) : 166-70. DOI: 10.1099/00207713-42-1-166. |
| [11] | Rudi K, Skulberg OM, Skulberg R, et al. Application of sequencespecific labeled 16S rRNA gene oligonucleotide probes for genetic profiling of cyanobacterial abundance and diversity by array hybridization[J]. Appl Environ Microbiol,2000, 66 (9) : 4004-11. DOI: 10.1128/AEM.66.9.4004-4011.2000. |
| [12] | 王捷. 念珠藻属(蓝藻)的分类及分子系统研究[D]. 太原: 山西大学, 2011. |
| [13] | Ago K, Hayashi T, Ago M, et al. The number of diatoms recovered from the lungs and other organs in drowning deaths in bathwater[J]. Leg Med,2011, 13 (4) : 186-90. DOI: 10.1016/j.legalmed.2011.04.002. |
| [14] | 何方刚, 黄代新, 刘良, 等. PCR-DGGE法检测浮游生物16S rDNA 在溺死鉴定中的应用[J]. 中国法医学杂志,2008 (4) : 234-7. |
| [15] | 王林业. 腐败梭菌病病原诊断与防治[J]. 畜牧兽医科技信息,2011 (9) : 18-9. |
| [16] | 周玉倩. 硅藻18S rRNA鉴别实验家兔水中尸体死因的研究[D]. 芜湖: 皖南医学院, 2013. |
 2015, Vol. 35
2015, Vol. 35
