2. 生物学教研室,徐州医学院 江苏 徐州 221004 ;
3. 苏州大学附属第一医院神经外科,江苏 苏州 215006
2. Department of Biology, Xuzhou Medical College, Xuzhou 221004, China ;
3. Department of Neurosurgery, First Affiliated Hospital of Soochow University, Suzhou 215006, China
胶质细胞系源性神经营养因子(GNDF)是一种新型的神经营养因子,属于转化生长因子-β(TGF-β)超家族成员,在胚胎发育过程中的表达量迅速上调,至成年期维持在较低水平[1]。由于GDNF对多巴胺能神经元特异的营养作用,一直作为治疗帕金森病的潜在药物而备受关注[2]。最近的研究表明,GDNF还是胶质瘤细胞强有力的促增殖因子[3-4],它在胶质瘤细胞中的表达量显著高于正常胶质细胞[5],并且能够促进胶质瘤细胞的迁移[6-7]。目前,有关GDNF对胶质瘤细胞促增殖和迁移机制的研究很多,但是对于gdnf基因高转录的发生机制却知之甚少。
基因的异常高转录通常与基因突变以及表观遗传学改变有关[8]。我们前期的研究发现,胶质瘤细胞中gdnf基因的异常高转录与基因突变无关,而与该基因启动子II区组蛋白H3K9的高乙酰化修饰有关[9-10]。诸多的研究已表明,启动子区组蛋白的高乙酰化修饰可以使该区域染色质中的核小体呈排列松散的开放状态,从而利于转录因子与之的结合[11],进而促进基因的转录[12]。近来的研究发现,gdnf基因启动子II区转录起始位点上游(-186 bp)存在3个连续且高度保守的转录因子Egr-1的结合序列[13],且Egr-1参与了胶质细胞中成纤维细胞生长因子和丙咪嗪诱导的gdnf转录激活[13-15]。由此我们推测,胶质瘤细胞中gdnf基因启动子II区组蛋白高乙酰化可能以升高转录因子Egr-1与之结合能力的方式引发该基因的异常高转录。
为了验证以上假说,我们以大鼠C6星形胶质瘤细胞和正常星形胶质细胞为研究对象,利用染色质免疫共沉淀(ChIP)的方法检测了以上两种细胞中gdnf基因启动子II区Egr-1结合位点区域H3K9的乙酰化水平以及Egr-1与之的结合能力;通过组蛋白乙酰化酶(HAT)抑制剂Curcumin和组蛋白去乙酰化酶(HDAC)抑制剂TSA [16]建立的gdnf基因启动子II区乙酰化改变的C6胶质瘤细胞模型,检验了gdnf基因启动子区Egr-1结合位点区域组蛋白乙酰化修饰水平、Egr-1与之的结合能力以及该基因转录水平之间是否存在必然的联系。
1 材料和方法 1.1 主要试剂大鼠C6星形胶质瘤细胞株和正常星形胶质细胞为本实验室冻存。DMEM/F12培养基胎牛血清购自美国Gibco,ChIP级兔源抗乙酰化组蛋白H3(Lys9)多克隆抗体(货号07-352)、EZ-ChIP试剂盒购自Millipore,兔源抗Egr-1多克隆抗体(货号sc-110X)购自Santa Cruz,Curcumin、TSA和二甲基亚砜(DMSO)均购自SigmaAldrich,TRIzol购自Invitrogen,Prime ScriptTM II逆转录试剂盒、SYBR Premix Ex TaqTM II试剂盒购自大连TaKaRa。
1.2 细胞培养及药物处理将实验室冻存的大鼠正常星形胶质细胞和C6胶质瘤细胞复苏于含有10%胎牛血清(美国Gibco)的DMEM/F12培养基(美国Gibco公司)的培养液中,置于37 ℃,5% CO2饱和湿度培养箱内培养,每两天换1次液。取2~3代生长良好的大鼠C6星形胶质瘤细胞和正常星形胶质细胞用于后续试验。将处于对数生长期的C6胶质瘤细胞,以1×105/ml细胞浓度接种于六孔板,每孔2 ml。待细胞生长至80%汇合度时,将培养液更改为含有0.5%牛血清白蛋白(美国Sigma-Aldrich)的无血清Opti-MEM (美国Invitrogen),在相同条件下培养24 h。然后再更换为含有Trichostatin A (终浓度为200 nmol/L)或Curcumin (终浓度为50 μmol/L)(均购自美国Sigma-Aldrich)的新鲜无血清Opti-MEM培养基继续培养24 h,其中对照组添加等体积的溶剂DMSO。
1.3 ChIP-PCR染色质免疫共沉淀(ChIP)实验使用兔抗乙酰化H3K9抗体、兔抗Egr-1多克隆抗体和正常兔源IgG,按EZ-ChIP chromatin immunoprecipitation kit说明书的步骤操作。ChIP实验后,采用real-time PCR的方法检测目的抗体免疫沉淀下来的DNA。PCR反应体系为:12.5 μl SYBR Premix Ex TaqTM II;上下游引物(F:5' CGAGGAGGTGCAGAGTGAGG3',R:5'GGGAGCAAGAGCACGCAA 3';10 μmol/L)各1 μl以及DNA模板3 μl;加灭菌消毒的双蒸水补充至总体积25 μl。用三步法PCR反应程序(预变性:95 ℃ 2 min;PCR反应:95 ℃ 15 s,60 ℃ 30 s,72 ℃ 20 s,共40个循环)进行扩增。PCR产物大小为283 bp。Real-time PCR后采用2-ΔΔCt法对数据进行相对定量,以%Input的形式计算结果:%Input=2(CtInput-CtChIP)×Input dilution factor×100。Real-time PCR引物由康成生物(上海)公司合成。
1.4 Real-time PCR采用TRIzol (Invitrogen)一步法从细胞中提取总RNA,以1 μg总RNA为模板按Prime ScriptTM II逆转录试剂盒说明行逆转录反应合成cDNA第一链。利用SYBR Premix Ex TaqTM II试剂盒在反应体系(20 μl)下进行实时定量PCR:SYBR Premix Ex TaqTM II 10 μl、上下游引物(10 μmol/L)各0.5 μl、cDNA模板1 μl和无核酶水8 μl。通过LightCycler® 480荧光定量PCR系统(Roche)进行实时荧光定量分析。反应条件设置如下:95 ℃预变性30 s;95 ℃变性20 s,60 ℃退火15 s;72 ℃延伸15 s,扩增40个循环。通过溶解曲线分析PCR产物特异性。每个样品以gapdh基因的mRNA表达作为内参照,通过相对定量(2-△△CT)法计算目的基因在各样品中相对mRNA的表达水平。靶基因和内参基因扩增的上下游引物序列见表 1。
| 表 1 荧光定量PCR引物序列 Table 1 Primer sequence for real-time PCR |
所有数据均采用统计软件SPSS16.0进行处理,计量资料以均数±标准差表示。其中,两样本均数比较采用独立样本t检验,多个样本均数的比较采用单因素方差分析。取P < 0.05表示差异有统计学意义。
2 结果 2.1 超声断裂染色质后DNA的电泳结果大鼠C6胶质瘤细胞和正常星形胶质细胞经超声破碎染色质后各取20 μl直接进行解交联和纯化2%琼脂糖凝胶电泳显示:切割后的DNA片段主要分布在200~1000 bp之间(图 1)。

|
图 1 超声波切割染色质后DNA的电泳图 Figure 1 Sheared DNA size determination by agarose gel electrophoresis. M: DL2000 Marker; Lanes 1-4: Sheared DNA of rat C6 glioma cells; Lanes 5-8: Sheared DNA of rat normal astrocytes. |
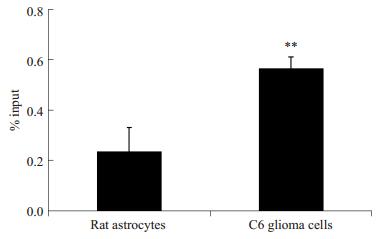
ChIP-PCR结果显示:大鼠C6胶质瘤细胞和正常星形胶质细胞中gdnf基因启动子II区的组蛋白H3K9均发生了不同程度的乙酰化修饰。与正常星形胶质细胞相比,C6胶质瘤细胞中gdnf基因启动子II区Egr-1结合位点区域组蛋白H3K9的乙酰化程度极显著升高(P < 0.01,图 2)。
|
图 2 大鼠C6胶质瘤细胞和正常星形胶质细胞中gdnf基因启动子II区Egr-1结合位点处H3K9乙酰化水平 Figure 2 H3K9 acetylation level of Egr-1 binding sites in gdnf gene promoter region II in rat C6 astroglioma cells and normal astrocytes. **P < 0.01 vs astrocytes. |
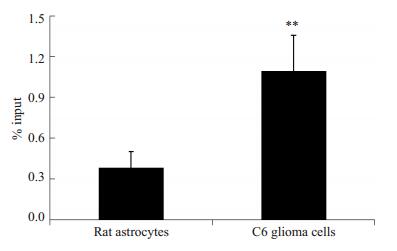
ChIP-PCR结果显示:C6胶质瘤细胞和正常星形胶质细胞中转录因子Egr-1都能够不同程度地与gdnf基因启动子II区潜在的Egr-1结合位点区域结合。与正常星形胶质细胞相比,C6胶质瘤细胞中Egr-1与gdnf基因启动子II区的结合量极显著升高(P < 0.01,图 3)。
|
图 3 大鼠C6胶质瘤细胞和正常星形胶质细胞中转录因子Egr-1与gdnf基因启动子II区的结合情况 Figure 3 Egr-1 binding to gdnf gene promoter region II in rat C6 astroglioma cells and normal astrocytes. **P < 0.01 vs astrocytes. |
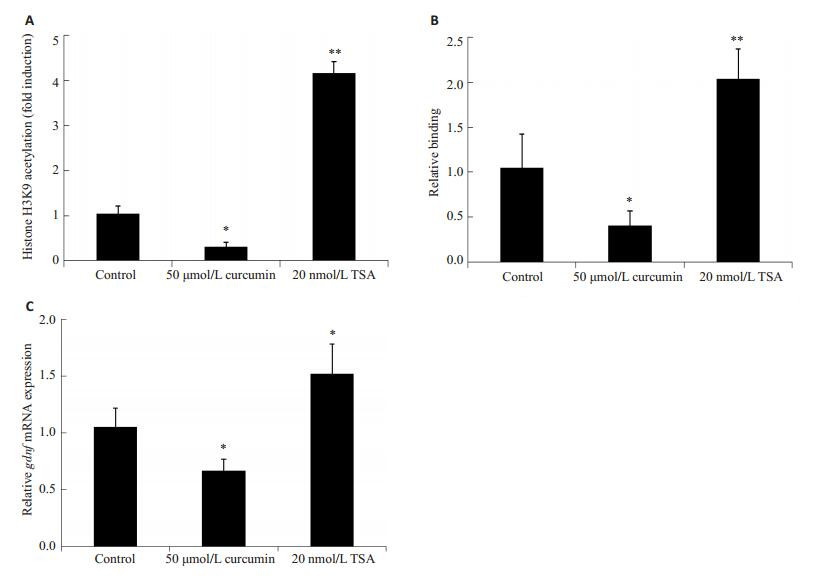
我们采用Curcumin和TSA的药物处理建立了gdnf基因启动子II区Egr-1结合位点处乙酰化水平改变的C6胶质瘤细胞模型,即50 μmol/L Curcumin处理24 h后,显著降低了gdnf基因启动子II区Egr-1结合位点处H3K9的乙酰化水平;相反的,200 nmol/L TSA处理24 h后,显著提高gdnf基因启动子II区Egr-1结合位点处H3K9的乙酰化水平(图 4A)。在此基础上,采用ChIP-PCR和real-time PCR技术检测了gdnf基因启动子II区H3K9乙酰化修饰改变对Egr-1与之的结合能力及该基因转录水平的影响。结果显示,gdnf基因启动子II区Egr-1结合位点处H3K9低乙酰化修饰,减少Egr-1与之的结合的同时,降低了gdnf mRNA的表达(P < 0.05);而且,gdnf基因启动子II区Egr-1结合位点处H3K9高乙酰化修饰,在促进Egr-1与之结合的同时,提高了gdnf mRNA的表达(P < 0.05,图 4B,C)。
|
图 4 大鼠C6胶质瘤细胞中gdnf基因启动子II区Egr-1结合位点H3K9乙酰化修饰介导的Egr-1与之的结合能力调节该基因的转录水平 Figure 4 gdnf gene promoter region II H3K9 acetylationmediated modulation of Egr-1 binding regulates gdnf transcription in C6 astroglioma cells. A: H3K9 acetylation level at the Egr-1 binding site of gdnf gene promoter region II in C6 astroglioma cells after treatment with curcumin and TSA for 24 h; B: Binding status of Egr-1 at gdnf gene promoter region II in cells after treatment with curcumin and TSA for 24 h; C: Relative GDNF mRNA levels in cell after treatment detected with real-time PCR. *P < 0.05, **P < 0.01 vs control group. |
Egr-1是gdnf基因活化的关键转录因子[13-15]。为了探明gdnf基因启动子II区组蛋白H3K9高乙酰化修饰促进该基因高转录的作用机制,我们利用ChIP-PCR的方法检测了大鼠C6星形胶质瘤细胞和正常星形胶质细胞中该启动子区Egr-1结合位点区域组蛋白H3K9的乙酰化状态。K9是组蛋白H3调节基因转录激活最为活跃的乙酰化位点[17-19]。结果显示,C6胶质瘤细胞中gdnf基因启动子II区Egr-1靶向区域发生了组蛋白H3K9的高乙酰化修饰(图 2),与我们之前的研究结果相似[9],表明组蛋白H3K9高乙酰化修饰可能影响到Egr-1与gdnf基因启动子II区的结合。
为了验证以上推测,我们利用ChIP-PCR的方法检测了大鼠C6星形胶质瘤细胞和正常星形胶质细胞中Egr-1与gdnf基因启动子II区潜在的结合位点的结合情况,发现C6胶质瘤细胞中Egr-1能够与gdnf基因启动子II区潜在的结合位点结合,并且较之正常胶质细胞Egr-1的结合量显著升高(图 3),与我们的推测相一致。此外,Shin等[15]人发现缺失Egr-1结合位点显著下调了gdnf基因启动子II区的启动活性,表明Egr-1的结合能够促进gdnf基因的转录活性。由此,我们提出本文的假说,gdnf基因启动子II区组蛋白高乙酰化介导的Egr-1与之结合量的升高促进了C6胶质瘤细胞中该基因的高转录。
为了进一步证实以上假说,我们利用以往的实验结果筛选了Curcumin和TSA处理C6胶质瘤细胞的最佳作用时间及作用浓度[9],并在此基础上建立了gdnf基因启动子II区Egr-1结合位点乙酰化改变的C6胶质瘤细胞模型。发现,Egr-1呈组蛋白H3K9乙酰化依赖的方式结合到gdnf启动子II区,并且促进了gdnf基因的转录,与我们的预期相符(图 4)。
综上所述,本研究发现了大鼠C6星形胶质瘤细胞中,gdnf基因启动子II区Egr-1结合位点区域组蛋白H3K9发生了高乙酰化修饰,并且Egr-1与之的结合能力也显著升高(P < 0.05);gdnf基因的转录水平以及Egr-1与该基因启动子的结合能力,都随着Egr-1结合位点区域乙酰化水平的升高而增强(P < 0.05);相似的,gdnf基因的转录水平以及Egr-1与该基因启动子的结合量,也随着Egr-1结合位点区域乙酰化水平的降低而减少(P < 0.05),表明gdnf基因启动子II区H3K9高乙酰化能够通过上调Egr-1与之的结合促进胶质瘤细胞中该基因的高转录,为探讨胶质瘤细胞中gdnf基因异常高转录的机制提供了新的线索。
| [1] | Strömberg I, Björklund L, Johansson M, et al. Glial cell LineDerived neurotrophic factor is expressed in the developing but not adult striatum and stimulates developing dopamine neurons in vivo[J]. Exp Neurol,1993, 124 (2) : 401-12. DOI: 10.1006/exnr.1993.1214. |
| [2] | Backman CM, Shan LF, Zhang YJ, et al. Gene expression patterns for GDNF and its receptors in the human putamen affected by Parkinson's disease: A real-time PCR study[J]. Mol Cell Endocrinol,2006, 252 (1/2, SI) : 160-6. |
| [3] | Ng WH, Wan GQ, Peng ZN, et al. Glial cell-line derived neurotrophic factor (GDNF) family of ligands confer chemoresistance in a ligand-specific fashion in malignant gliomas[J]. Journal of Clinical Neuroscience,2009, 16 (3) : 427-36. DOI: 10.1016/j.jocn.2008.06.002. |
| [4] | Ku MC, Wolf SA, Respondek D, et al. GDNF mediates glioblastoma-induced microglia attraction but not astrogliosis[J]. Acta Neuropathol,2013, 125 (4) : 609-20. DOI: 10.1007/s00401-013-1079-8. |
| [5] | Yu ZQ, Zhang BL, Ren QX, et al. Changes in transcriptional factor binding capacity resulting from promoter region methylation induce aberrantly high GDNF expression in human glioma[J]. Mol Neurobiol,2013, 48 (3) : 571-80. DOI: 10.1007/s12035-013-8443-5. |
| [6] | Lu DY, Leung YM, Cheung CW, et al. Glial cell line-derived neurotrophic factor induces cell migration and matrix metalloproteinase-13 expression in glioma cells[J]. Biochem Pharmacol,2010, 80 (8) : 1201-9. DOI: 10.1016/j.bcp.2010.06.046. |
| [7] | Song H, Moon A. Glial cell-derived neurotrophic factor (GDNF) promotes low-grade Hs683 glioma cell migration through JNK, ERK-1/2 and p38 MAPK signaling pathways[J]. Neurosci Res,2006, 56 (1) : 29-38. DOI: 10.1016/j.neures.2006.04.019. |
| [8] | Nagarajan RP, Costello JF. Molecular epigenetics and genetics in neuro-oncology[J]. Neurotherapeutics,2009, 6 (3) : 436-46. DOI: 10.1016/j.nurt.2009.04.002. |
| [9] | Yu ZQ, Zhang BL, Ni HB, et al. Hyperacetylation of histone H3K9 involved in the promotion of abnormally high transcription of the gdnf gene in glioma cells[J]. Mol Neurobiol,2014, 50 (3) : 914-22. DOI: 10.1007/s12035-014-8666-0. |
| [10] | Castro LM, Gallant M, Niles LP. Novel targets for valproic acid: up-regulation of melatonin receptors and neurotrophic factors in C6 glioma cells[J]. J Neurochem,2005, 95 (5) : 1227-36. DOI: 10.1111/j.1471-4159.2005.03457.x. |
| [11] | Lee DY, Hayes JJ, Pruss D, et al. A positive role for histone acetylation in transcription factor access to nucleosomal DNA[J]. Cell,1993, 72 (1) : 73-84. DOI: 10.1016/0092-8674(93)90051-Q. |
| [12] | Becker PB, Workman JL. Nucleosome remodeling and epigenetics[J]. Cold Spring Harb Perspect Biol,2013, 5 (9) : 14. |
| [13] | Kim Y, Kim SH, Kim YS, et al. Imipramine activates glial cell line-derived neurotrophic factor via early growth response gene 1 in astrocytes[J]. Prog Neuropsychopharmacol Biol Psychiatry,2011, 35 (4) : 1026-32. DOI: 10.1016/j.pnpbp.2011.02.012. |
| [14] | Obara Y, Nemoto W, Kohno S, et al. Basic fibroblast growth factor promotes glial cell-derived neurotrophic factor gene expression mediated by activation of ERK5 in rat C6 glioma cells[J]. Cell Signal,2011, 23 (4) : 666-72. DOI: 10.1016/j.cellsig.2010.11.020. |
| [15] | Shin SY, Song H, Kim CG, et al. Egr-1 is necessary for fibroblast growth factor-2-induced transcriptional activation of the glial cell line-derived neurotrophic factor in murine astrocytes[J]. J Biol Chem,2009, 284 (44) : 30583-93. DOI: 10.1074/jbc.M109.010678. |
| [16] | Zhang XH, Wharton W, Yuan ZG, et al. Activation of the growthdifferentiation factor 11 gene by the histone deacetylase (HDAC) inhibitor trichostatin A and repression by HDAC3[J]. Mol Cell Biol,2004, 24 (12) : 5106-18. DOI: 10.1128/MCB.24.12.5106-5118.2004. |
| [17] | Kouzarides T. Chromatin modifications and their function[J]. Cell,2007, 128 (4) : 693-705. DOI: 10.1016/j.cell.2007.02.005. |
| [18] | Strahl BD, Allis CD. The language of covalent histone modifications[J]. Nature,2000, 403 (6765) : 41-5. DOI: 10.1038/47412. |
| [19] | Fry CJ, Peterson CL. Chromatin remodeling enzymes: who's on first?[J]. Current Biology,2001, 11 (5) : R185-97. DOI: 10.1016/S0960-9822(01)00090-2. |
 2015, Vol. 35
2015, Vol. 35
