蚊是一类重要的医学昆虫,不仅叮咬骚扰人类,同时还可能传播多种疾病,如疟疾、登革热、丝虫病、流行性乙型脑炎等,对人类的健康产生极大的危害。长期以来,由于蚊基因组中高度重复序列的广泛存在,给一些重要基因特别是非编码序列的分子克隆和功能分析带来了困难,也制约了媒介蚊虫及其传播疾病的防治。近年来随着二代测序等生物技术的的快速发展,蚊虫基因组学、转录组学、小RNA组学等领域的研究得到了迅速提升,也给蚊虫媒介生物学及其传播疾病的防治研究,提供了广阔的、实用的大数据分析平台。本文对该领域的研究进展综述如下。
1 蚊虫的基因组学研究基因组学(genomics)是研究生物基因组和如何利用基因的一门学问。迄今为止,已有多种蚊虫包括冈比亚按蚊、致倦库蚊、埃及伊蚊的基因组被报道[1-3]。作为入侵性最强的媒介蚊虫,世界各地也有多个团队在尝试解析白纹伊蚊的基因组,但一直未获成功。我们对白纹伊蚊进行的Genome Survey显示:其基因组巨大,约3Gb;variation很高,大于0.5%;且重复序列非常多。这样的基因组序列无论是用一代测序技术还是二代测序技术都难以完成。后来我们采用了Isofemale family的策略纯化基因组背景,结合全基因扩增(whole genome amplification, WGA)技术扩增单只蚊蛹的基因组DNA,并构建了不同插入长度的测序文库进行了大量测序,现已基本完成了白纹伊蚊的测序、组装和注释。目前已完成的重要蚊虫基因组特征见表 1 [1-8]。
| 表 1 目前已发布的蚊虫基因组一览表 Table 1 Published mosquito genome so far |
对比以前发表的蚊基因组序列,我们发现不同蚊种的基因组大小有很大的不同。如疟疾媒介冈比亚按蚊的基因组约为278 Mb,丝虫病和乙型脑炎媒介致乏库蚊的基因组约为540 Mb,黄热病和登革热媒介埃及伊蚊的基因组约为1376 Mb,而我们对白纹伊蚊的初步结果显示其基因组高达3000 Mb。通过对白纹伊蚊组装基因组的注释分析,我们发现重复序列在其基因组中占很大比例,将近其基因组的65%。对比其他蚊种已发表的基因组序列,重复片段都是基因组的主要构成,而且其所占比例与基因组的大小呈正相关,如冈比亚按蚊基因组中重复序列为14%左右,致乏库蚊基因组中重复序列为29%左右,埃及伊蚊基因组中重复序列为44%左右。同为伊蚊属的埃及伊蚊和白纹伊蚊其重复片段比例为什么在基因组中会有如此大的不同呢?通过对重复序列的主要组成Transposal Element(TE)的分析,我们发现白纹伊蚊TE的产生数目和插入率较埃及伊蚊要高很多,特别自70百万年以来尤为明显,而这正是埃及伊蚊与白纹伊蚊在生物进化上的分离点。这是首次应用大数据分析平台找到的媒介伊蚊基因组进化的证据。
蚊虫基因组序列的揭示为其基因的克隆鉴定和功能分析提供了很好的平台。由于蚊基因组中高度重复序列的广泛存在,给一些基因特别是非编码序列的分子克隆带来了困难,而基因组序列的提供给这一问题的解决带来了福音。利用已获得的白纹伊蚊基因组序列,我们已顺利克隆了其嗅觉结合蛋白(OBP)和嗅觉受体(OR)基因及其调控序列,为其嗅觉发生分子机制的阐明奠定了基础[9]。Criscione等[10]通过比较斯氏按蚊雌蚊和雄蚊的基因组DNA和RNA样本的Illumina测序结果,发现了一个Y染色体特有的基因GUY1。Hall等[11]则发明了一种更为有效的染色体熵法,该方法的优势是仅利用高通量测序获得的基因组和转录组数据,而不需要一个固定在染色体上的参考基因组来进行比对。通过比对两种疟疾重要传播媒介斯氏按蚊和冈比亚按蚊的高通量基因组和转录组数据,系统性地在斯氏按蚊和冈比亚按蚊中各发现了3个Y基因。同时通过对鉴定的Y基因进行生物进化分析,结果表明按蚊的Y染色体进化迅速。随后,Hall等[12]又使用染色体熵的方法比对了雄性和雌性埃及伊蚊基因组DNA和RNA的Illumina测序数据,筛选并鉴定了一个新的基因myo-sex。myo-sex基因几乎只存在于雄蚊的基因组,但是由于基因重组偶尔地能在雌蚊的基因组中发现,具有雄性偏好性,是一个可能具有性别拮抗效应的肌球蛋白重链基因。
蚊虫基因组数据也为蚊虫性别决定网络底部基因dsx的研究提供了依据。dsx是性别决定网络中的调控核心,主要行使决定体细胞和生殖细胞性别的功能[13],也可调控中枢神经相关基因fruitless,进而调节性行为[14]。冈比亚按蚊基因组数据发布之后,Scali等[15]率先鉴定了冈比亚按蚊dsx的性别特异性转录本,其横跨2号染色体85kb的区域,通过选择性拼接产生多个外显子组成的雌性和雄性特异性转录本。而随着2014年斯氏按蚊基因组的发布,有研究者将Scali等[16]报导的Angdsx与刚发布的斯氏按蚊基因组和转录组进行序列比对,发现一致性达到了97%,而与冈比亚按蚊基因组及转录组序列一致性仅为85%。Scali等[15]在埃及伊蚊中发现了两种雌性特异性的可变剪接方式,这不同于黑腹果蝇和冈比亚按蚊具有的特异性DsxF。
2 蚊虫的转录组学研究转录组学(transcriptomics)是一个活细胞所能转录出来的所有RNA的总和,是研究细胞表型和功能的一个重要手段。传统上用于转录组数据获得和分析的方法主要有基于杂交技术的芯片技术包括cDNA芯片和寡聚核苷酸芯片,但目前使用最普遍的是RNA-seq即转录组测序技术。基于Illumina高通量测序平台的转录组测序技术能够在单核苷酸水平对任意物种的整体转录活动进行检测,在分析转录本的结构和表达水平的同时,还能发现未知转录本和稀有转录本,精确地识别可变剪切位点以及cSNP(编码序列单核苷酸多态性),提供最全面的转录组信息。相对于传统的芯片杂交平台,转录组测序无需预先针对已知序列设计探针,即可对任意物种的整体转录活动进行检测,提供更精确的数字化信号,更高的检测通量以及更广泛的检测范围,是目前深入研究转录组复杂性的强大工具。
巨蚊属是蚊科中三种不吸血的蚊属之一,其幼虫阶段以同在小型水体中孳生的白纹伊蚊和埃及伊蚊为食,两性成蚊均不吸血,以植物汁液和花蜜为食[17]。为了探究巨蚊与其它吸血蚊种在搜寻宿主方面的基因水平上有何差异,国外有学者从巨蚊上分离出触须、触角和身体其他部分,分别提取这三部分的RNA,利用RNA-seq技术,将获得的序列片段从头组装,与目前已公布的致倦库蚊、冈比亚按蚊、埃及伊蚊基因组数据进行系统进化树分析,发现巨蚊与埃及伊蚊的种属关系最近,并且在上述四种蚊种中均发现了编码气味分子受体(odorant receptor, OR)蛋白和离子转移受体(ionotropic receptor, IR)蛋白的基因,但值得注意的是,巨蚊受体蛋白的表达量与丰度上较其它蚊种都有所降低[18]。因为这些受体蛋白被认为与吸血昆虫搜寻宿主气味分子如CO2有关[19-21],所以,巨蚊在长期的生物进化过程中,丧失了原本存在的吸血习性。
蚊唾液腺蛋白与其吸血传病密切相关。国外有学者提取白纹伊蚊雌性成蚊的唾液腺RNA后进行转录组和蛋白质组分析,发现至少有32个基因在雌性成蚊的唾液腺中表达程度或者增高或者降低,另外有17个基因表达在雌性成蚊唾液腺和雄性成蚊中,但不表达在雌性成蚊的其他组织中[22]。通过分析发现,其中大约三分之一的基因功能表现在吸血、消化糖、免疫应答等方面,但是并未发现其余基因的明确功能,所以非常有可能是长期吸血的过程中进化出的新的功能分子。利用同样的方法,分析冈比亚按蚊[23-24]、斯氏按蚊[25-26]、达氏按蚊[27]、埃及伊蚊[28-29]、白纹伊蚊[22]、致倦库蚊[30-33]和致死按蚊(Anopheles funestus)[32]的唾液蛋白相关的转录组,可以将这些蛋白归纳为:(1)昆虫唾液腺中普遍存在的唾液蛋白,包括抗原-5蛋白家族、核酸酶、碳水化合物水解酶等;(2)在吸血的长角亚目昆虫(包括白蛉、蚋、蠓等[33-34])中丰富表达的D7蛋白;(3)仅在蚊唾液腺中存在的蛋白,包括30 000左右的过敏原蛋白家族(allergen family)和一些粘蛋白。
很多昆虫都被发现具有一种独特的生物学现象--滞育(diapause)。昆虫的滞育现象被认为是一种休眠的形式,在昆虫发育时遇到不适宜的环境时,就会马上由体内激素调节并控制,暂时停止发育[35]。白纹伊蚊被发现同样具有滞育现象,这是它能适应环境气候变化,实现快速扩张入侵的生物学基础之一[36-37]。白纹伊蚊的雌性成蚊在每日受到较短时间的光照后,产下的卵不会立即孵化,这便是一种滞育的现象。有趣的是,同是伊蚊属的埃及伊蚊,其雌性成蚊产的卵如果没有接触到水,也不会孵化、发育,这却被认为是一种静息状态(quiescence)。这两种现象的区别在于,发生滞育后,白纹伊蚊的卵即使收到合适的外界环境的刺激,仍需要经过一段时间的恢复才会孵化,而处于静息状态的埃及伊蚊的卵,只要受到适宜条件的刺激(如接触到水),就会马上进入发育阶段。国外有学者利用RNA-seq技术,对这两种现象进行分析,发现这两种现象在发育停止的阶段,分子水平上是很相近的,不同之处在于滞育现象的早期准备阶段和后期修复阶段,是其所独有的[38]。关于滞育现象的早期准备阶段,国外学者通过RNA-seq技术,比较滞育前阶段(pre-diapause)的白纹伊蚊胚胎与同时期非滞育的白纹伊蚊胚胎基因表达水平上的差异,发现前者在基因表达模式上有非常大的改变[39-40]。目前,已有学者归纳和总结出了一套利用RNA-seq技术研究白纹伊蚊滞育现象的方法[41],为今后更全面、彻底地认识白纹伊蚊以及其他媒介昆虫的滞育现象提供了坚实的基础。
利用RNA-seq技术,我们对白纹伊蚊不同发育时期(卵、幼虫、蛹、雄蚊、雌蚊)和感染登革病毒前后的转录组进行了分析。对比分析不同发育阶段特别是雌雄蚊的基因表达谱(图 1),我们找到了在胚胎早期对性别分化具有重要作用的候选基因和对雌蚊吸血传病相关的性别偏爱基因。对比分析登革病毒感染与否的白纹伊蚊转录组,我们发现了可能与蚊媒与病原相互作用有关的免疫分子(未发表结果)。目前,针对这些候选基因的进一步功能分析正在进行之中。另外,对白纹伊蚊抗药品系和敏感品系的RNA-seq对比分析也在进行中,这对于其抗药机制的阐明非常重要。
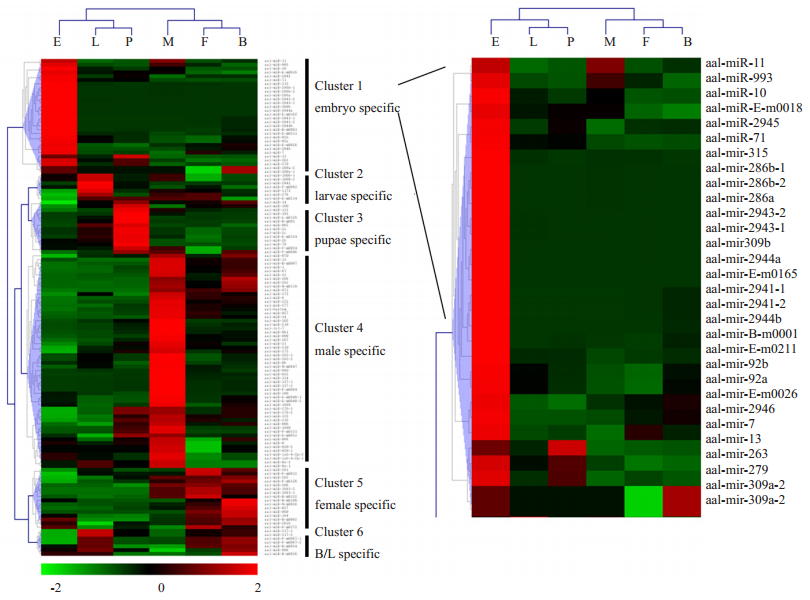
|
图 1 白纹伊蚊不同发育时期miRNA表达谱 Figure 1 The miRNA profiles of Aedes albopictus in different development stages. E: Egg; L: Larva; P: Pupa; M: Male; F: Female; B: Blood-fed female mosquito. cited by reference[51]. |
小RNA(small RNAs)主要指长度在18~30 nt的一类非编码RNA(ncRNAs),在真核生物中,具有基因表达调控功能的小RNA主要有微小RNA(microRNAs, miRNAs)、内源小干扰RNA(endo-siRNAs)和piwi干扰RNA(piRNAs)。piRNA长度集中在26-31nt, 目前只在动物的生殖系细胞及干细胞中被发现,其主要功能是参与转座子的沉默。miRNAs和endo-siRNAs长度主要集中在20~24 nt。miRNAs在动植物和微生物中都普遍存在,据估计一个物种中约1/3的基因会受到miRNA的调控,大量的实验也表明miRNAs参与了诸多生命过程的调控,例如细胞周期、细胞分化、组织器官的发生、营养代谢、信号途径以及对外界生物的非生物的环境的反应;同时,miRNAs在生产实践与临床治疗上也具有很大的应用前景。
以往用于寻找miRNAs等小RNA的方法有实验克隆法、计算机预测法。克隆法可以直接用于鉴定新小RNA,是初期发掘小RNA的常用方法,不足之处是实验周期较长,对低表达的小RNA的发现能力十分有限。计算机预测法多是针对某一已知的小RNA特征设计算法,从全基因组或EST数据库中快速发掘大量潜在的小RNA,一定程度上弥补了克隆法的缺点,然而,预测的小RNA最终还需要实验证明,同时计算机预测法对新类型小RNA的发掘能力十分有限。随着第二代高通量测序技术的问世,小RNA高通量测序(small RNA-Seq)技术开始逐渐取代原始的小RNA发掘法方法,该法具有速度快、成本低、覆盖度深等多方面的优点,对鉴定与发现生命体内的小分子RNA及其功能与机理研究起极大的推动作用。
全世界有超过3000种蚊虫,目前为止仅有冈比亚按蚊、斯氏按蚊、埃及伊蚊、致倦库蚊以及白纹伊蚊鉴定出miRNA[42]。一些miRNA的文库和功能分析表明miRNA对蚊虫的卵巢发育和吸血后的血液消化具有调节作用[43-47]。病毒感染可以对宿主细胞miRNA的表达水平产生深远影响,可能与宿主抗病毒机制及病毒入侵后改变细胞内环境有关,雌蚊中miRNA的表达模式会随着病原体的感染而发生变化[43-44, 48]。Hussain等对登革病毒(DENV)编码的miRNA或病毒小RNA(vsRNAs)的进行了功能研究,他们发现6个vsRNAs能通过作用于病毒基因组RNA茎环结构中的5'和3'的UTR区,显著增加病毒复制[49]。中肠屏障是蚊虫防止病原体入侵而建立的重要屏障,Alexander等的研究发现miR-1174仅在伊蚊和按蚊的中肠中表达,且雌蚊吸血后其表达量明显上调;而当miR-1174表达下调后,蚊子吸血率明显降低,寿命明显缩短。作者认为:蚊特异性miRNAs,特别是miR-1174具有重要的生物学意义,它们可能影响人们今后控制蚊虫的策略[50]。
我们对白纹伊蚊不同发育时期(卵、幼虫、蛹、雄蚊、雌蚊、吸血后雌蚊)的小RNA进行了深度测序分析。结果在白纹伊蚊中筛选出119条已知的miRNA基因,确定了15条novel miRNA基因,其中11条是伊蚊特异的,并且观察到许多miRNA呈现期特异表达的特点。经过实验验证,miR-286、miR-2492和miR-1891分别在白纹伊蚊的卵、幼虫和成虫期特异高效表达,敲低/敲除这些miRNA会对蚊虫的生长发育造成显著影响[49]。这些研究为新型生物杀虫剂的研发提供了靶标。我们还对感染登革病毒前后白纹伊蚊的细胞和成虫的小RNA进行了深度测序分析[51]。结果在感染登革病毒的白纹伊蚊中找到了10条表达上调的miRNA和11条表达下调的miRNA。通过对这些差显表达miRNA的功能分析,发现miR-252通过与E蛋白3'-UTR区域的结合,对登革病毒的复制起到抑制作用[48];而miR-281则通过与E蛋白5'-UTR区域的结合,对登革病毒的复制具有促进作用[49]。这些研究为抗登革病毒药物的设计和研发提供了线索。
piRNA来源于转座元件(transposable elements, TEs)、基因间隔区和一些编码蛋白质基因的3'UTRs,对维持基因的完整性和稳定性有一定作用,但最近的研究证明它在抗病毒免疫中也有较大作用[52]。Schnettler等的研究证明:对蚊虫细胞感染虫媒病毒可以引发piRNA路径,而敲除piRNA蛋白质会使病毒产生增多[53]。Castellano等确定了多个24-30 nt的Piwi相互作用RNAs基因组簇,通过比对到转座元件和蛋白质编码基因的3'UTRs,发现许多TEs和一些内源性基因的3'UTR产生大量具有piRNA样特征的29-nt小RNAs峰。此外,来自冈比亚按蚊和黑腹果蝇TEs的正义和反义piRNAs揭示了piRNA序列偏差的新特征[54]。弗吉尼亚理工大学的研究人员最近在库蚊中发现了一种新型的抗病毒途径,Morazzani等在无dicer-2和无突变的蚊细胞中进行的实验表明,病毒产生的piRNA样小RNA可以在病毒产生siRNA的过程中调节病毒感染的发生。同时也表明新的piRNA途径存在于蚊媒的体细胞中并且可能发挥着比siRNA途径更宽泛的的抗病毒作用,显示出其为强大的免疫系统。因此,理解病毒如何绕开蚊虫的双重抗病毒反应对于科学家来说是越来越有趣的挑战[55]。
4 结语随着新型测序技术和生物信息学分析技术的不断发展,生物医学研究也迎来了大数据分析时代。近年蚊虫组学的快速发展,给其媒介生物学、入侵扩散的机制研究等提供了广阔的、深远的大数据分析平台,也必将为蚊虫的媒介控制和传播疾病的防制带来更多的机遇和指引。
| [1] | Arensburger P, Megy K, Waterhouse RM, et al. Sequencing of culex quinquefasciatus establishes a platform for mosquito comparative genomics[J]. Science,2010, 330 (60) : 86-8. |
| [2] | Nene V, Wortman JR, Lawson D, et al. Genome sequence of Aedes aegypti, a major arbovirus vector[J]. Science,2007, 316 (5832) : 1718-23. DOI: 10.1126/science.1138878. |
| [3] | Holt RA, Subramanian GM, Halpern A, et al. The genome sequence of the malaria mosquito Anopheles gambiae[J]. Science,2002, 298 (5591) : 129-49. DOI: 10.1126/science.1076181. |
| [4] | Neafsey DE, Waterhouse RM, Abai MR, et al. Mosquito genomics. Highly evolvable malaria vectors: the genomes of 16 Anopheles mosquitoes[J]. Science,2015, 347 (6217) : 1258522. DOI: 10.1126/science.1258522. |
| [5] | Clark AG, Messer PW. Evolutionary genomics. Conundrum of jumbled mosquito genomes[J]. Science,2015, 347 (6217) : 27-8. DOI: 10.1126/science.aaa3600. |
| [6] | Jiang X, Peery A, Hall AB, et al. Genome analysis of a major urban malaria vector mosquito, Anopheles stephensi[J]. Genome Biol,2014, 15 (9) : 459. DOI: 10.1186/s13059-014-0459-2. |
| [7] | Marinotti O, Cerqueira GC, De Almeida LG, et al. The genome of Anopheles darlingi, the main neotropical malaria vector[J]. Nucleic Acids Res,2013, 41 (15) : 7387-400. DOI: 10.1093/nar/gkt484. |
| [8] | Severson DW, Behura SK. Mosquito genomics: progress and challenges[J]. Annu Rev Entomol,2012, 57 : 143-66. DOI: 10.1146/annurev-ento-120710-100651. |
| [9] | Deng Y, Yan H, Gu J, et al. Molecular and functional characterization of odorant-binding protein genes in an invasive vector mosquito, Aedes albopictus[J]. PLoS One,2013, 8 (7) : e68836. DOI: 10.1371/journal.pone.0068836. |
| [10] | Criscione F, Qi Y, Saunders R, et al. A unique Y gene in the Asian malaria mosquito Anopheles stephensi encodes a small lysine-rich protein and is transcribed at the onset of embryonic development[J]. Insect Mol Biol,2013, 22 (4) : 433-41. DOI: 10.1111/imb.2013.22.issue-4. |
| [11] | Hall AB, Qi Y, Timoshevskiy V, et al. Six novel Y chromosome genes in Anopheles mosquitoes discovered by independently sequencing males and females[J]. BMC Genomics,2013, 14 : 273. DOI: 10.1186/1471-2164-14-273. |
| [12] | Hall AB, Timoshevskiy VA, Sharakhova MV, et al. Insights into the preservation of the homomorphic sex-determining chromosome of Aedes aegypti from the discovery of a male-biased gene tightly linked to the M-locus[J]. Genome Biol Evol,2014, 6 (1) : 179-91. DOI: 10.1093/gbe/evu002. |
| [13] | Shukla JN, Nagaraju J. Doublesex: a conserved downstream gene controlled by diverse upstream regulators[J]. J Genet,2010, 89 (3) : 341-56. DOI: 10.1007/s12041-010-0046-6. |
| [14] | Von Philipsborn AC, Jörchel S, Tirian L, et al. Cellular and behavioral functions of fruitless isoforms in Drosophila courtship[J]. Curr Biol,2014, 24 (3) : 242-51. DOI: 10.1016/j.cub.2013.12.015. |
| [15] | Scali C, Catteruccia F, Li Q, et al. Identification of sex-specific transcripts of the Anopheles gambiae doublesex gene[J]. J Exp Biol,2005, 208 (Pt 19) : 3701-9. |
| [16] | 刘培文, 李小聪, 顾金保, 等. 冈比亚按蚊性别决定基因doublesex的克隆、序列分析及表达谱[J]. 昆虫学报,2015 (2) : 122-31. |
| [17] | Weterings R, Umponstira C, Buckley HL. Container-breeding mosquitoes and predator community dynamics along an urbanforest gradient: The effects of habitat type and isolation[J]. Basic Applied Ecol,2014, 15 (6) : 486-95. DOI: 10.1016/j.baae.2014.07.006. |
| [18] | Zhou X, Rinker DC, Pitts RJ, et al. Divergent and conserved elements comprise the chemoreceptive repertoire of the nonbloodfeeding mosquito Toxorhynchites amboinensis[J]. Genome Biol Evol,2014, 6 (10) : 2883-96. DOI: 10.1093/gbe/evu231. |
| [19] | Hodges TK, Cosme LV, Athrey G, et al. Species-specific chemosensory gene expression in the olfactory organs of the malaria vector Anopheles gambiae[J]. BMC Genomics,2014, 15 : 1089. DOI: 10.1186/1471-2164-15-1089. |
| [20] | Obiero GF, Mireji PO, Nyanjom SR, et al. Odorant and gustatory receptors in the tsetse fly glossina morsitans morsitans[J]. PLoS Negl Trop Dis,2014, 8 (4) : e2663. DOI: 10.1371/journal.pntd.0002663. |
| [21] | Tauxe GM, Macwilliam D, Boyle SM, et al. Targeting a dual detector of skin and CO2 to modify mosquito host seeking[J]. Cell,2013, 155 (6) : 1365-79. DOI: 10.1016/j.cell.2013.11.013. |
| [22] | Arcà B, Lombardo F, Francischetti IM, et al. An insight into the sialome of the adult female mosquito Aedes albopictus[J]. Insect Biochem Mol Biol,2007, 37 (2) : 107-27. DOI: 10.1016/j.ibmb.2006.10.007. |
| [23] | Das S, Radtke A, Choi YJ, et al. Transcriptomic and functional analysis of the Anopheles gambiae salivary gland in relation to blood feeding[J]. BMC Genomics,2010, 11 : 566. DOI: 10.1186/1471-2164-11-566. |
| [24] | Calvo E, Pham VM, Lombardo F, et al. The sialotranscriptome of adult male Anopheles gambiae mosquitoes[J]. Insect Biochem Mol Biol,2006, 36 (7) : 570-5. DOI: 10.1016/j.ibmb.2006.04.005. |
| [25] | Dixit R, Rawat M, Kumar S, et al. Salivary gland transcriptome analysis in response to sugar feeding in malaria vector Anopheles stephensi[J]. J Insect Physiol,2011, 57 (10) : 1399-406. DOI: 10.1016/j.jinsphys.2011.07.007. |
| [26] | Valenzuela JG, Francischetti IM, Pham VM, et al. Exploring the salivary gland transcriptome and proteome of the Anopheles stephensi mosquito[J]. Insect Biochem Mol Biol,2003, 33 (7) : 717-32. DOI: 10.1016/S0965-1748(03)00067-5. |
| [27] | Calvo E, Andersen J, Francischetti IM, et al. The transcriptome of adult female Anopheles darlingi salivary glands[J]. Insect Mol Biol,2004, 13 (1) : 73-88. DOI: 10.1111/imb.2004.13.issue-1. |
| [28] | Almeras L, Fontaine A, Belghazi M, et al. Salivary gland protein repertoire from Aedes aegypti mosquitoes[J]. Vector Borne Zoonotic Dis,2010, 10 (4) : 391-402. DOI: 10.1089/vbz.2009.0042. |
| [29] | Valenzuela JG, Pham VM, Garfield MK, et al. Toward a description of the sialome of the adult female mosquito Aedes aegypti[J]. Insect Biochem Mol Biol,2002, 32 (9) : 1101-22. DOI: 10.1016/S0965-1748(02)00047-4. |
| [30] | Calvo E, Ribeiro JM. A novel secreted endonuclease from Culex quinquefasciatus salivary glands[J]. J Exp Biol,2006, 209 (Pt 14) : 2651-9. |
| [31] | Ribeiro JC, Charlab R, Pham VM, et al. An insight into the salivary transcriptome and proteome of the adult female mosquito Culex pipiens quinquefasciatus[J]. Insect Biochem Mol Biol,2004, 34 (6) : 543-63. DOI: 10.1016/j.ibmb.2004.02.008. |
| [32] | Calvo E, Dao A, Pham VM, et al. An insight into the sialome of anopheles funestus reveals an emerging pattern in anopheline salivary protein families[J]. Insect Biochem Mol Biol,2007, 37 (2) : 164-75. DOI: 10.1016/j.ibmb.2006.11.005. |
| [33] | Chagas AC, Calvo E, Pimenta PF, et al. An insight into the sialome of Simulium guianense (DIPTERA: SIMulIIDAE), the main vector of river blindness disease in brazil[J]. BMC Genomics,2011, 12 : 612. DOI: 10.1186/1471-2164-12-612. |
| [34] | Andersen JF, Pham VM, Meng Z, et al. Insight into the sialome of the black fly, simulium vittatum[J]. J Proteome Res,2009, 8 (3) : 1474-88. DOI: 10.1021/pr8008429. |
| [35] | Denlinger DL, Armbruster PA. Mosquito diapause[J]. Annu Rev Entomol,2014, 59 (12) : 73-93. |
| [36] | Poelchau MF, Reynolds JA, Denlinger DL, et al. Transcriptome sequencing as a platform to elucidate molecular components of the diapause response in the asian tiger mosquito, Aedes albopictus[J]. Physiol Entomol,2013, 38 (2) : 173-81. DOI: 10.1111/phen.12016. |
| [37] | Urbanski J, Mogi M, O'donnell D, et al. Rapid adaptive evolution of photoperiodic response during invasion and range expansion across a climatic gradient[J]. Am Nat,2012, 179 (4) : 490-500. DOI: 10.1086/664709. |
| [38] | Poelchau MF, Reynolds JA, Elsik CG, et al. RNA-Seq reveals early distinctions and late convergence of gene expression between diapause and quiescence in the Asian tiger mosquito, Aedes albopictus[J]. J Exp Biol,2013, 216 (Pt 21) : 4082-90. |
| [39] | Poelchau MF, Reynolds JA, Elsik CG, et al. Deep sequencing reveals complex mechanisms of diapause preparation in the invasive mosquito, Aedes albopictus[J]. Proc Biol Sci,2013, 280 (1759) : 20130143. DOI: 10.1098/rspb.2013.0143. |
| [40] | Poelchau MF, Reynolds JA, Denlinger DL, et al. A de novo transcriptome of the Asian tiger mosquito, Aedes albopictus, to identify candidate transcripts for diapause preparation[J]. BMC Genomics,2011, 12 : 619. DOI: 10.1186/1471-2164-12-619. |
| [41] | Poelchau MF, Huang X, Goff A, et al. An experimental and bioinformatics protocol for RNA-seq analyses of photoperiodic diapause in the Asian tiger mosquito, Aedes albopictus[J]. J Vis Exp,2014 (93) : e51961. |
| [42] | 周艳河, 言慧, 刘艳霞, 等. 虫媒病毒及其媒介蚊虫microRNA的研究现状和展望[J]. 分子诊断与治疗杂志,2011, 3 (02) : 134-7. |
| [43] | Winter F, Edaye S, Hüttenhofer A, et al. Anopheles gambiae miRNAs as actors of defence reaction against Plasmodium invasion[J]. Nucleic Acids Res,2007, 35 (20) : 6953-62. DOI: 10.1093/nar/gkm686. |
| [44] | Mead EA, Tu Z. Cloning, characterization, and expression of microRNAs from the Asian malaria mosquito, Anopheles stephensi[J]. BMC Genomics,2008, 9 : 244. DOI: 10.1186/1471-2164-9-244. |
| [45] | Li S, Mead EA, Liang S, et al. Direct sequencing and expression analysis of a large number of miRNAs in Aedes aegypti and a multi-species survey of novel mosquito miRNAs[J]. BMC Genomics,2009, 10 : 581. DOI: 10.1186/1471-2164-10-581. |
| [46] | Bryant B, Macdonald W, Raikhel AS. microRNA miR-275 is indispensable for blood digestion and egg development in the mosquito Aedes aegypti[J]. Proc Natl Acad Sci USA,2010, 107 (52) : 22391-8. DOI: 10.1073/pnas.1016230107. |
| [47] | Hussain M, Walker T, O'neill SL, et al. Blood meal induced microRNA regulates development and immune associated genes in the Dengue mosquito vector, Aedes aegypti[J]. Insect Biochem Mol Biol,2013, 43 (2) : 146-52. DOI: 10.1016/j.ibmb.2012.11.005. |
| [48] | Skalsky RL, Vanlandingham DL, Scholle F, et al. Identification of microRNAs expressed in two mosquito vectors, Aedes albopictus and Culex quinquefasciatus[J]. BMC Genomics,2010, 11 : 119. DOI: 10.1186/1471-2164-11-119. |
| [49] | Hussain M, Asgari S. MicroRNA-like viral small RNA from Dengue virus 2 autoregulates its replication in mosquito cells[J]. Proc Natl Acad Sci USA,2014, 111 (7) : 2746-51. DOI: 10.1073/pnas.1320123111. |
| [50] | Liu S, Lucas KJ, Roy S, et al. Mosquito-specific microRNA-1174 targets serine hydroxymethyltransferase to control key functions in the gut[J]. Proc Natl Acad Sci USA,2014, 111 (40) : 14460-5. DOI: 10.1073/pnas.1416278111. |
| [51] | Liu Y, Zhou Y, Wu J, et al. The expression profile of Aedes albopictus miRNAs is altered by dengue virus serotype-2 infection[J]. Cell Biosci,2015, 5 (16) : 16. |
| [52] | Lucas KJ, Myles KM, Raikhel AS. Small RNAs: a new frontier in mosquito biology[J]. Trends Parasitol,2013, 29 (6) : 295-303. DOI: 10.1016/j.pt.2013.04.003. |
| [53] | Schnettler E, Donald CL, Human S, et al. Knockdown of piRNA pathway proteins results in enhanced Semliki Forest virus production in mosquito cells[J]. J Gen Virol,2013, 94 (Pt 7) : 1680-9. |
| [54] | Castellano L, Rizzi E, Krell J, et al. The germline of the malaria mosquito produces abundant miRNAs, endo-siRNAs, piRNAs and 29-nt small RNAs[J]. BMC Genomics,2015, 16 : 100. DOI: 10.1186/s12864-015-1257-2. |
| [55] | Morazzani EM, Wiley MR, Murreddu MG, et al. Production of virus-derived ping-pong-dependent piRNA-like small RNAs in the mosquito soma[J]. PLoS Pathog,2012, 8 (1) : e1002470. DOI: 10.1371/journal.ppat.1002470. |
 2015, Vol. 35
2015, Vol. 35
