抗体是指机体的获得性免疫系统在抗原刺激下,由B淋巴细胞经历产生、成熟、增殖、分化并分泌的可与相应抗原发生特异性结合的一类免疫球蛋白[1]。抗体是由两条完全相同的重链和两条完全相同的轻链由二硫键和非共价键结合[2]形成的“Y”形对称结构的复合物[3]。
根据理化性质和生物学功能不同,抗体分为IgG、IgM、IgA、IgD、IgE五类(表 1)。IgG抗体占血清Ig总量的80%,分为IgG1、IgG2、IgG3、IgG4四个亚类,是机体抗感染的“主力军”[4-5]。IgG在血清中持续的时间长,半衰期为20~23 d,是唯一能在母亲妊娠期穿过胎盘的抗体。IgM占血清Ig总量的5%~10%,是免疫应答中最先分泌的抗体,是机体抗感染的“先头部队”,可用于感染的早期诊断[4-5]。IgA抗体占血清Ig总量的10%~15%,分为单体型IgA和分泌型IgA。分泌型IgA主要分布于呼吸、消化、生殖等管道的粘膜表面和乳汁、唾液、泪液中,可中和感染因子,参与局部粘膜免疫,是机体抗感染的“边防军”[4-5]。IgD抗体占血清Ig总量的0.2%,主要出现在成熟的B淋巴细胞表面上,与B细胞的分化相关[4]。IgE抗体是血清中含量最少的抗体,约占血清Ig总量的0.003%,主要由粘膜下淋巴组织中的浆细胞分泌,参与超敏反应,与机体抗寄生虫免疫相关[4-5]。
| 表 1 人抗体的主要理化性质和生物学功能 Table 1 Physicochemical property and biological function of human antibodies |
抗体是B淋巴细胞抗原受体(B cell antigen receptor, BCR)的分泌形式,抗体的产生和成熟过程也就是B细胞的产生、发育、增殖和分化的过程[2, 3, 5]。其重链由V、D、J、C四个基因片段编码的,轻链由V、J、C三个基因片段编码。成熟的B细胞从骨髓释放到外周血中,经过特异性抗原的刺激后会进入淋巴结的生发中心,在生发中心中经过细胞增殖、体细胞超突变、亲和力成熟、抗原受体编辑、种型转化等复杂事件,分化成能分泌特异性抗体的浆细胞和少数的记忆性B细胞[2, 3, 5]。经过生发中心的体细胞超突变和亲和力成熟过程,进一步增加了抗体谱(Antibody repertoire)的多样性,使抗体的数量达到了1013的天文数字,并提高了抗体对特异性抗原的亲和力[2, 5]。
抗体是获得性免疫的主要组分,在防御性及致病性的免疫反应中起关键的作用。通过对抗体发生、发展和成熟过程的解析,促进了对传染性疾病病理机制、疫苗设计、自免疫疾病治疗等免疫学相关的研究。例如,通过对HIV-1缓慢进展患者体内中和广谱抗体产生及成熟通路的解析,以获得具有高效中和效应的HIV-1特异抗体,并开始应用于HIV-1疫苗的研发[3, 6-10]。在类风湿性关节炎自免疫疾病中,自身抗体基因IGHV4-34明显富集,可以作为类风湿性关节炎检测的分子标记和免疫治疗的靶点[11]。通过对抗体发生、发展的深入及全面解析,不仅能增加我们对免疫调节的认识,而且为疾病的治疗和疫苗的研发等临床应用提供新的技术与方案,引领新型治疗性抗体研究的时代。
低通量测序在抗体研究中的应用随着测序技术的发展,抗体组学的研究经历了从低通量到高通量的时代变革。在1990~2000年时,主要利用RT-PCR扩增和Sanger测序技术研究少量B细胞的抗体特征[12]。应用这一技术,人们开展了致病病毒如HIV-1、流感病毒的单克隆抗体和自免疫疾病相关自身抗体的研究。通过抗体测序结合克隆表达,人们获得了第一代HIV-1的广谱抗体如b12、4E10、2G12等抗体[13-17],解析了它们的抗原识别位点,为HIV-1疫苗的研发提供了指导[18-19]。运用这一技术,Wardemann等[20]阐明骨髓中产生自身抗体及多效抗体的B细胞至少经过2步独立的自体耐受性的检测,当这一过程发生异常时会导致产生分泌自身抗体的浆细胞,诱发自身免疫疾病如系统性红斑狼疮和风湿性关节炎[21]。
由于技术的低通量性,每次实验只能分析几十到几百个B细胞的抗体特性,与抗体谱的巨大数据相比,低通量技术只为我们揭示了冰山的一角。
高通量测序在抗体组学中的应用对免疫系统的认识及对抗体的挖掘及应用需要人们能全面深入的了解抗体谱的整体特征,因通量限制,这个目标是传统技术无法实现的。罗氏454和Illumina Miseq为代表的下一代测序技术的应用,一次测序即可获得数以百万计的B细胞的抗体可变区域序列。结合分子标记与细胞标记技术,NGS为人们展示了更广阔的抗体谱特性,数据量更加接近人体抗体谱真实数量,使抗体的研究进入了大数据的时代。高通量数据的捕获也使得研究抗体家系及抗体的成熟途径成为现实。以下将从实验的操作、测序平台的选择、数据的分析三个方面详细介绍高通量测序在抗体组学中的进展与应用(图 1)。
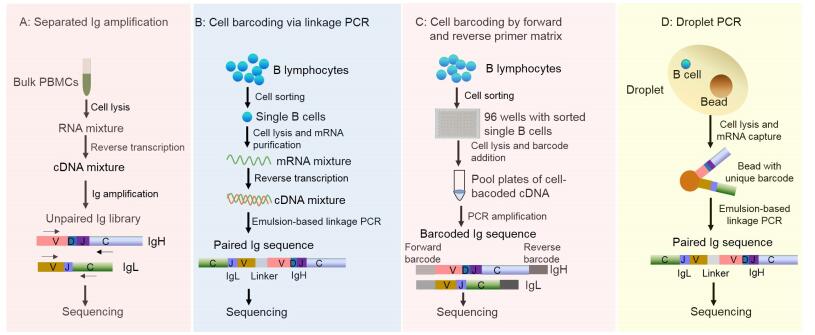
|
图 1 抗体高通量测序技术流程(修订自Robinson, 2015)[12] Figure 1 Technical workflow for ultra-high throughput antibody repertoire sequencing (revised from Robinson, 2015).A:Schematic of the library preparation for deep sequencing using 454-pyrosequencing.mRNA was isolated from PBMCs and reverse transcribed. Individual PCR reactions were performed for the IGH and IGL using family-specific mix primers; B:Cell barcoding via linkage PCR.Single B cells are isolated and lysed, then their RNA is captured by poly-T beads.cDNAs of the IgH and IgL expressed by individual B cells are then linked by emulsion to RT-PCR, and then pooled and sequenced; C:Cell barcoding by forward and reverse primer matrix.Single B cells are sorted, then V-gene forward primers and C-region reverse primers are used to add cell-specific barcodes to and amplify by PCR, IgH and IgL cDNA generated from an individual B cell.Single-barcoded immunoglobulin genes are then pooled and sequenced; D:Microfluidic combination of beads with unique barcodes and single B cells into individual droplets.Using microfluidics, single B cells and beads with unique barcodes are combined in individual droplets, followed by lysis of the B cell, PCR and sequencing. |
2009年Weinstein等[22]首次应用高通量测序技术全面研究了斑马鱼的抗体库特征,揭示不同斑马鱼个体间重链VDJ基因的使用频率存在相似性,促进了高通量测序技术在人抗体组分析中的应用。抗体的高通量测序需要考虑实验设计、重链与轻链配对、数据的分析、挖掘及可视化等问题。近5年高通量测序技术在抗体组学的应用可以分为2个时期:第一个时期为2009~2013年,利用抗体可变区与恒定区的混合引物,通过巢式PCR从外周血单个核细胞cDNA中分别扩增抗体重链与轻链基因,然后通过罗氏454测序平台测序(图 1A)。应用这一技术从HIV-1患者中分离出了中和力更强的第二代广谱中和抗体,如VRC01、VRC03、CH103-CH106、CH01-CH04、10E8等。结合结构生物学研究者解析出了HIV-1病毒4个重要的保守表位结构域,分别是CD4结合表位、V1/V2区、V3区、gp41蛋白近膜外周区[23-28]。对这些抗体成熟通路的解析及HIV-1保守表位的分析,可以指导HIV-1疫苗的设计和生产,从而为预防HIV-1提供了新的思路。尽管这些方法发展迅速,但仍存在诸多问题:如混合B细胞样本增加了特异性增殖B细胞克隆分析的难度;设计在V区的正向引物并不能覆盖所有的抗体基因片段,多个正向引物的混合增加了PCR的偏好性;尽管可以从混合的mRNA样本中同时测出抗体的重链与轻链,但失去了体内重链与轻链的配对信息;454测序平台容易引入碱基缺失与插入,增加了后续分析的难度。
针对以上问题,新的技术被开发并成功应用到抗体测序中(图 1B-D)。DeKosky等[29]通过物理方法将单个B细胞中的重链与轻链基因连结在一起,实现了内源重链与轻链的配对测序(图 1B)。Robinson[30-32]实验室通过细胞标记的方法,在反转录过程中对不同的B细胞cDNA产物5`端引入特异性的标记,有利于纠正PCR过程及测序过程中的错误,并保证了内源重链与轻链的配对(图 1C)。最近Georgiou[33]实验室开发了一套新的低花费、单细胞的油滴PCR技术,既可以保持体内重链与轻链的配对又实现了高通量,每次实验可以分析多达2×106个B细胞,提高了测序的广度与深度度(图 1D)。5'RACE技术的应用,解决了混合引物扩增的偏好性问题,能够更全面的扩增到抗体基因,结合PGM与Illumina MiSeq平台提高了读段的长度、测序的深度与数据量,更利于抗体组的高通量分析[34]。因此,单细胞测序、重链与轻链配对、5'RACE、PGM与Illumina MiSeq平台应用,将进一步推动抗体组学在抗体生产、自免疫疾病诊断、疫苗生产及癌症相关疾病的研究。
抗体组学的生物信息分析如何从数据的海洋中挖掘出有用的信息,用于指导抗体的生产和疫苗的设计,是抗体组学工作中面临的巨大挑战。目前抗体分析中常用的软件及工具如图 2所示。其中包括的主要步骤如下:对测序得到的原始数据(Raw reads)进行质量控制,滤掉质量低与长度短的读段并获得干净的数据(Clean reads);利用IgBlast和IMGT对抗体的可变区进行V(D)J生殖系基因定位(VDJ annotation)[35-36];根据生殖系基因定位将每个合并的读段剪切至恰好包含整个可变区域;去除冗余数据,对于每个唯一读段(Unique reads)记录其总数量;系统化注释获得reads的可变区域并提取CDR3区域;分析CDR3区域并获得抗体的家系信息(Clone or lineage);获得每个家系中轻重链可变区域的序列特性,并利用重链和轻链各家系的reads数量进行轻重链配对;对配对好的抗体序列通过实验验证功能。
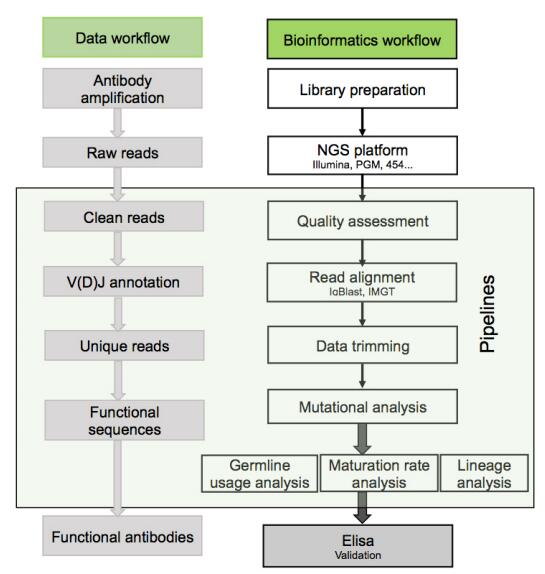
|
图 2 抗体组分析的基本流程 Figure 2 Basic workflow for antibody repertoire sequencing projects. |
利用上述分析软件与流程,人们开展了大量的抗体相关工作的分析,促进了数据的分析与挖掘并加快了免疫学的进展。例如我们课题组最近参与的HIV-1中和抗体的研究,通过连续15年样本的大数据量的分析,解析了HIV-1广谱中和抗体VRC01的成熟与变异速率,结合结构生物学的分析,揭示了VRC01抗体的进化机制,对开发HIV-1疫苗具有重大的意义[37]。同时通过对免疫反应中抗体谱数据特征的分析,揭示了年龄、遗传等因素对抗体产生与成熟的影响,为开发相应的针对不同人群的有效疫苗提供了指导[38-39]。然而,相对于高通量数据的快速发展,目前还没有一个统一的分析流程,所有的分析软件主要是借助了一些免费的公用软件,针对抗体分析的专一性软件还没有开发出来。现有的分析流程也存在一些问题,如:IgBlast和IMGT对抗体序列进行比对时,不能纠正序列中的错误,导致比对的不准确性;由于D基因较短,IgBlast和IMGT不能准确地定位D基因;抗体成熟过程中AID酶会引入新的突变,增加了抗体的多样性并提高了抗体的亲和力,然而目前的分析不能区分出AID所介导的突变热点;家系进化树的构建完全依赖于序列相似性,没有考虑原始的生殖基因及AID突变的偏好(Hotspot),会遗漏掉许多基因并使得进化树不能完全反映细胞内重链与轻链的配对情况。所有这些问题是需要下一步抗体数据分析中得到解决。
展望从以上对抗体组学的发展及应用简述中可以看到,结合高通量测序的抗体组学具有效率高、深度大、基因多样性更丰富、数据反映更准确等优势。最近5年里,高通量测序的抗体组学分析已经成为免疫学的热门方法,促进了包括抗体发生、疫苗研发、肿瘤发生及自免疫疾病等相关免疫学的研究并取得了众多创新性的成果。但到目前为止,抗体组学分析的应用潜力还远未发挥出来,其技术仍受到一些现实条件的约束,如高通量PCR抗体基因扩增技术仍不够完善、测序读段长度与准确性还有待进一步提高、标准的分析流程有待开发完善。随着单细胞分选及测序的应用、PCR扩增基因方法的改善、细胞标记的应用、分析标准的开发,未来抗体组学将在诊断、药理学、疫苗设计及临床应用中发挥前所未有的重大作用,引领新型治疗性抗体研究的时代。
| [1] | Dorner T, Radbruch A. Antibodies and B cell memory in viral immunity[J]. Immunity,2007, 27 : 384-92. DOI: 10.1016/j.immuni.2007.09.002. |
| [2] | Murphy K. Janeway, s immunobiology[M]. 2012: 127-56. |
| [3] | Corti D, Lanzavecchia A. Broadly neutralizing antiviral antibodies[J]. Annu Rev Immunol,2013, 31 : 705-42. DOI: 10.1146/annurev-immunol-032712-095916. |
| [4] | Lavinder JJ, Wine Y, Giesecke C, et al. Identification and characterization of the constituent human serum antibodies elicited by vaccination[J]. PNAS,2014, 111 (6) : 2259-64. DOI: 10.1073/pnas.1317793111. |
| [5] | Male D, Brostoff J, Roth DB, et al. Immnuology[M]. 2012: 51-70. |
| [6] | Wu XL, Yang ZY, Li YX, et al. Rational design of envelope identifies broadly neutralizing human monoclonal antibodies to HIV-1[J]. Science,2010, 329 : 856-61. DOI: 10.1126/science.1187659. |
| [7] | Wu XL, Zhou TQ, Zhu J, et al. Focused evolution of HIV-1 neutralizing antibodies revealed by structures and deep sequencing[J]. Science,2011, 333 : 1593-602. DOI: 10.1126/science.1207532. |
| [8] | Liao HX, Lynch R, Zhou TQ, et al. Co-evolution of a broadly neutralizing HIV-1 antibody and founder virus[J]. Nature,2013, 496 (7446) : 469-76. DOI: 10.1038/nature12053. |
| [9] | Gao F, Bonsignori M, Liao HX, et al. Cooperation of B cell lineages in induction of HIV-1-broadly neutralizing antibodies[J]. Cell,2014, 158 : 481-91. DOI: 10.1016/j.cell.2014.06.022. |
| [10] | Mouquet H. Antibody B cell responses in HIV-1 infection[J]. Trends Immnuol,2014, 35 (11) : 549-61. DOI: 10.1016/j.it.2014.08.007. |
| [11] | Doorenspleet ME, Klarenbeek PL, de Hair MJ, et al. Rheumatoid arthritis synovial tissue harbours dominant B-cell and plasma-cell clones associated with autoreactivity[J]. Ann Rheum Dis,2014, 73 (4) : 756-62. DOI: 10.1136/annrheumdis-2012-202861. |
| [12] | Robinson WH. Sequencing the functional antibody repertoirediagnostic and therapeutic discovery[J]. Nat Rev Rheumatol,2015, 11 (3) : 171-82. |
| [13] | Gorny MK, Xu JY, Karwowska S, et al. Repertoire of neutralizing human monoclonal antibodies specific for the V3 domain of HIV-1 gp120[J]. J Immunol,1993, 150 (2) : 635-43. |
| [14] | Thali M, Moore JP, Furman C, et al. Characterization of conserved human immunodeficiency virus type 1 gp120 neutralization epitopes exposed upon gp120-CD4 binding[J]. J Virol,1993, 67 (7) : 3978-88. |
| [15] | Burton DR, Barbas 3rd CF, Persson MA, et al. A large array of human monoclonal antibodies to type 1 human immunodeficiency virus from combinatorial libraries of asymptomatic seropositive individuals[J]. PNAS,1991, 88 (22) : 10134-7. DOI: 10.1073/pnas.88.22.10134. |
| [16] | Trkola A, Purtscher M, Muster T, et al. Human monoclonal antibody 2G12 defines a distinctive neutralization epitope on the gp120 glycoprotein of human immunodeficiency virus type 1[J]. J Virol,1996, 70 (2) : 1100-8. |
| [17] | Buchacher A, Predl R, Strutzenberger K, et al. Generation of human monoclonal antibodies against HIV-1 proteins; electrofusion and Epstein-Barr virus transformation for peripheral blood lymphocyte immortalization[J]. AIDS Res Hum Retroviruses,1994, 10 (4) : 359-69. DOI: 10.1089/aid.1994.10.359. |
| [18] | Burton DR, Ahmed R, Barouch DH, et al. A Blueprint for HIV vaccine discovery[J]. Cell Host Microbe,2012, 12 (4) : 396-407. DOI: 10.1016/j.chom.2012.09.008. |
| [19] | Haynes BF, Kelsoe G, Harrison SC, et al. B-cell-lineage immunogen design in vaccine development with HIV-1 as a case study[J]. Nat Biotechnol,2012, 30 (5) : 423-33. DOI: 10.1038/nbt.2197. |
| [20] | Wardemann H, Yurasov S, Schaefer A, et al. Predominant autoantibody production by early human B cell precursors[J]. Science,2003, 301 (5638) : 1374-7. DOI: 10.1126/science.1086907. |
| [21] | Meffre E, Wardemann H. B-cell tolerance checkpoints in health and autoimmunity[J]. Curr Opin Immunol,2008, 20 : 632-8. DOI: 10.1016/j.coi.2008.09.001. |
| [22] | Weinstein JA, Jiang N, White Ⅲ RA, et al. High-throughput sequencing of the Zebrafish antibody repertoire[J]. Science,2009, 324 (5928) : 807-10. DOI: 10.1126/science.1170020. |
| [23] | Scheid JF, Mouquet H, Ueberheide B, et al. Sequence and structural convergence of broad and potent HIV antibodies that mimic CD4 binding[J]. Science,2011, 333 (6049) : 1633-7. DOI: 10.1126/science.1207227. |
| [24] | Bonsignori M, Wiehe K, Grimm SK, et al. An autoreactive antibody from an SLE/HIV-1 individual broadly neutralizes HIV-1[J]. J Clin Invest,2014, 124 (4) : 1835-43. DOI: 10.1172/JCI73441. |
| [25] | Bonsignori M, Hwang KK, Chen X, et al. Analysis of a clonal lineage of HIV-1 envelope V2/V3 conformational epitope-specific broadly neutralizing antibodies and their inferred unmutated common ancestors[J]. J Virol,2011, 85 (19) : 9998-10009. DOI: 10.1128/JVI.05045-11. |
| [26] | Liao HX, Chen X, Munshaw S, et al. Initial antibodies binding to HIV-1 gp41 in acutely infected subjects are polyreactive and highly mutated[J]. J Exp,2011, 208 (11) : 2237-49. DOI: 10.1084/jem.20110363. |
| [27] | Doria-Rose NA, Schramm CA, Gorman J, et al. Developmental pathway for potent V1V2-directed HIV-neutralizing antibodies[J]. Nature,2014, 509 (7498) : 55-62. DOI: 10.1038/nature13036. |
| [28] | Zhu J, òDell S, Ofek G, et al. Somatic populations of PGT135-137 HIV-1-neutralizing antibodies identified by 454 pyrosequencing and bioinformatics[J]. Front Microbiol,2012, 3 : 315. |
| [29] | DeKosky BJ, Ippolito GC, Deschner RP, et al. High-throughput sequencing of the paired human immunoglobulin heavy and light chain repertoire[J]. Nat Biotech,2013, 31 (2) : 166-9. DOI: 10.1038/nbt.2492. |
| [30] | Tan YC, Konqpachith S, Blum LK, et al. Barcode-enabled sequencing of plasmablast antibody repertoires in rheumatoid arthritis[J]. Arthritis Rheumatol,2014, 66 (10) : 2706-15. DOI: 10.1002/art.v66.10. |
| [31] | Tan YC, Blum LK, Konqpachith S, et al. High-throughput sequencing of natively paired antibody chains provides evidence for original antigenic sin shaping the antibody response to influenza vaccination[J]. Clin Immunol,2014, 151 (1) : 55-65. DOI: 10.1016/j.clim.2013.12.008. |
| [32] | Lu DR, Tan YC, Konqpachith S, et al. Identification of functional anti-Staphylococcus aureus antibodies by sequencing patient plasmablast antibody repertoires[J]. Clin Immunol,2014, 152 (1-2) : 77-89. DOI: 10.1016/j.clim.2014.02.010. |
| [33] | DeKosky BJ, Kojima T, Rodin A, et al. In-depth determination and analysis of the human paired heavy-and light-chain antibody repertoire[J]. Nat Med,2015, 21 (1) : 86-91. |
| [34] | He LL, Sok D, Azadnia P, et al. Toward a more accurate view of human B-cell repertoire by next-generation sequencing, unbiased repertoire capture and single-molecule barcoding[J]. Sci Rep,2014, 4 : 6778. DOI: 10.1038/srep06778. |
| [35] | Brochet X, Lefranc MP, Giudicelli V. IMGT/V-QUEST: the highly customized and integrated system for IG and TR standardized V-J and V-D-J sequence analysis[J]. Nucleic Acids Res,2008, 36 : W503-8. DOI: 10.1093/nar/gkn316. |
| [36] | Ye J, Ma N, Madden TL, et al. IgBlast: an immunoglobulin variable domain sequence analysis tool[J]. Nucleic Acids Res,2013, 41 : W34-40. DOI: 10.1093/nar/gkt382. |
| [37] | Wu XL, Zhang ZH, Schramm CA, et al. Maturation and diversity of the VRC01-antibody lineage over 15 years of chronic HIV-1 infection[J]. Cell, 2015(In press). |
| [38] | Wu YC B, Kipling D, Dunn-Walters DK. Age-related changes in human peripheral blood IGH repertoire following vaccination[J]. Frontiers in Immunology,2012, 3 (193) : 1-12. |
| [39] | Jiang N, He JK, Weinstein JA, et al. Lineage structure of the human antibody repertoire in response to influenza vaccination[J]. Sci Transl Med,2013, 5 (171) : 171-19. |
 2015, Vol. 35
2015, Vol. 35
