2. 西安交通大学 环境与疾病相关基因教育部重点实验室,陕西 西安 710061 ;
3. 西安交通大学 2010级临床硕士,陕西 西安 710061
2. Key Laboratory of Environment and Genes Related to Diseases of Ministry of Education, Xi'an Jiaotong University Health Science Center, Xi'an 710061, China ;
3. Clinical Master of Grade 2010, Xi'an Jiaotong University Health Science Center, Xi'an 710061, China
Toll样受体(toll-like receptors, TLRs)广泛表达于天然免疫系统,是一类Ⅰ型跨膜糖蛋白,由胞外区、跨膜区和胞质区组成。作为一类重要的模式识别受体,Toll样受体可以识别一种或多种特定的微生物病原体及其产物共有的高度保守的分子结构,即病原相关分子模式(PAMP),启动针对病原体的早期应答,诱发获得性免疫反应[1]。由于TLRs在识别细菌等病原微生物及介导的炎症反应信号转导中发挥着重要作用,是目前研究的热点。多项研究证实TLRs介导的信号与炎症和免疫密切相关[2-3]。TLRs识别配体后,可传递给细胞内的接头分子髓样分化因子88(myeloid differentiation factor 88, MyD88)、MyD88接头样蛋白(MyD88 adaptor-like protein, Mal)、Toll受体相关的干扰素活化剂(Toll-receptor-associated activator of interferon, TRIF)和TRIF相关接头分子(TRIF-related adaptor molecule, TRAM)[1]。这些分子可以通过激活炎症的主要调节物NF-кB、MAPKs和IFN-β,引起多种细胞因子的释放,上调抗原提呈细胞(APCs)表面CD80(B7-1)、CD86(B7-2)等共刺激分子,并最终激活特异性免疫系统,保护细胞[3]。在我们前期的研究中发现TLR2可能通过肝巨噬细胞参与肝脏炎症的发生,但其具体机制不明。
近几年越来越多的证据表明一些microRNA(miRNAs)可能通过调节TLRs以及TLR信号通路上关键分子如MyD88、NFκB等的表达,从而影响疾病的发生。miRNA是一个约22nt的非编码RNA,与其靶基因mRNA 3'UTR区结合位点完全或部分互补配对结合,通过mRNA降解或压抑翻译过程而抑制靶基因的表达[4-6],在细胞增殖与分化[7-8]、细胞凋亡与死亡[9]、发育[10]、代谢[6, 11-12]、免疫反应[13-14]、肿瘤发生[9, 13]等生物过程中担任了非常重要的作用。我们推测可能有miRNAs的改变调节了巨噬细胞TLR2的表达。生物信息学预测显示大鼠TLR2可能是miR-144的靶基因。为了证实此机制,我们用不同浓度的miR144的模拟物和抑制剂处理大鼠巨噬细胞系,明确其对TLR2及其下游分子TNF-α的作用,并构建了在含miR-144野生及突变结合位点的TLR2 mRNA 3'UTR区报告基因载体,通过其3'UTR区报告基因进一步明确miR-144与大鼠TLR2 mRNA 3'-UTR的靶向关系。
1 材料与方法 1.1 实验动物、主要试剂及仪器本实验选用4周龄近交系雄性E3大鼠2只,来自西安交通大学医学部生物化学与分子生物学系的SPF级实验动物中心。主要试剂:miR-144的mimics和inhibitor(上海吉玛),pmirGLO Dual-Luciferase miRNA Target Expression Vector(简记为pmirGLO载体)及双荧光素酶报告基因检测试剂盒(Dual Luciferase Reporter Gene Assay Kit)(Promega),TRIzol®试剂及Lipofactamine 2000(Invitrogen),反转录试剂盒RevertaidTM First Strand cDNA Synthesis(Fermentas),microRNA反转录试剂盒PrimeScript® miRNA cDNA Synthesis kit,SYBR® PrimeScriptTM RTPCR Kit II,Taq DNA聚合酶,SacⅠ、XbaⅠ限制性酶切酶及T4 DNA连接酶(TaKaRa),E.Z.N.A.® Fastfilter Endo-free Plasmid Mini Kit(OMEGA),DNA琼脂糖凝胶纯化试剂盒(博大泰克),脂质体TM2000(Invitrogen),F-12K(Sigma-Aldrich)及低糖DMEM(含1 g/L葡萄糖)液体培养基及胎牛血清(Hyclone)。其余试剂均按照分子克隆实验指南(第3版)的实验方法配制。主要仪器有凝胶成像系统(SYNGENE),实时定量PCR仪(BIORAD),倒置荧光显微镜显微镜(OLYMPUS),CO2培养箱(HERA cell 150, Thermo electron)。
1.2 细胞培养与转染大鼠巨噬细胞系NR8383细胞按5×105/孔种于六孔板中,用含20%胎牛血清(Hyclone)的F-12K培养于37 ℃含有5% CO2的培养箱内,待细胞融合度达到80%~90%,用不同浓度(10、50、100 nmol/L)的miR-144 mimics和inhibitor以及相应的阴性对照(mNC和iNC)通过脂质体2000在无血清培养基中转染细胞,5 h后更换培养基为完全培养基,24 h后收获细胞用于后续实验。
1.3 RT-qPCR所有的组织和细胞用TRIzol®试剂提取总RNA。MicroRNA表达的检测:取2 μg总RNA用microRNA反转录试剂盒PrimeScript® miRNA cDNA Synthesis kit反转为miRNA cDNA,用SYBR® PrimeScriptTM RT-PCR Kit II进行Realtime-PCR检测miR144、U6的表达。miRNA特异的RT-qPCR反应条件为:Cycle 1的Step 1:95 ℃ 2 min;Cycle 2的Step 1:95 ℃ 15 s,Step 2:60 ℃ 45 s,共40循环。引物序列和退火温度见表 1。miR-144的下游引物为反转录试剂盒提供的Uni-miR RT-qPCR通用引物。
| 表 1 microRNA RT-qPCR引物相关信息 Table 1 Information of the primers for microRNA RT-qPCR |
基因mRNA表达的检测:取5 μg总RNA用反转录试剂盒RevertaidTM First Strand cDNA Synthesis反转为cDNA,通过用SYBR® PrimeScriptTM RT-PCR Kit II进行Realtime-PCR检测β-actin、TLR2、TNF-α的表达。引物序列和退火温度见表 2。RT-qPCR的反应条件为:Cycle 1的Step 1:95 ℃ 3 min;Cycle 2的Step 1:95 ℃ 10 s,Step 2:退火30 s,共40循环,Step 3:72 ℃ 30 s并收集荧光数据;Cycle 3的Step 1:55 ℃ 30 s,共41循环,并收集荧光数据做熔解曲线。
| 表 2 RT-qPCR引物相关信息 Table 2 Primers for RT-qPCR of TLR2 and TNF-α mRNAs |
在GeneBank里查找Rattus norvegicus Toll like receptor2 mRNA的全序列(序列号:NM_198769)。利用Primer Premier 5.0软件设计包含miR-144野生及突变结合位点的TLR2 mRNA 3'UTR区报告基因载体的引物,上游引物带有限制性内切酶SacⅠ的酶切位点,序列为:5'-TCCGAGCTCGTTCTCCACCCTGTTTC-3',下游引物带有限制性内切酶XbaⅠ的酶切位点,序列为:5'-GCCGCTCTAGATATATAACAGTATTCAAG-3',PCR扩增产物长186 bp;包含miR-144突变结合位点的TLR2 mRNA 3'UTR区报告基因载体的引物,上游引物带有限制性内切酶Sac Ⅰ的酶切位点,序列为:5'-TCCGAGCTCGTTCTCCACCCTGTTTC-3',下游引物带有限制性内切酶XbaⅠ的酶切位点,序列为: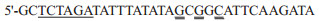
首先取E3大鼠肝组织50~60 mg TRIzol®试剂说明书提取肝总RNA并定量,RT-PCR获取含miR-144野生及突变结合位点的TLR2 mRNA 3'UTR的目的片段。复苏并扩增pmirGLO载体菌种,提取质粒并定量。用限制性内切酶SacⅠ和XbaⅠ对目的片段PCR产物和pmirGLO质粒进行双酶切,酶切产物经1%琼脂糖凝胶电泳后,再紫外透射下切下目的条带和pmirGLO质粒,用DNA琼脂糖凝胶纯化试剂盒分别回收并定量。
用T4 DNA连接酶连接双酶切纯化后的pmirGLO载体和目的片段(载体DNA:目的片段DNA摩尔数之比为1:3),选用10 μl连接体系,12 ℃连接过夜。取连接产物5 μl加入100 μl DH5α感受态细胞,分设加PBS的阴性对照和加pmirGLO质粒阳性对照。具体操作遵照分子克隆指南。
1.6 用PCR、SacⅠ和XbaⅠ双酶切以及DNA测序鉴定重组质粒pmir-TLR2-3'UTR及pmir-mutant-TLR2-3'UTR从pmir-TLR2-3'UTR及pmir-mutant-TLR2-3'UTR LB平板上随机挑取3~5个阳性克隆,加入5 ml含有100 μg/ml氨苄青霉素的LB液体培养基中,在恒温振荡器,37 ℃剧烈震荡培养16 h。在超净工作台内,取1 ml菌液4 ℃保存,用于测序。剩余菌液,按照质粒小提试剂盒提取重组质粒pmir-TLR2-3'UTR及pmir-mutantTLR2-3'UTR。以提取的pmirGLO质粒和重组质粒pmir-TLR2-3'UTR及pmir-mutant-TLR2-3'UTR为模版,PCR扩增含miR-144野生及突变结合位点的TLR2mRNA 3'UTR的目的片段;按照20 μl双酶切体系,用限制性内切酶SacⅠ和XbaⅠ双酶切鉴定重组质粒pmir-TLR2-3'UTR及pmir-mutant-TLR2-3'UTR后;1%琼脂糖凝胶电泳检测PCR及双酶切产物并照像。将重组质粒pmir-TLR2-3'UTR及pmir-mutant-TLR2-3'UTR菌液送给公司测序。
1.7 荧光素酶活性的检测上述构建的载体通过去E.Z.N.A.TM Endo-free Plasmid Maxi Kit(Omega Bio-tek)提取试剂盒提取无内毒素质粒。HEK-293T细胞按5×104/孔种于48孔板,培养于含10%胎牛血清的DMEM培养基中,待细胞融合度达到80%,在脂质体2000的介导下,分别用100 ng三种载体(pmir vector、pmir-TLR2-3'UTR vector、pmirmutant-TLR2-3'UTR vector)与100 nmol/L miR-144 mimics共转染293T细胞24 h后,在Luminometer(VICTORTM 2030 Multilabel Plate Reader, PE, USA)用检测双荧光素酶试剂盒Dual-Luciferase® Reporter 1000 Assay System(Promega)检测荧光素酶的活性,相对荧光素酶活性值通过对每个样品用海蜃荧光素酶的活性校正萤火虫荧光素梅的活性从而得到的(firefly luciferase activity/renilla luciferase activity, FL/RL),不同组间标准化后的相对荧光素酶活性用于进行统计分析。
1.8 统计学处理采用StatView统计软件进行统计分析,实验组与对照组之间显著性比较用单因素方差分析(One-way ANOVA, Bonferroni),以P < 0.05为有显著性差异。
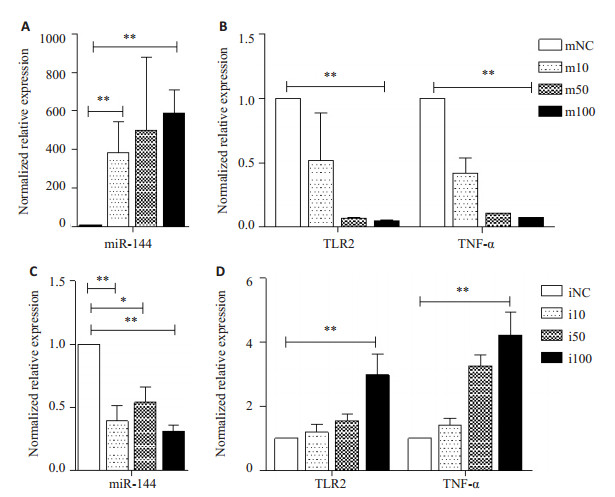
2 结果 2.1 不同浓度miR-144的mimics和inhibitor对TLR2及其下游促炎因子表达的影响结合文献,用10、50、100 nmol/L miR-144 mimics转染NR8383细胞,RT-qPCR结果显示,与mNC组相比,3个浓度转染组miR-144的表达均升高(U6为内参基因),其中以100 nmol/L升高最显著(高于对照组约600倍)(图 1A)。另外,我们发现在不同浓度mimics作用下,TLR2的表达与对照组相比显著下降(β-actin为内参基因),其中以100 nmol/L组下调最多。其下游促炎因子TNF-α也在该浓度miR144 mimics作用下,表达显著降低(图 1B)。因此,我们选用100 nmol/L作为miR144 mimics的后续工作浓度。
|
图 1 miR-144的mimics和inhibitor对大鼠巨噬细胞系NR8383 TLR2及其下游分子的影响 Figure 1 Effect of miR-144 mimics and inhibitor on TLR2 and its target genes in NR8383 cells. |
用10、50、100 nmol/L miR144 inhibitor处理NR8383细胞后,RT-qPCR结果显示:与iNC组相比,3个浓度转染组miR-144的表达均降低(U6为内参基因),其中以100 nmol/L降低最显著(图 1C)。另外,我们发现在该浓度下,TLR2其下游促炎因子TNF-α的表达与对照组相比均显著升高(β-actin为内参基因)(图 1D),我们选用100 nmol/L作为miR-144 inhibitor的后续工作浓度。
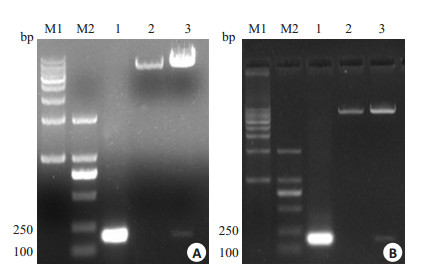
2.2 pmir-TLR2-3'UTR和pmir-mutant-TLR2-3'UTR重组载体的鉴定为了明确miR-144与TLR2的靶向关系,我们构建了miR-144野生及突变结合位点的TLR2 mRNA 3'UTR区报告基因载体。挑选出含氨苄青霉素的LB平板上挑选重组质粒的阳性克隆,液体LB培养基扩增后提取质粒并定量。以提取的pmir-TLR2-3'UTR重组质粒分别为模板,用构建pmir-TLR2-3'UTR载体所用引物分别进行PCR扩增,1%琼脂糖凝胶电泳检测PCR产物,结果显示在100 bp和250 bp之间(图 2A)有一条明显的条带,它们与目的片段的大小相一致;以提取的pmirGLO质粒为模板,用构建pmir-TLR2-3'UTR载体所用引物进行PCR扩增,1%琼脂糖凝胶电泳检测PCR产物,结果均显示无任何条带。这说明pmir-TLR2-3'UTR重组质粒中均包含有目的片段。
|
图 2 pmir-TLR2-3' UTR重组载体和突变体的鉴定 Figure 2 Identification of the constructs pmir-TLR2-3'UTR and pmir-mutant-TLR2-3'UTR by PCR and SacⅠ and XbaⅠ double digestion. A: Identification of pmir-TLR2-3'UTR. M1 and M2 are markers for 1 kb and 250 bp, respectively; Lane 1: PCR product; Lanes 2, 3: Double digestion product of pmirGLO vector and pmir-TLR2-3'UTR, respectively; B: Identification of pmir-mutant-TLR2-3'UTR. M1, M2: Markers for 1 kb and 250 bp, respectively. Lane 1: PCR product; Lanes 2, 3: Double digestion products of pmirGLO vector and pmir-mutantTLR2-3'UTR, respectively. |
限制性内切酶SacⅠ和XbaⅠ双酶切pmir-TLR2-3'UTR重组质粒,1%琼脂糖凝胶电泳图上可见2条清晰条带,其中1个位于DNA marker所示的7000~8000 bp之间,另一个位于100~250 bp之间,这分别与pmirGLO质粒(7350 bp)及目的片段(186 bp)的大小相一致(图 2A);然而pmirGLO质粒用限制性内切酶SacⅠ和XbaⅠ双酶切后,在1%琼脂糖凝胶电泳图上只有1个条带位于DNAmarker所示的7000~8000 bp之间,这只与pmirGLO质粒(7350 bp)的大小相一致(图 2A)。这些说明pmir-TLR2-3'UTR重组质粒经过SacⅠ和XbaⅠ双酶切后可以释放出目的片段。对构建的pmir-TLR2-3'UTR重组质粒进行测序,并用DNAssist2.2软件将测序结果与从GenBank中拷贝的TLR2 mRNA 3'UTR序列进行比对,pmir-TLR2-3'UTR重组质粒中插入片段与GenBank中TLR2mRNA 3'UTR序列完全相同。
用PCR和酶切的方法鉴定pmir-mutant-TLR2-3'UTR重组载体,结果见图 2B。对构建的pmir-mutantTLR2-3'UTR重组质粒进行测序,并用DNAssist2.2软件将测序结果与从GenBank中拷贝的TLR2 mRNA 3'UTR序列进行比对,pmir-mutant-TLR2-3'UTR重组质粒中插入片段与GenBank中TLR2 mRNA 3'UTR序列有3个碱基不同,这恰好是我们设计的突变位点。
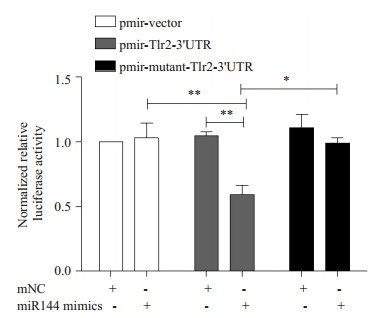
2.3 miR-144通过与TLR2 mRNA 3'UTR区靶向结合调节TLR2的表达为了证实miR-144是否影响TLR2的表达,我们选择上述携带了野生型和突变体并鉴定成功的两种载体pmir-TLR2-3'UTR vector和pmir-mutant-TLR2-3'UTR,在脂质体2000介导下,用100 nmol/L miR-144 mimics或者mNC分别与100 ng 3种载体(空载体以及两种构建体)共转染HEK 293T细胞24 h。结果表明,与mNC组相比,在miR-144 mimics作用下,293T细胞转染pmir-TLR2-3'UTR载体后相对荧光素酶的活性显著降低,而pmir-vector和pmir-mutant-TLR2-3'UTR组的荧光素酶活性则没有因miR-144 mimics的作用而明显改变。另外,即使在miR44 mimics和载体共转染的作用下,与空载体pmir-vector和突变体pmir-mutantTLR2-3'UTR组相比,野生型的pmir-TLR2-3'UTR组转染的293T细胞相对荧光素酶活性显著降低。但是与mNC共转染的3组间荧光素酶活性没有明显差异(图 3)。这些结果说明miR-144可以直接靶向结合于大鼠TLR2 mRNA的3'UTR区,即大鼠TLR2是miR-144的靶基因。
|
图 3 鉴定MiR-144与大鼠TLR2 mRNA 3'-UTR的作用关系 Figure 3 Identification of the relation between MiR-144 and 3'-UTR of rat TLR2 mRNA. *P < 0.05, **P < 0.01. |
多项报道证实肝脏TLR信号通路的激活和促炎细胞因子的产生在非酒精性脂肪肝炎(NASH)的发病过程中有关键意义[15-17]。其中,TLR2,TLR4和TLR9与NASH的联系尤为密切。被饱和脂肪酸触发的TLR4信号可通过激活X-盒结合蛋白1从而介导肥胖诱导的NASH的发生[18],它同样可以由肠道微生物群的内毒素所触发,继而通过肝库夫细胞TNF-α信号介导果糖诱导的NASH的发生[19]。另外,TLR2和TLR9也被认为可分别通过激活炎性体或诱导白细胞介素1β,从而促进胆碱缺乏氨基酸缺乏的饮食诱导的NASH的发生[14, 20]。在前期研究成果中,我们发现在高脂饮食诱导的代谢综合征模型中,TLR2及其下游分子TNF-α在模型E3大鼠肝脏表达显著升高,这与另外两个研究小组的报道一致[17, 20]。这也说明TLR2的激活与促炎因子的产生与高脂饮食诱导的非酒精性脂肪肝炎的发生密切相关。
近年来,越来越多的报道证实miRNAs参与TLR信号通路的异常激活。比如miR-105和miR-19能调节口腔角质细和人类风湿纤维细胞样滑膜细胞TLR2的表达[21-22]。因此,我们有理由相信在高脂饮食诱导的代谢综合征模型中TLR2的高表达可能与某些miRNAs的表达改变有关。研究表明,TLR2和TLR4的激动剂,以及TNF-α可以诱导单核细胞miR-132和miR-9的表达,而上调的miR-132和miR-9可能通过激活TLR2和/或升高TNF-α从而促进NASH的发生[23-24]。我们通过生物信息学预测软件,预测了一系列可能与TLR2 mRNA 3'UTR区结合的miRNAs,在前期检测的miRNAs预实验中,仅有miR-144在高脂饮食诱导的E3大鼠代谢综合征模型肝脏中显著低表达,推测TLR2可能是miR-144的靶基因。miR-144被认为是一种常见人类肿瘤疾病的的miRNA标志[25],可促进红细胞的生成[26]。在2型糖尿病中,升高的循环miR-144可以通过抑制胰岛素受体底物从而损害胰岛素信号[27]。然而,目前尚无文献报道它与TLR2的靶向调节关系,因此我们设计一系列实验明确它们的作用关系。
有文献报道在炎症性肝损害发生时,定居在肝内的巨噬细胞库夫细胞会被损伤和病原体相关的分子模式通过TLRs激活[28-29]。前期研究发现,在高脂饮食诱导的代谢综合征模型肝、表达上调的TLR2主要位于肝库夫细胞,故推测肝库夫细胞高表达的TLR2可能与触发肝脏炎症和相关炎症因子的产生密切相关,进而导致NASH的发生有关。TNF-α是TLR2信号通路的下游靶向分子,被认为在NASH发生发展中起到招募炎症细胞[30]、损害胰岛素信号[31]的作用。因此,用不同浓度的miR-144的mimics和inhibitor作用于大鼠巨噬细胞系NR8383细胞,进一步研究miR-144对TLR2及其下游促炎因子的影响。结果显示,随着miR-144的升高或降低,TLR2以及促炎因子TNF-α的表达呈现相反的趋势,尤其在100 nmol/L浓度时最明显。因此,miR-144能负性调节TLR2及其下游信号分子TNF-α的表达。
为了明确miR-144是否靶向调节TLR2的表达,用miR-144mimics和成功构建的野生型pmir-TLR2-3'UTR和突变型pmir-mutant-TLR2-3'UTR双荧光素酶报告基因载体同时转染293T细胞,研究miR144与TLR2 mRNA的3'UTR区靶向结合关系。结果发现miR144的mimics与野生型pmir-TLR2-3'UTR共转染时,与空载体组和突变体pmir-mutant-TLR2-3'UTR组相比,293T细胞的荧光素酶活性显著降低,说明大鼠TLR2 mRNA的3'UTR区存在miR-144的结合位点,即TLR2是miR144的靶基因。这为我们今后研究miR-144对大鼠TLR2调节及其在NASH发展中的作用机制提供了坚实的基础。
| [1] | Akira S, Takeda K. Toll-like receptor signaling[J]. Nat Rev Immunol,2004, 4 (7) : 499-511. DOI: 10.1038/nri1391. |
| [2] | Kawai T, Akira S. The role of pattern-recognition receptors in innate immunity:update on Toll-like[J]. Nat Immunol,2010, 11 (5) : 373-84. DOI: 10.1038/ni.1863. |
| [3] | Kawai T, Akira S. Toll-like receptors and their crosstalk with other innate receptors in infection and immunity[J]. Immunity,2011, 34 (5) : 637-50. DOI: 10.1016/j.immuni.2011.05.006. |
| [4] | Bartel DP. MicroRNAs: target recognition and regulatory functions[J]. Cell,2009, 136 (2) : 215-33. DOI: 10.1016/j.cell.2009.01.002. |
| [5] | Gu S, Kay MA. How do miRNAs mediate translational repression[J]. Silence,2010, 1 (1) : 11. DOI: 10.1186/1758-907X-1-11. |
| [6] | Davalos A, Goedeke L, Smibert P, et al. miR-33a/b contribute to the regulation of fatty acid metabolism and insulin signaling[J]. Proc Natl Acad Sci USA,2011, 108 (22) : 9232-7. DOI: 10.1073/pnas.1102281108. |
| [7] | Ambros V. The functions of animal microRNAs[J]. Nature,2004, 431 (76) : 350-5. |
| [8] | He JF, Luo YM, Wan XH, et al. Biogenesis of MiRNA-195 and its role in biogenesis, the cell cycle, and apoptosis[J]. J Biochem Mol Toxicol,2011, 25 (6) : 404-8. DOI: 10.1002/jbt.v25.6. |
| [9] | Zhu W, Zhu D, Lu S, et al. miR-497 modulates multidrug resistance of human cancer cell lines by targeting BCL2[J]. Med Oncol,2012, 29 (1) : 384-91. DOI: 10.1007/s12032-010-9797-4. |
| [10] | Zhong N, Sun J, Min Z, et al. MicroRNA-337 is associated with chondrogenesis through regulating TGFBR2 expression[J]. Osteoarthritis Cartilage,2012, 20 (6) : 593-602. DOI: 10.1016/j.joca.2012.03.002. |
| [11] | Frost RJ, Olson EN. Control of glucose homeostasis and insulin sensitivity by the Let-7 family of microRNAs[J]. Proc Natl Acad Sci USA,2011, 108 (52) : 21075-80. DOI: 10.1073/pnas.1118922109. |
| [12] | Ryu HS, Park SY, Ma D, et al. The induction of microRNA targeting IRS-1 is involved in the development of insulin resistance under conditions of mitochondrial dysfunction in hepatocytes[J]. PLoS One,2011, 6 (3) : e17343. DOI: 10.1371/journal.pone.0017343. |
| [13] | Dudda JC, Salaun B, Ji Y, et al. MicroRNA-155 is required for effector CD8(+) T cell responses to virus infection and cancer[J]. Immunity,2013, 38 (4) : 742-53. DOI: 10.1016/j.immuni.2012.12.006. |
| [14] | Miura K, Yang L, Van Rooijen N, et al. Toll-Like receptor 2 and palmitic acid cooperatively contribute to the development of nonalcoholic steatohepatitis through inflammasome activation in mice[J]. Hepatology,2013, 57 (2) : 577-89. DOI: 10.1002/hep.26081. |
| [15] | Miura K, Seki E, Ohnishi H, et al. Role of toll-like receptors and their downstream molecules in the development of nonalcoholic fatty liver disease[J]. Gastroenterol Res Pract,2010, 2010 : 1-9. |
| [16] | Wagnerberger S, Spruss A, Kanuri GA, et al. Toll-like receptors 1-9 are elevated in livers with fructose-induced hepatic steatosis[J]. Br J Nutr,2012, 107 (12) : 1727-38. DOI: 10.1017/S0007114511004983. |
| [17] | Petrasek J, Csak T, Ganz M, et al. Differences in innate immune signaling between alcoholic and non-alcoholic steatohepatitis[J]. J Gastroenterol Hepatol,2013, 28 (1, SI) : 93-8. |
| [18] | Ye D, Li FY, Lam KS, et al. Toll-like receptor-4 mediates obesityinduced non-alcoholic steatohepatitis through activation of X-box binding protein-1 in mice[J]. Gut,2012, 61 (7) : 1058-67. DOI: 10.1136/gutjnl-2011-300269. |
| [19] | Sawada K, Ohtake T, Hasebe T, et al. Augmented hepatic Toll-like receptors by fatty acids trigger the pro-inflammatory state of nonalcoholic fatty liver disease in mice[J]. Hepatol Res,2014, 44 (8) : 920-34. DOI: 10.1111/hepr.2014.44.issue-8. |
| [20] | Miura K, Kodama Y, Inokuchi S, et al. Toll-like receptor 9 promotes steatohepatitis by induction of interleukin-1beta in mice[J]. Gastroenterology,2010, 139 (1) : 323-34.e7. DOI: 10.1053/j.gastro.2010.03.052. |
| [21] | Benakanakere MR, Li QY, Eskan MA, et al. Modulation of TLR2 protein expression by miR-105 in human oral keratinocytes[J]. J Biol Chem,2009, 284 (34) : 23107-15. DOI: 10.1074/jbc.M109.013862. |
| [22] | Philippe L, Alsaleh G, Suffert G, et al. TLR2 expression is regulated by microRNA miR-19 in rheumatoid fibroblast-like synoviocytes[J]. J Immunol,2012, 188 (1) : 454-61. DOI: 10.4049/jimmunol.1102348. |
| [23] | Bazzoni F, Rossato M, Fabbri M, et al. Induction and regulatory function of miR-9 in human monocytes and neutrophils exposed to proinflammatory signals[J]. Proc Natl Acad Sci USA,2009, 106 (13) : 5282-7. DOI: 10.1073/pnas.0810909106. |
| [24] | Taganov KD, Boldin MP, Chang KJ, et al. NF-kappaB-dependent induction of microRNA miR-146, an inhibitor targeted to signaling proteins of innate immune responses[J]. Proc Natl Acad Sci USA,2006, 103 (33) : 12481-6. DOI: 10.1073/pnas.0605298103. |
| [25] | Wang W, Peng B, Wang D, et al. Human tumor microRNA signatures derived from large-scale oligonucleotide microarray datasets[J]. Int J Cancer,2011, 129 (7) : 1624-34. DOI: 10.1002/ijc.v129.7. |
| [26] | Rasmussen KD, Simmini S, Abreu-Goodger CA, et al. The miR-144/ 451 locus is required for erythroid homeostasis[J]. J Exp Med,2010, 207 (7) : 1351-8. DOI: 10.1084/jem.20100458. |
| [27] | Karolina DS, Armugam A, Tavintharan S, et al. MicroRNA 144 impairs insulin signaling by inhibiting the expression of insulin receptor substrate 1 in type 2 diabetes mellitus[J]. PLoS One,2011, 6 (8) : e22839, 1-19. |
| [28] | Schwabe RF, Seki E, Brenner DA. Toll-Like receptor signaling in the liver[J]. Gastroenterology,2006, 130 (6) : 1886-900. DOI: 10.1053/j.gastro.2006.01.038. |
| [29] | Jaeschke H. Reactive Oxygen and mechanisms of inflammatory liver injury: Present concepts[J]. J Gastroenterol Hepatol,2011, 26 (1) : 173-9. |
| [30] | Tomita K, Tamiya G, Ando S, et al. Tumour necrosis factor alpha signalling through activation of Kupffer cells plays an essential role in liver fibrosis of non-alcoholic steatohepatitis in mice[J]. Gut,2006, 55 : 415-24. DOI: 10.1136/gut.2005.071118. |
| [31] | Tosello-Trampont AC, Landes SG, Nguyen V, et al. Kuppfer cells trigger nonalcoholic steatohepatitis development in diet-induced mouse model through tumor necrosis factor-α production[J]. J Biol Chem,2012, 287 (48) : 40161-72. DOI: 10.1074/jbc.M112.417014. |
 2016, Vol. 35
2016, Vol. 35
