2. 美国加州大学欧文分校健康科学院公共卫生部,加州 尔湾 92617 ;
3. 广东普通高校新发传染病防治重点实验室,广东 广州 510515 ;
4. 广西壮族自治区疾病预防控制中心,广西 南宁 530028 ;
5. 江苏省血吸虫病防治研究所,江苏 无锡 214064
2. Program in Public Health, College ofHealth Sciences, University of California at Irvine, Irvine 92617, USA ;
3. Key Laboratory of Prevention and Control for Emerging Infectious Diseases of Guangdong Higher Institutes, Guangzhou 510515, China ;
4. Guangxi Zhuang Autonomous Region Center for Diseases Control and Prevention, Nanning 530028, China ;
5. Jiangsu Institute of Parasitic Diseases, Wuxi 214064, China
中华按蚊(Anopheles sinensis)属于节肢动物门、昆虫纲、双翅目蚊科,分布于除新疆和青海以外的全国各省区,是广大平原地区,特别是水稻种植区疟疾和马来丝虫病的重要媒介[1]。有研究显示不同地理分布区的中华按蚊在生态习性、传病能力和对杀虫剂的抗性等方面均存在一定的差异[2-5]。中华按蚊不同地理种群的遗传分化研究将有助于解释这些差异。目前已有研究[6-9]分析我国按蚊属内蚊虫的系统发育关系,但仅有运用微卫星标记分析了我国中华按蚊的种群遗传结构报道[10]。近年,分子遗传学多用于研究种群遗传结构,常用的分子标记为微卫星和线粒体基因,其中线粒体DNA细胞色素氧化酶亚基(mitochondria l DNA cytochrome oxidase subunit I, mtDNA-COI)基因序列因位置比较保守,已被用于我国多种蚊虫的种群遗传结构分析,成为具重要价值的一种有效的分子标记[6-8, 11]。本研究采用线粒体DNA(mitochondrial DNA,mtDNA)分子标记技术,对mtDNA-COI片段进行序列分析,以探讨我国中华按蚊种群遗传多样性、遗传分化、种群系统发育关系。通过对中华按蚊的遗传现状的了解,以期为传疟媒介的治理提供基础资料。
1 材料与方法 1.1 样本来源实验中华按蚊为2010年~2012年的7~9月份采集自中国境内6个地理自然种群的现场样本(图 1),主要用吸蚊器、诱蚊灯及吸蚊管采集各地区中华按蚊的成蚊。样本采集地区地理形势变化多样,由北向南包括:山东省济宁市(N35o20',E116o40)、'安徽省蒙城县(N33o14',E116o35)、'江苏省南京市(N31o51',E119o01)、'贵州省独山县(N25o49',E107o32)、'广西省东兰县(N24o30',E107o22)、'云南省元阳县(N23o13',E102o50)。' 6个地理自然种群中,山东、江苏和安徽采集点三省位于我国华东地区的黄河、长江中下游平原水网地带,是以中华按蚊为媒介的间日疟流行区;贵州、广西和云南三省位于我们西南部的云贵高原地带,地理环境特殊和气候复杂多样,疟区分布较为复杂,中华按蚊是该地区主要传疟媒介之一,6个地理自然种群能较好的代表了我国中华按蚊的不同地理种群特点。野外现场采集到的样本带回室内按照陆宝麟[12]的方法进行形态学鉴定,95%乙醇保存,置-20 ℃备用。
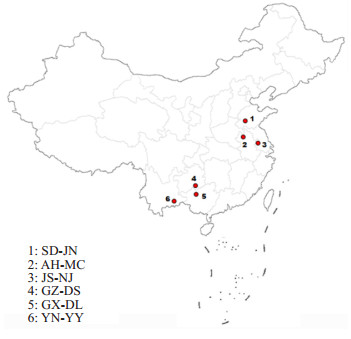
|
图 1 采样点地图 Figure 1 Map of the sampling sites. |
将单蚊置于吸水纸上晾干放入1.5 ml EP管,加入120 μl新配置的DEB裂解液和1.5 μl RNAse酶(10 μg/μl),用研磨棒使其匀浆化,置37 ℃孵育60 min;加入3.0 μl蛋白K(20 μg/μl),置50 ℃孵育60 min;每管加入氯仿和酚各60 μl,涡旋混匀以13 000 g离心10 min;将上清液移入0.5 ml EP管中,加入300 μl预冷95%乙醇,振荡10 min放置-20 ℃沉淀过夜;以13 000 g离心10 min,弃上清,加入300 μl预冷70%乙醇,以13 000 g离心10 min,弃上清,开盖置于37 ℃恒温箱中干燥后,100 μl 1×TE(PH7.4)重悬DNA沉淀,-20 ℃长期保存备用。对提取的DNA样本进行中华按蚊种群分子鉴定,采用Gao等[13]和Joshi等[14]建立的,已为国内外研究者广泛采用的方法对单蚊DNA样本进行中华按蚊种群分子鉴定,确认为中华按蚊的样本用于后续研究。
1.3 mtDNA-COI扩增和测序以中华按蚊基因组DNA为模板,根据Folmer [15]设计mtDNA-COI基因片段的兼并引物:上游5'-CMCTT TCATCTGGAATTGCT-3',下游5'-AAAAATGTTGA GGGAArAATGTTA-3'。PCR反应体系22 μl,DreamTaq Green PCR Master Mix(Thermo公司)11.50 μl、上下游引物各0.50 μ(l 10 μmol/L)、基因组DNA模板1.00 μl、灭菌蒸馏水8.50 μl。PCR反应条件:95 ℃ 3 min;94 ℃ 30 s;55 ℃ 30 s;72 ℃ 1 min,共35个循环;72 ℃ 6 min;4 ℃保存。取5 μl PCR产物经1.5%琼脂糖凝胶电泳,凝胶成像系统观察拍照记录结果。扩增成功样品送美国Genewize公司用ABI 377测序仪进行双向测序。
1.4 数据分析采用Bioedit 7.0软件对测序结果进行拼接处理,正反链序列结果一致的样本再进行下一步的分析。登陆GenBank对序列进行BLAST同源性比对。采用DnaSP 5.0软件[16]确定变异位点,计算中华按蚊各种群的核酸多样性(nucleotide diversity)和单倍型多样性(haplotype diversity)及中性检验[17](test of selective neutrality)和错配分布(mismatch distribution)分析。应用Arlequin 3.1软件[18]计算各地方种群间的遗传距离及进行分子方差分析(Analysis of molecular variance,AMOVA),以明确种群内和种群间的遗传多样性和分化程度,通过IBDWS[19](IBD Web Service)在线软件进行Mantel检验,以明确遗传距离与地理距离的相关性。以白跗按蚊(Anopheles albitarsis,GenBank: HQ335344.1)为外群,依据遗传距离Kimura 2-parameter双参数法,使用PHYLIP 3.6[20]软件中NJ(Neighbor-Joining,邻接法)进行聚类分析构建系统进化树,探讨中华按蚊各地理种群的系统发生关系。
2 结果 2.1 中华按蚊mtDNA-COI基因片段的碱基组成及变异分析六个种群共123个雌蚊经分子鉴定为中华按蚊,PCR扩增获得123条中华按蚊的mtDNA -COI基因序列,经比对后选用814 bp的片段序列进行分析。mtDNA -COI序列片段中A、C、G、T平均碱基组成统计结果显示:4种碱基的平均含量A:33.3%,T:37.9%,G:15.1%,C:13.7%。(A+T)%=71.2%>(G+C)%=28.8%,表现出AT偏倚特征,符合线粒体DNA的特性。
变异分析:mtDNA-COI基因序列片段814个位点中有730个保守位点,84个变异位点,35个单态位点,49个简约信息位点,无插入和缺失。本研究样本序列获得91种单倍型(至少一个碱基差异),其中不同种群共享单倍型21种,种群特有单倍型70种,单倍型多样性:0.992,核酸多样性:0.00907。中华按蚊不同种群mtDNA -COI序列遗传多样性具体见(表 1)。
| 表 1 基于mtDNA-COI基因序列中华按蚊种群遗传多样性 Table 1 Haplotype and nucleotide diversity of the mitochondrial DNA-COI gene |
对6个地理种群的中华按蚊进行分子变异等级分析(表 2),结果显示:种群内差异为92.67%,种群间差异为7.33%,可见种群内变异明显大于种群间变异,差异显著(P < 0.001),说明遗传分化主要来自于种群内部。
| 表 2 中华按蚊种群遗传变异的分子变异等级分析结果 Table 2 AMOVA analysis of genetic variation in Anopheles sinensis populations |
对6个中华按蚊种群的遗传分化系数(FST)和基因流(Nm)进行计算表明,种群间的遗传分化系数Fst范围在0.0005~0.2423之间(表 3),其中云南种群与其它5个种群之间的分化水平均达到显著(P < 0.05)或者极显著(P < 0.01)。该种群与其它5个种群之间遗传分化系数更是达到0.0743~0.2423之间。同时,它与各种群间的基因流Nm值均较小,表明种群间的基因交流程度较低。贵州,广西,江苏和山东种群间有丰富的基因流(Nm>10)。
| 表 3 中华按蚊群体之间的基因流Nm(右上角)和遗传距离FST (左下角) Table 3 Pairwise Nm, genetic distance (FST) for populations of Anopheles sinensis |
IBDWS在线软件Mantel检验(图 2),结果显示:本研究所涉及的中华按蚊6个地理种群遗传距离和地理距离的相关系数r=0.375(P < 0.05),基本呈现随距离增加基因流减少的趋势,表明种群的遗传距离和地理距离之间符合距离隔离模型,可能有地理隔离现象。
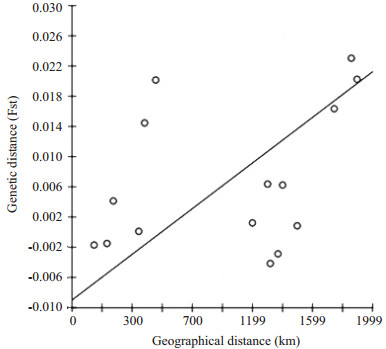
|
图 2 中华按蚊种群间遗传距离和地理距离相关性 Figure 2 Isolation by distance and relationship between geographic and genetic distances in Anopheles sinensis populations. |
中性检验(表 4)结果显示:除云南种群外,其余种群Tajima's D均为负值且不显著(P > 0.05),说明中华按蚊种群的线粒体COI基因符合中性模型,进化过程中选择压力小。Fu's Fs检验值均为负数且显著(P < 0.05),说明本研究所涉及的我国中华按蚊在近期历史发展过程中经历了种群扩张状态。错配分析(图 3)结果显示:本研究所涉及的6个中华按蚊地理种群分布曲线呈单峰,符合种群扩张模型所期望的理论分布,进一步说明中华按蚊近期出现过扩张发展。
| 表 4 基于mtDNA-COI序列中华按蚊种群中性检验数值 Table 4 Neutrality test of Anopheles sinensis based on mitochondrial DNA-COI |
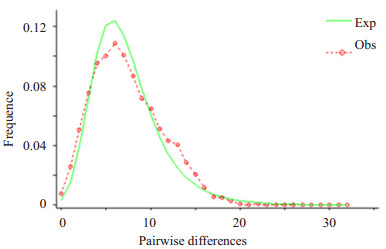
|
图 3 基于mtDNA-COI部分序列中华按蚊种群错配分析 Figure 3 Mismatch distribution of Anopheles sinensis based on partial sequence of COI gene. |
根据遗传距离Kimura 2-parameter(双参数法)对6个地理种群中华按蚊进行聚类关系分析(图 4),系统树结果显示:中华按蚊各地理种群聚为一大支,与外群白跗按蚊明显分开,形成2个不同的大分支。中华按蚊不同地理种群间又分成若干分支:江苏南京种群和山东济宁种群先聚在一起,然后与贵州罗甸种群、广西东兰种群及安徽蒙城种群聚为一支;云南元阳种群与其他种群相对独立,在系统树中为孤立一支。
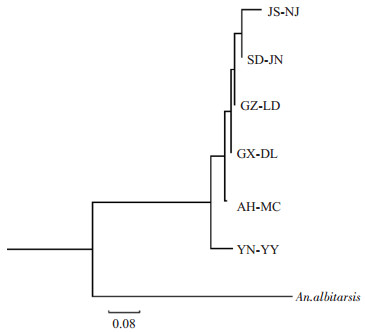
|
图 4 中华按蚊各种群间的聚类关系(NJ法) Figure 4 Clustering relationship among Anopheles sinensis populations based on pairwise genetic distance (NJ method). |
中华按蚊分布于除新疆和青海以外的全国各省区,是广大平原地区,特别是水稻种植区疟疾和马来丝虫病的重要媒介,对其种群遗传结构的研究,将对传播疾病的防控提供重要的指导依据。
线粒体DNA是真核细胞中的遗传物质,由卵细胞传递给后代,呈母系遗传。由于它具有分子量小、进化速度快,拷贝数量多的特点,从而成为目前研究物种起源进化及种群遗传分析的理想研究对象。在昆虫种群遗传学研究中,细胞色素C氧化酶亚基基因COI、COII,细胞色素b(cytochrome b, Cytb)基因等常作为分析亲缘关系较近的种、亚种的系统发育分析以及地理种群之间系统关系的分子工具[21]。本研究通过PCR扩增获得123条中华按蚊mtDNA -COI基因序列,经比对后,选用814 bp的片段序列进行分析,从中华按蚊COI基因碱基特点上看,(G+C)含量28.8%,(A+T)含量71.2%,统计结果与我国有报道的雷氏按蚊[6]COI基因序列(G+C)含量31.4%,(A+T)含量68.6 %,多斑按蚊复合体[7]COI基因序列(G+C)含量29.1%,(A+T)含量70.9%;及大劣按蚊[8]COI基因序列(A+T)含量71.01%~71.22%的碱基组成结果相近,具有明显的A/T碱基偏倚性。6个中华按蚊地理种群COI基因中检测到730个保守位点,84个变异位点,35个单态位点,49个简约信息位点,无插入和缺失。6个种群序列的高度相似提示我们,中华按蚊有着共同的祖先mtDNA-COI序列,随着群体扩张和迁移,以及地理以及气候等因子的变化,mtDNA-COI序列也不断发生突变,从而形成了目前的群体遗传结构格局。123个样品中检测到91种单倍型,其中共享单倍型21种,特有单倍型70种,单倍型多样性h:0.992,核酸多样性π:0.00907,表明以线粒体COI序列分析中华按蚊具有丰富的遗传多样性,线粒体COI序列可以作为研究物种起源进化及种群遗传分析的理想分子标志。
本研究对6个地理种群AMOVA分析显示,中华按蚊种群内变异明显大于种群间变异(P < 0.001),遗传分化主要来自于种群内部。而在Mantel检验中却显示,种群遗传距离和地理距离的相关系数r=0.375,表明6个中华按蚊种群遗传结构格局基本符合距离隔离模型。对6个中华按蚊种群的遗传分化系数(FST)和基因流(Nm)进行分析,结果表明,种群间存在遗传分化,其中云南种群与其它5个种群之间的分化水平最高。基因流分析结果进一步证实该种群与其它5个种群之间的基因交流程度较低。可能存在生殖隔离现象。马雅军等运用RAPD-PCR技术检测中华按蚊种群遗传分化同样显示分化主要来自于种群内部,但在遗传距离分析上显示,中华按蚊种群遗传距离与地理位置没有对应关系[22]。本研究提示地理隔离对中华按蚊种群间遗传分化从COI序列数据分析上看,似乎有着一定的影响。进一步的中性检验和错配分析结果显示:除云南种群外,其余种群D均为负值,中华按蚊种群在遗传进化中经历过扩张状态。在系统树结果显示:云南元阳种群与其他种群相对独立,在系统树中为孤立一支。一个物种种群的遗传结构受到诸多因素的影响,如分布的地理特征、气候、杀虫剂的使用等。在AMOVA分析中显示出中华按蚊种群内变异明显大于种群间变异是由于云南检测样品数量远远小于其他地方数量之和,从而表现出遗传分化主要来自于种群内部,当然若要证实上述中华按蚊群体的遗传分化现象,需要增加云、贵、广的样本检测。众所周知,我国云南省是一个著名的生物多样性的中心[23],云南省地处云贵高原,地理地貌复杂,气候变化从热带到亚热带,特殊地理气候条件,使得云南地区中华按蚊种群与其他种群间产生了地理隔离现象。Zhong等[24]研究发现,云南地区中华按蚊种群抗药性机理上与其他种群存在巨大的差别,这在一定程度上也与本研究发现的云南种群遗传结构与其他地方种群存在差异的结果相互印证,并显示出种群遗传结构的差异可能是药物抗性等蚊媒治理中倍受关注问题的产生机制不同的遗传基础。
| [1] | 边长玲, 龚正达. 我国蚊类及其与蚊媒病关系的研究概况[J]. 中国病原生物学杂志,2009, 4 (7) : 545-51. |
| [2] | Zhu Guoding, Xia Hui, Zhou Huayun, et al. Susceptibility of Anopheles sinensis to Plasmodium vivax in malarial outbreak areas of central China[J]. Parasite Vector,2013, 6 : 176. DOI: 10.1186/1756-3305-6-176. |
| [3] | 孙定炜, 王善青, 卓开仁, 等. 海南省中华按蚊对3种杀虫剂的抗药性研究[J]. 中国寄生虫学与寄生虫病杂志,2014, 32 (2) : 127-9. |
| [4] | 张玉勤, 刘吉起, 郭祥树, 等. 河南省淮滨县中华按蚊的抗药性研究[J]. 中国寄生虫学与寄生虫病杂志,2012, 30 (6) : 493-5. |
| [5] | 刘颖, 陈建设, 周瑞敏, 等. 中华按蚊对杀虫剂敏感性调查[J]. 中国寄生虫学与寄生虫病杂志,2012, 30 (4) : 9-11. |
| [6] | 杨曼尼, 马雅军. 基于mtDNA-COI基因序列的雷氏按蚊分子群体遗传结构研究[J]. 昆虫学报,2009, 52 (9) : 1000-7. |
| [7] | 武松, 黄芳, 周水森. 我国多斑按蚊复合体mtDNA-COI基因序列特征与系统发育学研究[J]. 中国病原生物学杂志,2013, 8 (2) : 158-60. |
| [8] | 王冬, 马雅军, 周红宁. 应用mtDNA-COI基因序列研究我国大劣按蚊A, D群体的遗传差异[J]. 中国寄生虫学与寄生虫病杂志,2007, 25 (5) : 368-71. |
| [9] | 刘茜, 武松, 汤林华, 等. 伪威氏按蚊mtDNA-Cytb基因群体遗传学研究[J]. 中国人兽共患病学报,2011, 27 (12) : 1071-4. |
| [10] | Ma Y, Yang M, Fan Y, et al. Population structure of the malaria vector Anopheles sinensis (Diptera: Culicidae) in China: two gene pools inferred by microsatellites[J]. PloS One,2011, 6 (7) : e22219. DOI: 10.1371/journal.pone.0022219. |
| [11] | 兰策介, 沈元, 鲁亮, 等. 5种库蚊的COI基因分子进化分析[J]. 中国媒介生物学及控制杂志,2013, 24 (1) : 28-30. |
| [12] | 陆宝麟, 陈汉彬, 瞿逢伊, 等. 中国动物志.昆虫纲.第八卷.双翅目.蚊科(上)[M]. 北京: 科学出版社, 1997 : 1 -884. |
| [13] | Gao Q, Beebe NW, Cooper RD. Molecular identification of the malaria vectors Anopheles anthropophagus and Anopheles sinensis (Diptera: Culicidae) in central China using polymerase chain reaction and appraisal of their position within the Hyrcanus group[J]. J Med Entomol,2004, 41 (1) : 5-11. DOI: 10.1603/0022-2585-41.1.5. |
| [14] | Joshi D, Park MH, Saeung A, et al. Multiplex assay to identify Korean vectors of malaria[J]. Mol Ecol Resour,2010, 10 (4) : 748-50. DOI: 10.1111/men.2010.10.issue-4. |
| [15] | Folmer O, Black M, Hoeh W, et al. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates[J]. Mol Mar Biol Biotechnol,1994, 3 (5) : 294-9. |
| [16] | Rozas J, Sanchez-DelBarrio JC, Messequer X, et al. DnaSP, DNA polymorphism analyses by the coalescent and other methods[J]. Bioinformatics,2003, 19 (18) : 2496-7. DOI: 10.1093/bioinformatics/btg359. |
| [17] | Kimura M. A simple model for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. J Mol Evol,1980, 16 (2) : 111-20. DOI: 10.1007/BF01731581. |
| [18] | Excoffier L, Smouse PE, Quattro JM. Analysis of molecular variance inferred from mrteic distances among DNA haplotypes: Application to human mitochondrial DNA restriction data[J]. Genetics,1992, 131 (2) : 479-91. |
| [19] | Jensen JL, Bohonak AJ, Kelley ST. Isolation by distance, web service[J]. BMC Genet,2005, 6 : 13. |
| [20] | Yoon HS, Hackett JD, Ciniglia C, et al. A molecular timeline for the origin of photosynthetic eukaryotes[J]. Mol Biol Evol,2004, 21 (5) : 809-18. DOI: 10.1093/molbev/msh075. |
| [21] | 袁佳, 王振营, 何康来, 等. 玉米螟赤眼蜂不同地理种群mtDNA基因序列分析及遗传分化研究[J]. 中国生物防治学报,2011, 27 (1) : 16-21. |
| [22] | 马雅军, 郑哲民. 我国中华按蚊群体分子遗传多态研究[J]. 昆虫学报,2001, 44 (1) : 33-9. |
| [23] | Zhu H, Xu ZF, Wang H, et al. Tropical rain forest fragmentation and its ecological and species diversity changes in southern Yunnan[J]. Biodiversity Conservation,2004, 13 (7) : 1355-72. DOI: 10.1023/B:BIOC.0000019397.98407.c3. |
| [24] | Zhong D, Chang X, Zhou G, et al. Relationship between knockdown resistance, metabolic detoxification and organismal resistance to pyrethroids in Anopheles sinensis[J]. PloS One,2013, 8 (2) : e55475. DOI: 10.1371/journal.pone.0055475. |
 2015, Vol. 35
2015, Vol. 35
